В практическом руководстве представлена информация по вопросам локального мониторинга антибиотикорезистентности, в том числе основных этапов его внедрения. Описаны функциональные возможности и область применения онлайн‑платформы AMRcloud для анализа, визуализации и обмена данных антибиотикорезистентности с целью совершенствования взаимодействия участников мониторинга. Практическое руководство предназначено для микробиологов, клинических фармакологов, эпидемиологов, организаторов здравоохранения и специалистов, заинтересованных в вопросах мониторинга антибиотикорезистентности.
Это многостраничный вид для печати этого раздела. Нажмите здесь для печати.
Практическое руководство по мониторингу антибиотикорезистентности с использованием платформы AMRcloud
- Предисловие
- Список сокращений
- 1. Введение
- 2. Организация локального мониторинга
- 2.1 Для чего нужен локальный мониторинг
- 2.2 Основные этапы внедрения системы мониторинга
- 2.3 Взаимодействие с лабораторией
- 3. Реализация системы мониторинга антибиотикорезистентности
- 3.1 Базовые понятия
- 3.2 Показания и особенности взятия биоматериала для микробиологического исследования
- 3.3 Оформление направления на микробиологическое исследование
- 3.4 Основные процедуры в микробиологической лаборатории
- 3.5 Методы определения чувствительности микроорганизмов к антимикробным препаратам и интерпретация результатов
- 3.6 Методы выявления механизмов резистентности, имеющих клиническое и эпидемиологическое значение
- 4. Подготовка данных для эпидемиологического анализа антибиотикорезистентности
- 4.1 Сценарии потоков данных
- 4.2 Шаблон для сбора и ввода данных
- 4.3 Сопроводительная эпидемиологическая, клиническая и демографическая информация
- 4.4 Правила оформления и хранения данных
- 5. Использование платформы AMRcloud в мониторинге антибиотикорезистентности
- 5.1 Создание проектов и наборов данных
- 5.2 Алгоритмы базового анализа данных
- 5.3 Углубленный анализ данных
- 5.4 Актуализация информации
- 5.5 Взаимодействие участников
- 5.6 Использование AMRcloud при формировании локальных протоколов антимикробной терапии
- 6. Приложения
- 6.1 Шаблон направления на микробиологическое исследование
- 6.2 Работа с шаблоном, справочниками и словарями
- 6.3 Работа с WHONET
- 6.4 Работа с BACLINK
- 6.5 Стандартные эпидемиологические определения случая инфекции
- Литература
Предисловие
«Автор пишет только половину книги: другую половину пишет читатель»
На протяжении последних десятилетий наблюдается неуклонный рост и распространение антибиотикорезистентности (АР) среди микроорганизмов различных видов [1] . По некоторым прогнозам, при сохраняющихся тенденциях к 2050 г. в результате АР возбудителей инфекций смертность достигнет показателей до 10 млн в год, а потери мирового ВВП составят около 100 трлн долларов США. Для предотвращения дальнейшего увеличения количества устойчивых микроорганизмов был разработан целый ряд инициатив различного уровня [2] . Так, в 2015 г. Всемирной ассамблеей здравоохранения был принят Глобальный план действий по борьбе с устойчивостью к антимикробным препаратам (АМП) [3] , а в РФ, 25.09.2017 г. издано Распоряжение Правительства РФ № 2045-р «О Стратегии предупреждения распространения антимикробной резистентности в РФ на период до 2030 г.» [4] .
Наряду с разработкой новых АМП, ключевым подходом для решения проблемы АР является рациональное использование существующих препаратов [5] . Соблюдение общих принципов использования АМП (в том числе выбор оптимального препарата с учетом его активности, дозы, кратности и пути введения) лежит в основе рациональной антимикробной терапии. Получение актуальной информации по активности АМП невозможно без постоянного наблюдения за динамикой чувствительности к ним возбудителей и своевременного анализа получаемых данных [6] . Для выполнения этой задачи разрабатываются системы мониторинга АР различного уровня.
Инициатором научно-исследовательских проектов по мониторингу АР на территории РФ являлся основатель НИИ антимикробной химиотерапии (НИИ АХ) и Межрегиональной ассоциации по клинической микробиологии и антимикробной химиотерапии (МАКМАХ), д.м.н., профессор, член-корреспондент РАМН Леонид Соломонович Страчунский (1952-2005) [7] [8] [9] . В настоящее время на базе НИИ АХ и МАКМАХ в рамках многоцентровых проспективных исследований продолжается всестороннее изучение возбудителей инфекций, включая механизмы приобретения резистентности и особенности их распространения [2] . Эти данные лежат в основе системы мониторинга национального уровня, открытой онлайн-платформы анализа данных по резистентности к антимикробным препаратам – AMRmap (https://amrmap.ru/) [10] .
Несмотря на несомненные преимущества системы мониторинга, включающей широкий перечень субъектов РФ, такой уровень представления данных может быть использован преимущественно в качестве справочной информации, поскольку для каждой медицинской организации характерны свои индивидуальные показатели в области АР (распределение возбудителей, чувствительность к АМП, клинико-эпидемиологические данные).
Поэтому наиболее объективную картину можно получить, проводя локальный мониторинг АР [11] . При организации локального мониторинга могут возникать существенные трудности, особенно на одном из самых объемных этапов – анализе и оценке итоговых (объединенных) данных. Для того чтобы упростить данный процесс была создана онлайн-платформа по анализу, визуализации и представлению данных по антибиотикорезистентности – AMRcloud (https://amrcloud.net/) [12] .
Настоящее издание содержит важные для микробиологов, лечащих врачей и организаторов здравоохранения сведения, касающиеся вопросов локального мониторинга АР, в том числе основных этапов его внедрения. Описаны функциональные возможности и область применения онлайн-платформы AMRcloud для анализа, визуализации и обмена данных АР с целью совершенствования взаимодействия участников мониторинга.
Надеемся, что научно-методическое пособие будет полезно при решении вопросов по локальному мониторингу АР.
Авторы приглашают читателей к сотрудничеству. Комментарии, критические замечания, вопросы и пожелания можно направлять на электронную почту: support@antibiotic.ru
Список сокращений
| Сокращение | Полное наименование |
|---|---|
| АР | антибиотикорезистентность |
| АБТ | антибактериальная терапия |
| АМП | антимикробный препарат |
| АМР | антимикробная резистентность |
| АМТ | антимикробная терапия |
| ВОЗ | Всемирная организация здравоохранения |
| ГОНФ | грамотрицательные неферментирующие бактерии |
| ДДМ | диско-диффузионный метод |
| ДИ | доверительный интервал |
| ИСМП | инфекции, связанные с оказанием медицинской помощи |
| ЛИС | лабораторная информационная система |
| МИ | микробилогическое исследование |
| МИС | медицинская информационная система |
| МО | микроорганизм |
| ЛС | лекарственные средства |
| ЛПС | липополисахарид |
| ЛПУ | лечебно-профилактическое учреждение |
| МАНК | методы амплификации нуклеиновых кислот |
| МПК | минимальная подавляющая концентрация |
| НИ | нозокомиальная инфекция |
| ПСБ | пенициллин-связывающий белок |
| ПЦР | полимеразная цепная реакция |
| CLSI | Институт по клиническим и лабораторным стандартам США |
| CPE | карбапенемазопродуцирующие энтеробактерии |
| DDM | диско-диффузионный метод (disk diffusion method) |
| ECOFF | эпидемиологические точки отсечения (epidemiological cut-off values) |
| ESBL | β-Лактамазы расширенного спектра |
| EUCAST | Европейский комитет по определению чувствительности к антимикробным препаратам |
| MIC | метод минимальных подавляющих концентраций (minimum inhibitory concentration) |
| MRSA | штаммы S. aureus, проявляющие фенотип метициллинорезистентности |
| PSB | методика защищенной щеточки (protected specimen brush) |
| VRE | ванкомицинорезистентные энтерококки |
| WGS | методы геномного секвенирования |
1. Введение
1.1 Важность мониторинга антибиотикорезистентности
Проблема роста антибиотикорезистентности (АР) в последние десятилетия занимает одну из ключевых позиций в системах общественного здравоохранения и требует особого внимания со стороны медицинского сообщества [5] [13] [14] . Базовой составляющей для отслеживания динамики чувствительности микроорганизмов к антимикробным препаратам является мониторинг АР [15] [16] .
Мониторинг антибиотикорезистентности
это систематический, непрерывный процесс сбора, анализа и представления данных по резистентности к антимикробным препаратам. Полученная в рамках мониторинга информация имеет практические точки приложения, включая организацию лечебных и профилактических мероприятий.Принципами мониторинга являются:
- непрерывность,
- стандартизация,
- комплексность,
- курируемость.
Непрерывность – мониторинг проводится на постоянной основе.
Стандартизация – соблюдение единого дизайна и принципов проведения исследования: сбора, анализа и интерпретации результатов [17] . Кроме того, сохранение результатов в электронном, пригодном для последующей пакетной статистической обработки виде (в электронных таблицах и/или лабораторных информационных системах) должно осуществляться в соответствии с разработанными правилами (использование словаря различных наименований, определенное оформление заголовочной строки таблицы и т. д.). При составлении справочников и словарей необходимо придерживаться принципа «разумной достаточности» - исключение излишних подробностей в записях, что позволяет рассмотреть динамику устойчивости в общем виде (при этом добавление новых записей не должно противоречить уже существующим).
Комплексность – данные по каждому возбудителю должны включать не только результаты определения чувствительности, но и дополнительную информацию о пациенте, заболевании и лечебном учреждении. Сбор данных в таком объеме позволяет выявить (или обнаружить) неявные причинно-следственные связи между появлением устойчивости микроорганизмов к АМП и наличием определенных факторов. Курируемость – регулярный контроль качества процесса получения данных (включая процессы сбора, микробиологических исследований, хранения и обработки данных).
Основными этапами работы с данными мониторинга являются:
- непрерывный сбор данных,
- анализ данных,
- интерпретация данных,
- практическое использование результатов.
Ключевые задачи мониторинга АР [18] :
- выявление АР;
- анализ АР;
- прогнозирование изменений в показателях АР;
- установление новых механизмов АР;
- сравнение активности новых антибиотиков до и после их внедрения в клиническую практику;
- обучение медицинского сообщества, пациентов, заинтересованных лиц;
- разработка практических мер для снижения уровня АР;
- оценка эффективности профилактических вмешательств по контролю АР.
1.2 Системы мониторинга
Система мониторинга
это комплекс мероприятий, который позволяет организовывать и анализировать данные с предоставлением результатов по запросу специалиста.Необходимость учета большого объема данных способствует развитию систем мониторинга [19] . При этом существуют особенности, которые отличают системы между собой и обуславливают их классификацию.
Типы систем мониторинга:
- По открытости:
- открытые – результаты доступны для любого пользователя
- закрытые – для доступа к данным необходимы логин и пароль
- По объему данных:
- без ограничений – включен максимально возможный объем данных
- с ограничениями – включен объем данных, отобранный согласно критериям (ограничения перечня АМП, видов микроорганизмов и т. д.)
- По уровню организации:
- локальные – данные по АР получены в рамках определенного географического объекта (медицинской организации)
- национальные – данные по АР получены из различных географических объектов, значительно отличающихся по географическому расположению, в рамках одной страны
- международные – данные по АР получены в различных странах
Примеры международных систем мониторинга:
- Европейская система по наблюдению за антимикробной резистентностью (The European Antimicrobial Resistance Surveillance Network; EARS-Net); https://www.ecdc.europa.eu/en/about-us/partnerships-and-networks/disease-and-laboratory-networks/ears-net
- Cистема эпиднадзора за устойчивостью к противомикробным препаратам в Центральной Азии и Восточной Европе (The Central Asian and Eastern European Surveillance of Antimicrobial Resistance; CAESAR); https://www.euro.who.int/en/health-topics/disease-prevention/antimicrobial-resistance/publications/2019/central-asian-and-european-surveillance-of-antimicrobial-resistance.-annual-report-2019
- ResistanceMap, разработанная Центром динамики заболеваний, экономики и политики (The Center for Disease Dynamics, Economics & Policy, CDDEP); https://resistancemap.cddep.org/AntibioticResistance.php
- Глобальная система надзора за антибиотикорезистентностью (The Global Antimicrobial Resistance Surveillance System; GLASS); https://www.who.int/glass/en/
Примеры национальных систем мониторинга:
- AMRmap (Российская Федерация); https://amrmap.ru/
- Национальная сеть безопасности здравоохранения (National Healthcare Safety Network; NHSN, США); https://arpsp.cdc.gov/
- BSAC Resistance Surveillance Programme (Великобритания); http://www.bsacsurv.org/
2. Организация локального мониторинга
2.1 Для чего нужен локальный мониторинг
В ЛПУ локальный мониторинг АР необходим для решения задач, связанных с двумя направлениями:
- оптимизация антимикробной терапии,
- планирование, реализация и оценка мероприятий инфекционного контроля.
Одним из способов оптимизации АМТ инфекций является составление протоколов по антимикробной терапии и формуляров антимикробных средств [20] . Однако меняющиеся тренды АР приводят к устареванию этих протоколов. Вышеупомянутые документы должны периодически актуализироваться на основе данных по активности АМП в отношении ключевых возбудителей инфекций.
Информация по активности АМП может быть получена из референсных источников: международных и национальных систем мониторинга антибиотикорезистентности [21] [22] [23] [24] . В то же время международные и национальные данные не могут в полной мере удовлетворить локальные потребности при составлении протоколов АМТ и формуляров антимикробных средств. Это обусловлено задержкой в публикации данных (более 1 года), локальными отклонениями в эпидемиологии АР и особенностями конкретной медицинской организации [25] .
Организация локального мониторинга АР является базовым принципом для своевременной оценки масштабов резистентности и модификации терапевтических подходов для надлежащего использования антимикробных препаратов [26] .
2.2 Основные этапы внедрения системы мониторинга
1. Формирование рабочей группы
Рекомендуемый состав:
- микробиолог;
- эпидемиолог;
- клинический фармаколог;
- администрация;
- лечащий врач;
- специалист по информационным технологиям.
Состав рабочей группы и количество участников может изменяться в зависимости от конкретного стационара, однако необходимо обеспечить присутствие компетентных представителей всех отделов и направлений деятельности, которые будут задействованы в ходе внедрения системы локального мониторинга.
В составе рабочей группы выделяются:
- лицо, принимающее решение;
- лицо, ответственное за исполнение;
- исполнители.
В некоторых случаях лицо, принимающее решение, и лицо, ответственное за исполнение, могут совпадать, но лучше разделить эти роли (например, лицо, принимающее решение – главный врач; лицо, ответственное за исполнение – микробиолог, эпидемиолог или клинический фармаколог). Нужно учитывать, что лицо, ответственное за исполнение, должно постоянно взаимодействовать с другими членами рабочей группы и контролировать процесс внедрения. Для координации процесса необходимо, чтобы исполнители не взаимодействовали с лицом, принимающим решения, напрямую, а только через лицо, ответственное за исполнение.
2. Проведение аудита
До каждого члена группы необходимо довести информацию о его роли в процессе внедрения системы мониторинга, целях и поставленных задачах. Каждый из членов рабочей группы должен оценить, насколько текущее состояние его сферы ответственности соответствует итоговым задачам процесса мониторинга.
Для такой оценки можно использовать следующий чеклист:
- Как организован поток данных в стационаре, на каких этапах присутствуют «узкие места»:
- где максимальный процент ручного заполнения;
- где больше всего используется бумажный носитель;
- где тратится больше всего времени на внесение данных;
- как организовано хранение и доступ к данным.
- Какие локальные справочники необходимо создать или модифицировать:
- антибиотики;
- локусы инфекции;
- диагнозы;
- клинические материалы;
- типы пациентов;
- отделения, с которыми осуществляется взаимодействие.
- Соответствуют ли бумажные направления/электронные формы ввода рекомендациям по оптимальному хранению/передачи информации. Подробная информация представлена в разделах 4.2. Шаблон для сбора и ввода данных и 4.4. Правила оформления и хранения данных.
- Существует ли график отчетности по текущим показателям АР лицу, принимающему решение и соответствует ли он требуемой периодичности.
- Соответствуют ли отчеты требуемой форме и достаточны ли они для принятия решений.
- Доступны ли данные о локальной эпидемиологии АР профильным специалистам.
- Соответствует ли уровень компетенции персонала предъявляемым требованиям, потребуется ли дополнительный инструктаж/обучение.
- Обеспечивает ли текущий технический уровень программно-аппаратных средств реализацию системы мониторинга, какое оборудование необходимо приобрести дополнительно.
3. Разработка программы внедрения
На основании проведенного аудита, обсуждения и согласованного мнения членами рабочей группы должна быть разработана программа внедрения системы мониторинга, включающая:
- бланк направления на микробиологическое исследование (МИ). Подробнее в разделе 6.1. Шаблон направления на микробиологическое исследование;
- перечень справочников, которые необходимо сформировать (и которые должны быть использованы в бланках направления, лабораторных информационных системах/медицинских информационных системах). Подробнее в разделе 6.2. Работа с шаблоном, справочниками и словарями;
- шаблоны форм данных, которые необходимо реализовать для этапа ввода данных. Подробная информация представлена в разделе 4.2. Шаблон для сбора и ввода данных;
- формы отчетов и периодичность их формирования;
- порядок составления протоколов и периодичность их пересмотра;
- перечень оборудования, программно-аппаратных средств, которые необходимо приобрести;
- перечень задач по внедрению системы мониторинга, которые необходимо выполнить в рамках каждого направления с указанием ответственного исполнителя и даты, к которой задачи должны быть выполнены;
- порядок инструктажа и обучения персонала новым целям/задачам/методикам системы мониторинга;
- график представления результатов внедрения лицу, ответственному за исполнение;
- сроки внедрения и количественные показатели, соответствующие итогу внедрения системы мониторинга.
4. Функционирование рабочей группы после внедрения
После перехода системы мониторинга в штатный режим работы необходимо определить:
- Лицо, ответственное за формирование, хранение и публикацию собранных данных (в доступных для участников группы ресурсах);
- Направления работы с собранными данными для членов рабочей группы:
- микробиолог – оптимизирует процессы в лаборатории (актуализация перечня АМП для определения чувствительности, осуществление контроля качества и т.д.);
- эпидемиолог – использует полученные данные как часть эпидемиологического мониторинга;
- клинический фармаколог – на основании собранных данных пересматривает протоколы антимикробной терапии и составляет формуляры АМП с установленной периодичностью;
- лечащий врач – взаимодействуют с клиническими фармакологами, эпидемиологами, микробиологами с целью получения и использования актуальных данных в повседневной практике;
- администрация – оптимизирует бюджетные расходы (закупка АМП, расходных материалов для диагностики АР).
- График и порядок проведения мероприятий по информированию персонала о текущем уровне АР;
- Периодичность пересмотра существующей системы мониторинга с целью её адаптации к изменяющимся условиям, включая пересмотр всех форм документов.

2.3 Взаимодействие с лабораторией
Ключевой структурой в локальном мониторинге АР является микробиологическая лаборатория и/или специалисты, осуществляющие МИ в отделении клинической лабораторной диагностики. Для налаживания системы мониторинга АР необходимо выстроить четкие механизмы взаимодействия с лабораторией, а при отсутствии в учреждении собственной микробиологической лаборатории – с централизованной (внешней) лабораторией.
Особое внимание следует уделить следующим аспектам:
- модификации направления на микробиологическое исследование в соответствии с требованием рабочей группы по мониторингу АР;
- внесению дополнительных параметров в медицинскую информационную систему (МИС) для автоматического формирования направления на МИ и интеграции с локальным журналом МИ;
- уточнению и оценке методов определения чувствительности к антибактериальным препаратам (как фенотипических, так и генотипических методов), использующихся в лаборатории;
- оценке и приведению в соответствие наборов антибактериальных препаратов, использующихся для определения чувствительности основных возбудителей инфекций;
- уточнению критериев интерпретации для определения клинической категории чувствительности, использующихся как в лаборатории, так и в централизованной лаборатории;
- для централизованной (внешней) лаборатории – наличию в предоставляемых результатах определения чувствительности количественных данных (минимальные подавляющие концентрации (МПК), диаметры зон подавления роста), помимо клинических категорий чувствительности;
- назначению сотрудника, ответственного за сбор и публикацию данных, которые будут получены из лаборатории, в доступных для участников рабочей группы ресурсах;
- регистрации направления на МИ в локальном журнале/форме/ шаблоне данных медицинской организации (предпочтительно в цифровом виде, с внесением всей сопутствующей информации в соответствии с локально утвержденными справочниками и словарями) перед отправлением в централизованную (внешнюю) лабораторию;
- внесению результатов определения чувствительности (в том числе количественных данных) в локальный журнал/форму/ шаблон данных медицинской организации после получения информации из централизованной (внешней) лаборатории;
- утверждению формы предоставления результатов из централизованной (внешней) лаборатории:
- по индивидуальным результатам пациентов (в виде стандартной формы, с учетом требований рабочей группы);
- в виде дополнительной выгрузки данных в заданном формате (таблицы) и периодичностью, утвержденной рабочей группой.
3. Реализация системы мониторинга антибиотикорезистентности
3.1 Базовые понятия
Микробная колонизация
это заселение участков кожи, слизистых оболочек и различных изделий медицинского назначения микроорганизмами, не сопровождающееся признаками и симптомами заболевания.Как правило, происходит колонизация тканей организма, которые находятся в контакте с внешней средой (слизистые оболочки мочеполового и пищеварительного тракта, дыхательных путей, кожа и конъюнктива). Микроорганизмы, колонизирующие эти области, имеют различные механизмы адгезии, способны преодолевать и выдерживать постоянное сопротивление защитных механизмов макроорганизма, таким образом, находясь с последним в состоянии динамического равновесия без причинения вреда в данный момент времени.
Инфекция
это инвазия и размножение чужеродных агентов в организме хозяина, приводящие к патологическому состоянию или заболеванию.В результате инфекции, т.е. проникновения чужеродного агента в организм хозяина, размножения в нем, высвобождения продуктов метаболизма (ферменты, эндо- и экзотоксины) нарушаются нормальные физиологические процессы, постоянство внутренней среды (гомеостаз) в организме хозяина. Это приводит к мобилизации неспецифических и специфических механизмов защиты, направленных на уменьшение или ликвидацию чужеродного агента с целью компенсации повреждения.
Почему для задач мониторинга АР важно различать колонизацию и инфекцию?
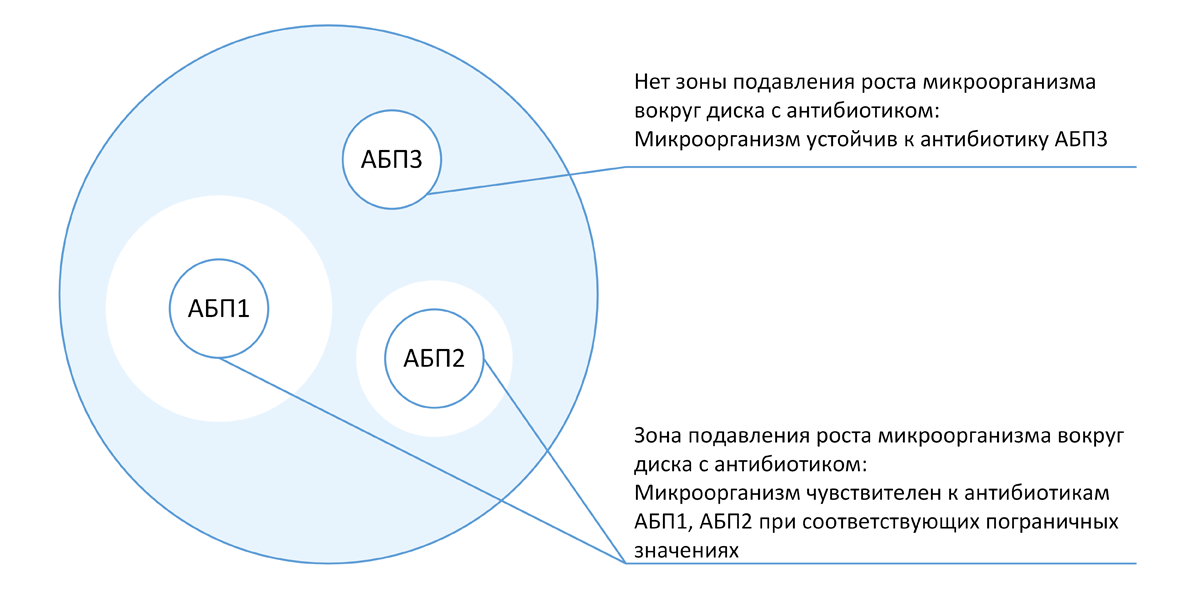
С точки зрения мониторинга АР принципиально важным является разделение всех выделенных от пациента микроорганизмов на:
- клинически значимые изоляты, т.е. изоляты являющиеся причиной инфекционного процесса конкретной локализации (например, S. pneumoniae при инфекциях нижних дыхательных путей, E. coli при глубоких инфекциях в области хирургического вмешательства);
- колонизирующие, т.е. микроорганизмы не являющиеся причиной инфекции конкретной локализации (например, зеленящие стрептококки в ротовой полости, S. epidermidis на поверхности кожи; E. coli на поверхности мочевого катетера при отсутствии симптомов инфекции мочевыводящих путей; микроорганизмы, выделенные из нестерильных локусов при исследовании на носительство). Следует отметить, что при наличии определенных условий (входные ворота инфекции, ослабленный иммунный статус и т.п.), колонизирующий изолят способен вызвать инфекционный процесс, т.е. стать клинически значимым.
Клинически значимые изоляты в действительности отражают этиологическую структуру с соответствующим профилем резистентности для конкретной нозологии (т.е. при уже совершившемся случае инфекции). В свою очередь, колонизирующие изоляты отражают состояние микробиоты (с характерным профилем резистентности) и представляют потенциальных возбудителей инфекций (однако сам случай инфекции отсутствует). Данная классификация является условной. С точки зрения оценки трендов АР и последующего составления локальных протоколов по антимикробной терапии наибольшую ценность представляют клинически значимые изоляты.
Следует обратить внимание, что для отдельных категорий пациентов (например, новорожденные дети, онкогематологические и иммунокомпрометированные пациенты) колонизирующие изоляты имеют особое значение, т.к. с большей вероятностью (в силу особенностей статуса макроорганизма) могут стать этиологическим агентом при наступлении случая инфекции.
С понятием «клинически значимые» изоляты неразрывно связано определение случая инфекции.
Что такое эпидемиологическое определение случая инфекции?
Эпидемиологическое определение случая инфекции
это набор стандартных критериев для выявления у пациента определенного заболевания, синдрома или другого состояния с целью эпидемиологического надзора.Использование согласованного стандартного определения случая инфекции гарантирует, что все случаи эквивалентны, независимо от времени и места их идентификации, что обеспечивает сопоставимость данных. Стандартные эпидемиологические определения случаев инфекции представлены в приложении 6.5.
Что такое нозокомиальные инфекции и в чем заключается важность определения случая нозокомиальной инфекции с точки зрения мониторинга АР?
Согласно формальному определению ВОЗ, нозокомиальная инфекция (НИ)
это любое клинически распознаваемое инфекционное заболевание, которое развивается у пациента не менее чем через 48 часов после госпитализации (и не находившееся в инкубационном периоде на момент госпитализации), в течение 3 дней после выписки или 30 дней после операции (до 1 года – при наличии имплантатов), а также любое инфекционное заболевание сотрудника стационара, развившееся вследствие его работы в данном учреждении, вне зависимости от времени появления симптомов.Наиболее распространенными являются следующие типы НИ: инфекции нижних дыхательных путей, инфекции мочевыводящих путей, инфекции в области хирургического вмешательства, инфекции кровотока.
С точки зрения мониторинга АР принципиально важным является разделение инфекций на два типа: нозокомиальные и внебольничные (т.е. не удовлетворяющие критериям нозокомиальных). Данное разделение необходимо, так как для внебольничных и нозокомиальных инфекций одних и тех же локализаций существуют значимые различия в этиологической структуре и профиле резистентности возбудителей. Поэтому при статистической обработке данных необходимо производить расчеты показателей (частоты встречаемости возбудителя, относительное количество резистентных изолятов и т.п.) отдельно для каждого типа инфекций (Рисунок 1).
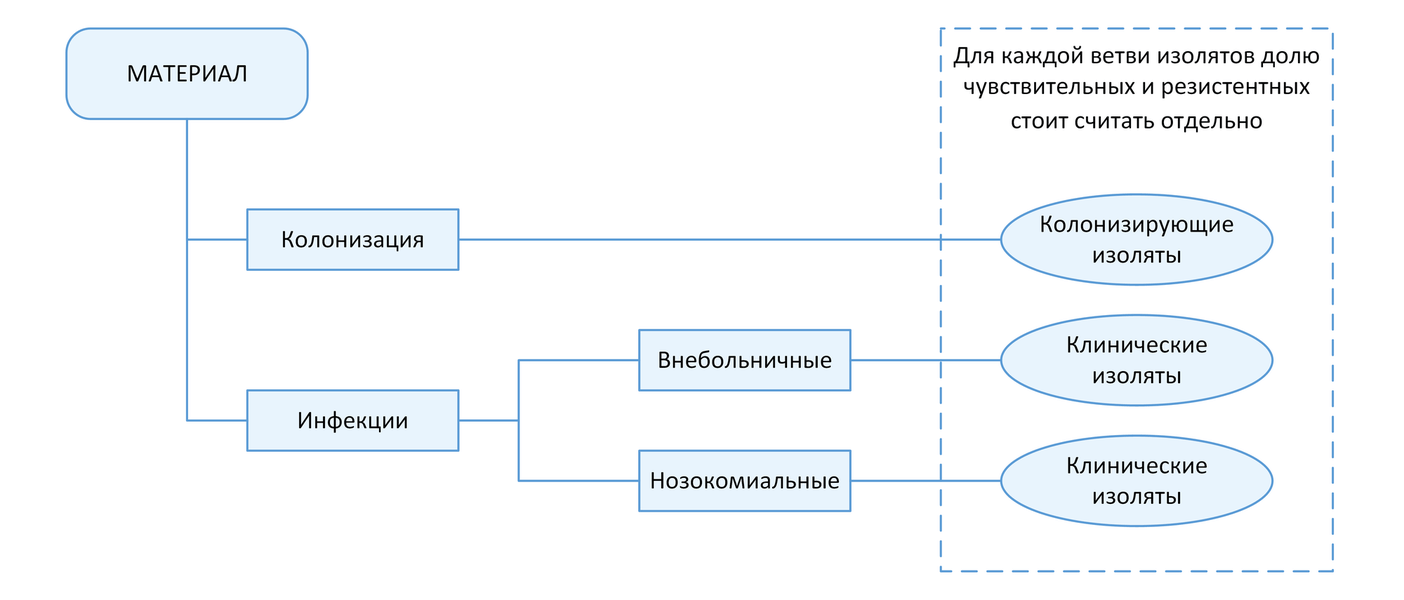
Какие микроорганизмы являются приоритетными для мониторинга АР?
Согласно рекомендациям ВОЗ (Cистема эпиднадзора за устойчивостью к противомикробным препаратам в Центральной Азии и Восточной Европе, 2019 г.) первоочередной интерес для мониторинга АР представляют девять видов (групп) микроорганизмов [27] :
- Escherichia coli (E. coli);
- Klebsiella pneumoniae (K. pneumoniae);
- Salmonella spp.;
- Pseudomonas aeruginosa (P. aeruginosa);
- Acinetobacter spp.;
- Staphylococcus aureus (S. aureus);
- Streptococcus pneumoniae (S. pneumoniae);
- Enterococcus faecalis (E. faecalis);
- Enterococcus faecium (E. faecium).
В качестве ориентира для формирования списка приоритетных для мониторинга АР возбудителей в РФ можно также использовать данные AMRmap (https://amrmap.ru система мониторинга АР национального уровня). Частота встречаемости различных возбудителей внебольничных и нозокомиальных инфекций в РФ (согласно данным AMRmap) представлена в таблице 3.1.1. Возбудители частота встречаемости которых не превышает 2% в таблицу не включены.
Таблица 3.1.1. Частота встречаемости различных видов микроорганизмов при внебольничных и нозокомиальных инфекция в РФ согласно данным AMRmap
| Вид | Абсолютная частота (N) | Относительная частота (%) |
|---|---|---|
| Нозокомиальные | ||
| Pseudomonas aeruginosa | 5049 | 19.31 |
| Klebsiella pneumoniae | 4975 | 19.02 |
| Acinetobacter baumannii | 3583 | 13.7 |
| Staphylococcus aureus | 3378 | 12.92 |
| Escherichia coli | 3187 | 12.19 |
| Enterococcus faecalis | 1005 | 3.84 |
| Enterobacter cloacae | 744 | 2.85 |
| Proteus mirabilis | 696 | 2.66 |
| Stenotrophomonas maltophilia | 616 | 2.36 |
| Enterococcus faecium | 603 | 2.31 |
| Serratia marcescens | 528 | 2.02 |
| Внебольничные | ||
| Escherichia coli | 3633 | 22.43 |
| Streptococcus pneumoniae | 2916 | 18.01 |
| Staphylococcus aureus | 2293 | 14.16 |
| Streptococcus pyogenes | 2262 | 13.97 |
| Haemophilus influenzae | 1105 | 6.82 |
| Klebsiella pneumoniae | 925 | 5.71 |
| Neisseria gonorrhoeae | 679 | 4.19 |
| Pseudomonas aeruginosa | 472 | 2.91 |
| Enterococcus faecalis | 335 | 2.07 |
Следует отметить, что при налаженном процессе локального мониторинга АР не представляет сложности расширить предложенный список микроорганизмов. Более того, включение всех выделяемых видов микроорганизмов в процесс мониторинга АР (учитывая тип инфекции и колонизацию) является предпочтительным, т.к. позволяет получить полное представление о локальной эпидемиологии АР.
Какое количество изолятов является репрезентативным для мониторинга АР?
В рамках мониторинга АР осуществляется расчет различных показателей, как правило выраженных в абсолютных и относительных частотах. Например, расчет и оценка доли (%) конкретного микроорганизма в этиологической структуре возбудителей инфекций или оценка доли резистентных изолятов. Ввиду различного объема выборки (количества изолятов) степень уверенности в достоверности полученных показателей может быть различной. Для объективной оценки степени уверенности (доверия) в полученных данных существует специальный показатель – доверительный интервал (ДИ). Наиболее часто используемым ДИ в биомедицинских исследованиях является 95%-ый ДИ [28] . Он представлен в виде интервальных значений, например, 50% резистентных изолятов (95%-ый ДИ: 30–70%). Доверительный интервал (95%-ый ДИ), означает, что при повторном проведении 100 исследований с таким же дизайном истинное значение оцениваемого показателя (в данном примере доли резистентных изолятов) в 95 исследованиях из 100 будет находиться в пределах указанной ширины интервала (в данном примере ширина – от 30 до 70%). Таким образом, в указанном примере несмотря на оценку доли резистентных изолятов в 50%, истинная доля может находиться в пределах 30–70%.
ДИ напрямую связан с объемом выборки (количеством изолятов): чем больше объем выборки (количество изолятов), тем меньше ширина ДИ (т.е. большая точность оценки), и наоборот. ДИ рассчитывается с помощью специальных формул. Однако для приблизительной оценки необходимого объема выборки (точности оценки) можно ориентрироваться на значения, представленные в таблице 3.1.2.
Таблица 3.1.2. Ширина 95%-ого ДИ в зависимости от объема выборки
| Объем выборки (количество изолятов) | Ширина 95%-ого ДИ (в %) |
|---|---|
| 20 | ± 20,07 |
| 40 | ± 14,08 |
| 80 | ± 10,70 |
| 100 | ± 6,86 |
| 200 | ± 4,88 |
| 300 | ± 3,99 |
| 500 | ± 3,09 |
Исходя из таблицы 3.1.2., при выборке в 20 изолятов истинное значение доли резистентных изолятов будет находиться в пределах ± 20,07%. Например, если 10 из 20 (т.е. 50%) протестированных изолятов резистентны к конкретному антимикробному препарату, то истинное значение доли резистентных изолятов будет находиться в пределах 50 ± 20,07%. Такой же принцип действителен при оценке этиологической структуры. Например, при инфекциях конкретной локализации идентифицировано 80 изолятов из них 40 изолятов (50%) составляет Klebsiella pneumoniae, используя таблицу 3.1.2. можно приблизительно оценить истинное значение доли Klebsiella pneumoniae в общей структуре (50 ± 10,70%).
Следует отметить, что наряду с использованием ширины 95%-ого ДИ для вероятностной оценки точности и необходимого количества изолятов, с целью формирования репрезентативной выборки необходимо также учитывать «смещающие» факторы (например, включение в исследование только изолятов резистентных к конкретному АМП или включение дублей и повторных изолятов, см. раздел 4.3.).
3.2 Показания и особенности взятия биоматериала для микробиологического исследования
Показания для проведения МИ могут быть условно разделены на клинические и эпидемиологические.
Клинические:
- подтверждение диагноза или установление этиологии инфекционного процесса;
- обоснование выбора или смены АБТ;
- оценка эффективности АБТ.
Эпидемиологические:
- определение условно-патогенных возбудителей в воздухе, на объектах окружающей среды в стационаре;
- определение широты распространения, уровня и массивности носительства медперсоналом и пациентами возбудителей инфекций.
При определении показаний и подготовке пациента для МИ необходимо учитывать соответствие назначения клиническим рекомендациям и стандартам оказания медицинской помощи для соответствующей патологии.
Общие правила взятия и транспортировки клинического материала
Определение правил взятия и транспортировки клинического материала для МИ и разработка программ обучения медицинского персонала, участвующего в данном этапе диагностики, является обязанностью лаборатории.
Перед взятием образца, особенно при применении инвазивных методов, необходимо оценить соотношение риска и пользы для пациента, а также значимость данного вида клинического материала для целей объективизации клинического диагноза и оценки проводимых или планируемых лечебных мероприятий. Неадекватные и/или неправильно собранные образцы для исследования не обеспечивают получение полезной клинической информации и могут ввести в заблуждение.
Все медицинские работники, выполняющие взятие материала, должны проходить обучение (инструктаж) по правилам взятия и транспортировки материала для МИ. Периодичность инструктажа устанавливается внутренними правилами медицинской организации.
В случае самостоятельного сбора клинического материала (моча, мокрота) пациент должен получить четкие пошаговые инструкции (устные и/или письменные).
Взятие материала для исследования должно проводиться предпочтительно в острой фазе заболевания, до начала АБТ или непосредственно перед введением (приемом) очередной дозы антибиотика.
Внимание!
Необходимость взятия материала для микробиологического исследования не должна быть причиной отложенного начала антибактериальной терапии.При взятии материала необходимо руководствоваться следующими принципами:
- соблюдать технику асептики и антисептики для предотвращения инфицирования пациента в процессе выполнения инвазивных манипуляций;
- проводить взятие клинического материала непосредственно из очага инфекции;
- собирать клинический материал в количестве (вес, объем) достаточном для проведения исследования;
- обеспечить минимально возможную контаминацию материала нормальной микробиотой; (присутствие в заключении информации о представителях нормальной микробиоты может привести к ошибочной интерпретации результатов исследования материала нестерильных локусов организма, например, при исследовании мокроты, мазков из ротои носоглотки, гениталий и др.);
- использовать специализированные стерильные одноразовые контейнеры, предназначенные для сбора и транспортировки биологического материала, или транспортные системы, содержащие или не содержащие транспортную среду для сохранения жизнеспособности и количественного состава микроорганизмов, с плотно закрывающимися крышками.
Выбор контейнера для сбора клинического материала определяется типом пробы и целью МИ.
Некоторые образцы требуют специальной транспортной среды для сохранения микроорганизмов (например, исследования при подозрении на анаэробную инфекцию).
Образцы материала для МИ следует доставить в лабораторию в максимально короткие сроки (для большинства образцов не позднее 1,5–2 ч после их получения), за исключением случаев использования специальных изделий, допускающих отсроченную доставку материала в лабораторию.
Это обеспечивает:
- сохранение жизнеспособности возбудителей и возможности выделения микроорганизмов, требующих особых условий культивирования;
- предотвращение избыточного роста быстрорастущих микроорганизмов;
- поддержание соотношения исходных концентраций микроорганизмов в образце.
Основными требованиями к транспортировке клинического материала в микробиологическую лабораторию являются:
- наличие четкой маркировки образца для безошибочной идентификации;
- наличие заполненного бланка направления, содержащего необходимую клинико-демографическую и эпидемиологическую информацию;
- предотвращение контакта направления на исследование (и других сопроводительных материалов) с клиническим материалом (например, упаковка документов в отдельный пластиковый пакет);
- соблюдение дополнительных правил, предъявляемых к транспортировке отдельных видов клинического материала для МИ.
При невозможности немедленной доставки в лабораторию образцы хранят при температуре 2–8 °С за исключением следующих случаев:
- использование специальных транспортных систем, содержащих транспортную среду;
- подозрение на наличие температурозависимых микроорганизмов (Neisseria spp.) – временное хранение и доставка образцов осуществляется при температуре 18–20 °С.
Требования к взятию, хранению и транспортировке материала в микробиологическую лабораторию доступны в действующих на данный момент национальном стандарте и методических указаниях:
- ГОСТ Р 53079.4-2008. Технологии лабораторные клинические. Обеспечение качества клинических лабораторных исследований. Часть 4. Правила ведения преаналитического этапа [29] .
- МУ 4.2.2039-05. Техника сбора и транспортирования биоматериалов в микробиологические лаборатории [30] .
Указанные нормативные документы введены в действие более 10 лет назад и носят общий характер, в связи с чем целесообразна разработка соответствующих локальных стандартных операционных процедур, учитывающих профиль выполняемых исследований. При этом полезно ориентироваться на рекомендации, отражающие современное состояние лабораторных технологий, например, для посева крови [31] .
При возникновении вопросов необходимо проконсультироваться со специалистами лаборатории.
3.3 Оформление направления на микробиологическое исследование
Клинические и демографические данные являются необходимыми для оптимизации процедуры исследования в лаборатории (выбор питательных сред для первичного посева и условий инкубации и др.), определения клинической значимости выделенных микроорганизмов и правильной интерпретации результатов исследования в целом. Сопроводительная информация, содержащаяся в направлении (вместе с полученными результатами идентификации и определения чувствительности выделенных возбудителей) будет являться частью массива данных для анализа в процессе мониторинга АР. Поэтому отдельное внимание должно быть уделено модификации или разработке бланка направления на МИ.
Образцы клинического материала должны отправляться в лабораторию в сопровождении бланка направления, включающего следующую информацию:
- фамилия, имя, отчество обследуемого;
- дата рождения;
- лечебное учреждение;
- отделение: наименование и профиль (например, хирургический, терапевтический);
- номер истории болезни или амбулаторной карты;
- диагноз и локализацию инфекционного процесса, явившегося поводом к МИ;
- тип инфекции в зависимости от времени возникновения (внебольничная или нозокомиальная);
- вид биологического материала;
- задачи исследования при необходимости (например, исследование мазка из ротоглотки с целью выявления Corynebacterium diphtheriae);
- дата и время взятия материала;
- получаемые антимикробные препараты;
- при направлении аутопсийного материала – отделение, в котором умер пациент, дату и время смерти, дату и время вскрытия;
- дополнительные клинические данные при необходимости (факторы риска развития инфекции и/или наличия резистентных возбудителей);
- фамилию, имя, отчество и контактный телефон врача, назначившего исследование.
Пример бланка направления на МИ представлен в приложении 6.1. Шаблон направления на микробиологическое исследование.
3.4 Основные процедуры в микробиологической лаборатории
В рамках микробиологического исследования проводят следующие процедуры:
1. Прием и оценка качества и репрезентативности биоматериала для микробиологического исследования в лаборатории.
При поступлении образцов биоматериала в лабораторию необходимо:
- Оценить полноту заполнения направления (сопроводительной информации) для МИ.
- Оценить качество образца (маркировка, целостность контейнера, соблюдение условий взятия и транспортировки (надлежащий контейнер/транспортная среда, сроки доставки и т.д.).
- Оценить соответствие вида клинического материала предполагаемой инфекционной патологии: локализация и возможность выделения вероятных возбудителей.
Внимание!
При выявлении несоответствий необходимо провести бракераж образцов, регистрацию выявленных несоответствий, а также информировать лечащего врача (или другое лицо, ответственное за взятие материла) и обсудить возможные дальнейшие действия. Основные процедуры описаны в ГОСТ ИСО 15189 «Лаборатории медицинские. Частные требования к качеству и компетентности».2. Посев на искусственные питательные среды, выделение чистой культуры и идентификация возбудителя
Питательные среды для первичного посева клинического материала и условия инкубации должны обеспечивать возможность выделения максимально широкого круга потенциальных возбудителей.
Выбор сред и условий инкубации определяется видом клинического материала, диагнозом инфекционной патологии и предполагаемыми возбудителями, задачами МИ, а также наличием дополнительных факторов риска присутствия нетипичных и/или резистентных возбудителей, и проводится в соответствии со стандартными процедурами лаборатории.
Внимание!
Выделение возбудителя в чистой культуре необходимо для выполнения фенотипического определения чувствительности к АМП и является обязательным условием осуществления мониторинга АР.3. Оценка клинического значения выделенных микроорганизмов
Проводится на основании результатов МИ с учетом клинико-демографических, эпидемиологических данных:
- видов выделенных микроорганизмов, их количественного соотношения при наличии ассоциаций;
- количества выделенных видов микроорганизмов;
- соответствие видов выделенных микроорганизмов локализации инфекции;
- соответствие видов выделенных микроорганизмов и результатов микроскопического исследования (обязательно: при исследовании крови, материала нижних дыхательных путей; рекомендуется при исследовании других видов материала по возможности);
- факторов риска развития инфекции и/или наличия резистентных возбудителей.
4. Определение чувствительности к антибиотикам и выявление механизмов резистентности клинически значимых возбудителей
Для мониторинга АР необходимо использовать результаты определения чувствительности возбудителей, выделенных в чистой культуре. Данные, полученные с помощью прямого определения чувствительности возбудителей в гемокультуре, не рекомендуется включать в мониторинг АР.
Молекулярно-генетические методы идентификации и выявления детерминант устойчивости возбудителей к АМП могут быть использованы как напрямую в клиническом материале, так и в выделенной чистой культуре возбудителя. Результаты выявления механизмов устойчивости к АМП молекулярно-генетическими методами также могут учитываться в процессе мониторинга, но они не заменяют в полном объеме и не исключают необходимости выполнения стандартных фенотипических методов определения чувствительности к АМП.
5. Формирование ответа (отчета) о результатах микробиологического исследования
В отчет о результатах индивидуального МИ следует включать информацию:
- результат микроскопического исследования (если проводилось);
- вид и количество (при количественном или полуколичественном исследовании) выделенных возбудителей, имеющих клиническое значение;
- результаты определения чувствительности к антибиотикам возбудителей, имеющих клиническое значение:
- клиническая категория чувствительности (Ч/S, У/I, Р/R, если установлены);
- значение МПК, мг/л (если определено);
- сравнение значения МПК с эпидемиологическими точками отсечения (где применимо при отсутствии клинических категорий чувствительности, см. раздел 3.5.);
- результаты выявления механизмов резистентности;
- дополнительные комментарии при необходимости.
Внимание!
Значение диаметров зон подавления роста, мм, не рекомендуется включать в отчет о результатах микробиологического исследования для лечащего врача. Но, как значения МПК, так и значения диаметров зон подавления роста, необходимо регистрировать в первичной документации лаборатории (протоколах исследования) на бумажных и/или электронных носителях.6. Организация, хранение данных
В рамках лаборатории могут использованы различные варианты работы с данными. Подробная информация представлена в разделе 4.1. Сценарии потоков данных.
3.5 Методы определения чувствительности микроорганизмов к антимикробным препаратам и интерпретация результатов
Основной целью оценки чувствительности микроорганизмов к антибиотикам является прогнозирование их эффективности при лечении инфекций у конкретных пациентов. Определение чувствительности также проводят с целью эпидемиологического наблюдения за распространением резистентности среди микроорганизмов и в процессе изучения новых препаратов.
Использование унифицированных методов определения чувствительности и подходов к интерпретации результатов является необходимым условием не только для правильного выбора АМТ у конкретного пациента, но и для формирования эффективной системы эпидемиологического наблюдения за антибиотикорезистентностью.
3.5.1 Методы определения чувствительности к антимикробным препаратам
Основным параметром, характеризующим взаимоотношения между микроорганизмом и антибиотиком, является величина минимальной подавляющей концентрации (МПК) препарата. МПК – это минимальная концентрация антибиотика, подавляющая видимый рост микроорганизма при его культивировании in vitro в стандартных условиях [32] . Относительно значений МПК, установленных референтным методом микроразведений, «калибруются» как наиболее распространенный в рутинной практике диско-диффузионный метод (ДДМ), так и различные коммерческие методы определения чувствительности [33] .
Результаты оценки чувствительности бактерий, полученные с помощью референтного метода, используют для обоснования микробиологических и клинических критериев оценки чувствительности [34] .
3.5.1.1 Метод последовательных разведений

Основным методом определения МПК является метод последовательных разведений. Для определения МПК заданные концентрации антибиотика (чаще всего с 2-кратным шагом) вносят в питательную среду, которую затем засевают культурой исследуемого микроорганизма и после инкубации оценивают наличие или отсутствие видимого роста в присутствии различных концентраций антибиотика. Известны два основных варианта постановки метода последовательных разведений: в агаре и в бульоне. Метод разведений в агаре в настоящее время используется в основном для определения чувствительности к отдельным антибиотикам, в частности, к фосфомицину. Метод последовательных разведений в бульоне, в свою очередь, может выполняться в макрои микро-варианте (в объеме ≤ 0,2 мл).
Метод последовательных микроразведений в бульоне является референтным методом определения МПК и регламентируется международным стандартом ISO 20776-1:2019 («Clinical laboratory testing and in vitro diagnostic test systems – Susceptibility testing of infectious agents and evaluation of performance of antimicrobial susceptibility test devices – Part 1: Reference method for testing the in vitro activity of antimicrobial agents against rapidly growing aerobic bacteria involved in infectious diseases») [35] . В Российской Федерации Приказом Федерального агентства по техническому регулированию и метрологии Российской Федерации от 23 ноября 2010 г. № 499-ст [36] утвержден и введен в действие Национальный Стандарт ГОСТ Р ИСО 20776-1-2010, идентичный международному стандарту ISO 20776-1 (в редакции 2006 г.).
Преимущества:
- Количественный (МПК – прямая количественная характеристика in vitro активности АМП);
- Стандартизированный;
- Гибкий (набор антибиотиков, широкий диапазон концентраций);
- Высокая воспроизводимость результатов;
- Возможность исследования чувствительности у большинства клинически значимых бактерий.
Ограничения:
- Трудоемкость;
- Высокие требования к квалификации и опыту персонала;
- Высокая стоимость (субстанции антибиотиков).
В настоящее время для практических лабораторий доступен целый ряд суррогатных методов определения МПК с использованием коммерческих расходных материалов, например: коммерческие варианты метода микроразведений в бульоне (автоматизированные и без автоматизации).
Преимущества:
- Меньше требований к контролю качества определения чувствительности в лаборатории (но не исключает его полностью!) при соблюдении условий транспортировки и хранения;
- При наличии программного обеспечения – автоматизированный учет результатов;
- Снижение трудозатрат;
- Не требуют высокой квалификации персонала.
Ограничения:
- Невозможность определения чувствительности всех клинически значимых видов бактерий;
- Фиксированный набор и ограниченный диапазон разведений антибиотиков;
- Относительно высокая стоимость.
3.5.1.2 Метод градиентной диффузии
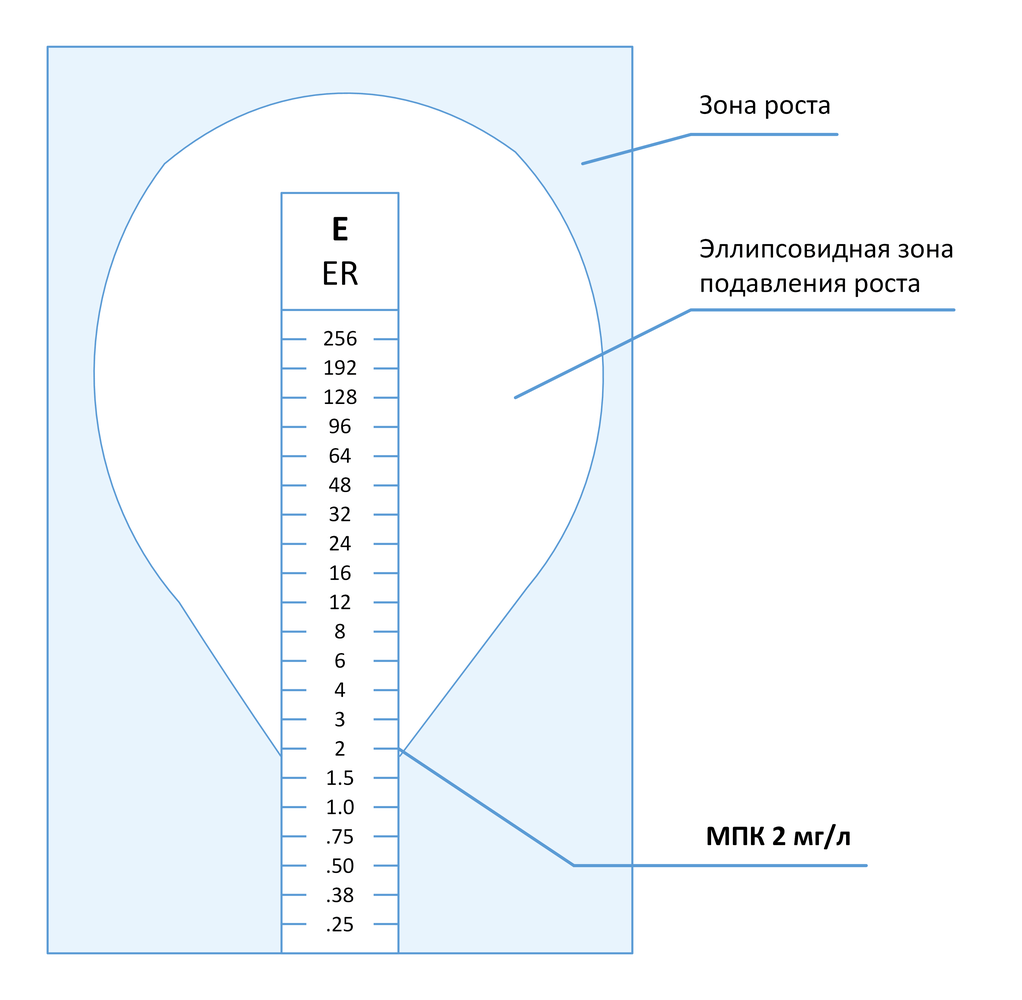
Метод градиентной диффузии представляет собой комбинацию методов последовательных разведений и диффузии антимикробных препаратов в агаре [37] . Коммерческие тесты, реализующие данный подход (Etest и M.T.S.), представляют собой тонкие полоски из инертного материала, на одну сторону которых нанесена шкала значений МПК и код препарата, а на другую – градиент концентраций препарата в сухом и стабилизированном виде. При наложении тест-полоски на поверхность агара, предварительно инокулированного тестируемой культурой микроорганизма, происходит высвобождение препарата и формирование стабильного и непрерывного градиента его концентраций в агаре. После инкубации вдоль тест-полоски формируется эллипсовидная зона подавления роста, в месте пересечения которой с нанесенной на полоску шкалой считывается значение МПК (в общем случае).
Преимущества:
- Высокая воспроизводимость результатов;
- Количественная оценка (определение МПК);
- Стандартизация;
- Гибкость;
- Возможность тестирования большинства клинически значимых видов бактерий;
- Широкий диапазон концентраций;
- Технологическая простота.
Ограничения:
- Высокая стоимость.
Качество коммерческих продуктов должно быть гарантировано производителем, а контроль качества результатов, получаемых при их использовании в лаборатории, является ответственностью пользователей.
3.5.1.3 Диско-диффузионный метод
Диско-диффузионный метод (ДДМ) является одним из первых методов определения чувствительности к антибиотикам и до настоящего времени остается наиболее распространенным в практических бактериологических лабораториях. Метод может применяться для исследования большинства бактериальных возбудителей, в том числе и наиболее распространенных бактерий со сложными питательными потребностями, является универсальным для широкого круга антибиотиков, не требует использования специального оборудования и, при условии строгого соблюдения процедуры тестирования, а также использования качественных материалов (дисков и сред), обеспечивает получение достоверных результатов.
Преимущества:
- Высокая стандартизация (при соблюдении методики исследования);
- Высокая воспроизводимость;
- Гибкость (широкий спектр и различные комбинации антибиотиков, возможность выявления отдельных механизмов резистентности);
- Технологическая доступность;
- Относительно низкая стоимость;
- Возможность автоматизации этапа учета и интерпретации результатов.
Ограничения:
- Непрямой полуколичественный метод;
- Используется преимущественно для быстро растущих бактерий;
- Необходимость строгого контроля качества;
- Невозможность достоверного определения чувствительности к отдельным антибиотикам (например, полимиксинам, гликои липопептидам).
3.5.2 Интерпретация результатов определения чувствительности к антимикробным препаратам
Для обеспечения точности и сопоставимости результатов определения чувствительности к АМП необходимо использовать научно-обоснованные международные стандарты, которые содержат гармонизированные пограничные значения для интерпретации результатов.
В настоящее время в мире общепризнанными являются стандарты Института по клиническим и лабораторным стандартам США (CLSI) и Европейского комитета по определению чувствительности к антимикробным препаратам (EUCAST) [38] .
Основой рекомендаций по определению чувствительности в РФ с 2014 года является комплекс подходов к оценке чувствительности и интерпретации результатов, предлагаемый EUCAST.
Главная идея EUCAST заключается в разделении понятий микробиологической и клинической чувствительности/устойчивости микроорганизмов. Для каждого из понятий используются отдельные пограничные значения:
- для микробиологической чувствительности – эпидемиологические точки отсечения;
- для клинической чувствительности – клинические пограничные значения МПК и диаметров зон подавления роста.
Пограничные значения являются справочной информацией.
3.5.2.1 Эпидемиологические точки отсечения
С микробиологической точки зрения в пределах популяций отдельных видов бактерий EUCAST предлагает выделять следующие типы [39] :
- Дикий тип (wild type – WT), к которому относятся микроорганизмы, лишенные мутационных или других приобретенных механизмов устойчивости к конкретному антибиотику.
- Недикий тип (non-wild type – NWT), к которому относятся микроорганизмы, обладающие мутационными или другими приобретенными механизмами устойчивости к конкретному антибиотику.
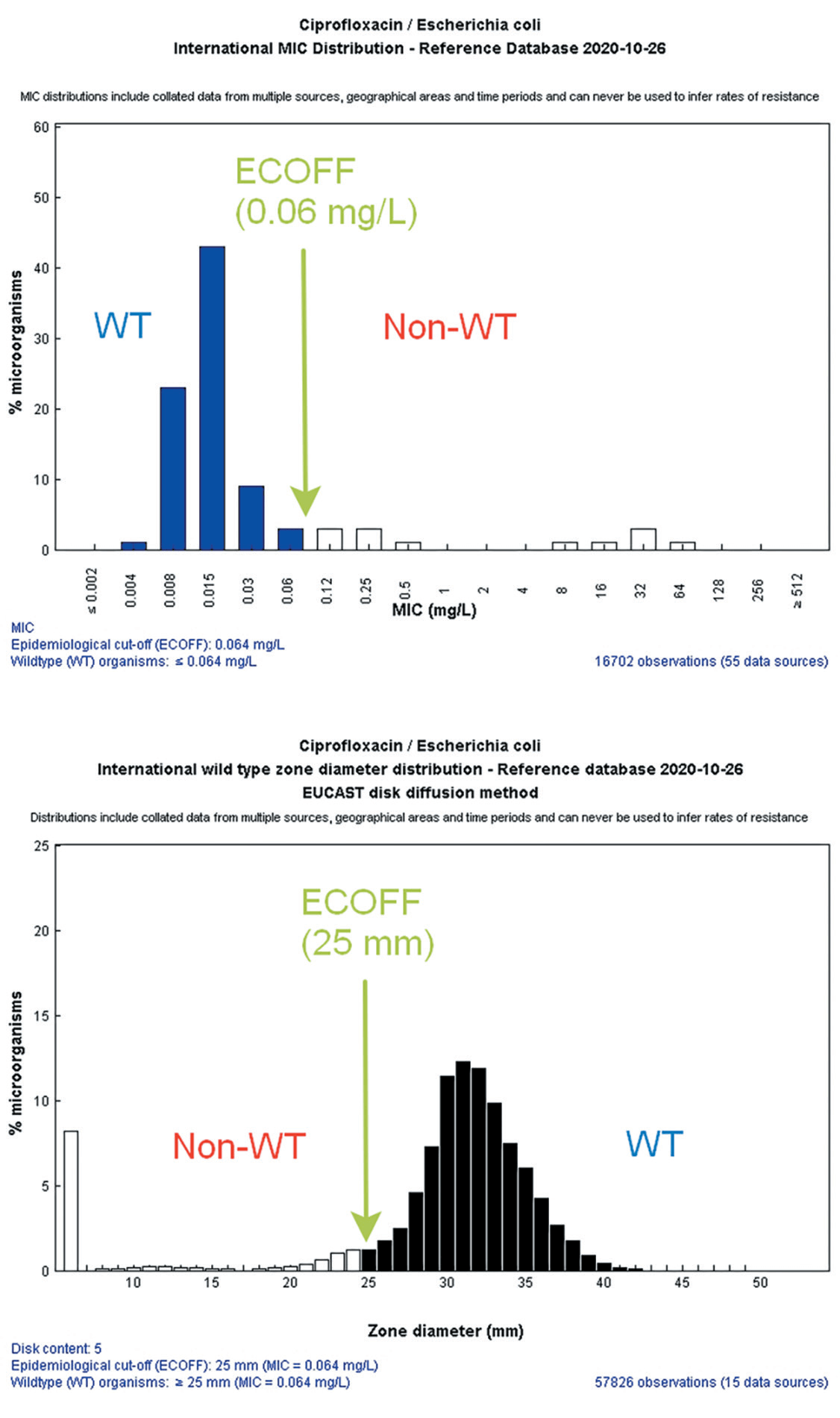
Принадлежность микроорганизма к одному из данных типов (дикий и недикий) оценивается на основании пороговых значений МПК антибиотиков, получивших название «эпидемиологические точки отсечения» (epidemiological cut-off values, ECOFF). ECOFF – это максимальное значение МПК (или наименьшее значение диаметра зоны подавления роста) конкретного АМП среди штаммов микроорганизмов в пределах одного вида, у которых отсутствует фенотипически выраженная устойчивость.
Значения ECOFF для конкретных комбинаций микроорганизм-антибиотик являются справочными и определяются статистическими методами на основании анализа характера распределения МПК антибиотика в отношении репрезентативной выборки изолятов соответствующего микроорганизма [40] . Эти значения являются постоянными видовыми признаками микроорганизмов и не зависят от изменяющихся обстоятельств. Гистограммы и таблицы распределения МПК (а также диаметров зон подавления роста, полученных с использованием ДДМ EUCAST) основных антибиотиков в отношении значительной части возбудителей инфекционных заболеваний человека доступны на веб-сайте EUCAST (http://mic.eucast.org). Значения ECOFF используются для дифференциации микроорганизмов, обладающих и не обладающих приобретенными механизмами резистентности, и могут отличаться от пограничных значений, используемых для определения клинических категорий чувствительности к антибиотику.
3.5.2.2 Клинические категории чувствительности
С практической точки зрения наиболее важной является классификация возбудителей инфекций по клиническим категориям чувствительности. Данные категории определяют вероятность достижения клинического эффекта при применении препарата. На протяжении ряда лет определения клинических категорий подвергались неоднократному пересмотру. С 2019 г. EUCAST использует следующие определения клинических категорий чувствительности [38] :
- Чувствительный при стандартном режиме дозирования (Ч) / Susceptible, standard dosing regimen (S). Микроорганизм оценивается как «Чувствительный при стандартном режиме дозирования» при высокой вероятности эффективности терапии при стандартном режиме дозирования.
- Чувствительный при увеличенной экспозиции антимикробного препарата (У) / Susceptible, Increased exposure (I). Микроорганизм оценивается как «Чувствительный при увеличенной экспозиции», при высокой вероятности эффективности терапии при увеличении экспозиции препарата путем коррекции режима дозирования или благодаря его концентрации в очаге инфекции [41] .
- Резистентный (Р) / Resistant (R). Микроорганизм оценивается как «Резистентный» при высокой вероятности терапевтической неудачи даже при увеличенной экспозиции препарата.
Для определения клинических категорий чувствительности устанавливаются клинические пограничные значения МПК и диаметров зон подавления роста. С целью определения пограничных значений проводятся исследования взаимосвязи между величиной МПК антибактериального препарата в отношении возбудителя, фармакокинетическими/фармакодинамическими характеристиками препарата и эффективностью лечения.
Клинические пограничные значения (МПК и ДДМ) могут пересматриваться при появлении новых данных о фармакокинетике и фармакодинамике антибиотиков и рекомендаций по режиму их применения. Ежегодно EUCAST публикует таблицы пограничных значений МПК и диаметров зон подавления роста для определения клинических категорий чувствительности бактерий к антимикробным препаратам (The European Committee in Antimicrobial Susceptibility Testing. Breakpoint tables for interpretation of MICs and zone diameters), которые находятся в свободном доступе на сайте www.eucast.org.
Важно отметить что категория Чувствительный при увеличенной экспозиции антимикробного препарата (У) / Susceptible, Increased exposure (I) была введена EUCAST взамен ранее использованной Умеренно-резистентный / Intermediate. Одновременно с введением новых определений категорий чувствительности были соответствующим образом изменены пограничные значения (МПК и ДДМ) для некоторых антибиотиков. Поэтому новые определения категорий чувствительности могут использоваться только при условии соблюдения методологии исследования и оценки результатов в соответствии с новыми рекомендациями EUCAST (версии начиная с 2019 г.).
Новые определения предполагают, что для терапии могут быть использованы антибиотики, чувствительность к которым была оценена как Ч (S), так и У (I). Однако категория У (I) означает, что экспозиция препарата должна быть увеличена в соответствии с инструкцией по применению препарата. В большинстве случаев (за исключением инфекций мочевых путей) это требует увеличения дозы, уменьшения интервала дозирования или изменения пути введения, например с перорального на в/в или с короткой в/в инфузии на продленную инфузию.
Для препаратов, экспозиция которых не может быть значимо увеличена, категории У (I) не существует.
3.5.3 Выбор антимикробных препаратов для определения чувствительности
Создание единого перечня АМП для определения чувствительности во всех лабораториях является трудно выполнимой задачей.
Основными причинами этого являются:
- разнообразие имеющихся на рынке АМП;
- различия между лечебными учреждениями по контингенту пациентов;
- различная этиологическая структура инфекций в разных регионах и медицинских организациях;
- распространенность механизмов приобретенной резистентности.
В то же время решение задач эпидемиологического наблюдения за антибиотикорезистентностью требует использования унифицированной панели АМП, включающей все группы антимикробных препаратов.
Согласно современным представлениям, правильно подобранный набор антибиотиков для тестирования в совокупности с корректной идентификацией выделенного возбудителя позволяет с высокой долей вероятности предположить наличие механизмов резистентности у исследуемого изолята и предсказать его чувствительность/резистентность к другим, близким по структуре и/или механизму действия АМП. Эти принципы лежат в основе правил интерпретации результатов определения чувствительности бактерий к антибиотикам [42] .
3.5.3.1 Принципы выбора антибиотиков для определения чувствительности
1. Проводить идентификацию микроорганизма-возбудителя.
Без видовой идентификации бактерии нельзя точно проанализировать профиль резистентности в силу наличия природных и множественных приобретенных механизмов резистентности. Клиническое значение одного и того же механизма резистентности неодинаково у разных видов бактерий. Также и резистентность, обусловленная одной и той же детерминантой, фенотипически может легко выявляться у одних видов и с трудом – у других.
С другой стороны, информация о природной резистентности может быть исключительно важной для выяснения или подтверждения видовой идентификации. По этой причине, ряд антибиотиков, которые не используются для лечения инфекции, вызванной предполагаемым возбудителем, могут включаться в набор для определения чувствительности представителей некоторых определенных видов и родов.
2. Выбирать АМП, активные в отношении выделенного микроорганизма.
Определение чувствительности к антибиотикам, природно неактивным в отношении данного вида (группы) бактерий, не имеет терапевтического значения. Природная (врожденная) резистентность к АМП, в противовес приобретенной и/или мутационной резистентности, является свойством всех или почти всех изолятов бактериального вида. Примерами природной резистентности является резистентность представителей порядка Enterobacterales к гликопептидам и линезолиду, Proteus mirabilis – к нитрофурантоину и колистину, Serratia marcescens – к колистину, Stenotrophomonas maltophilia – к карбапенемам, грамположительных бактерий – к азтреонаму, энтерококков – к фузидиевой кислоте и т.д. Подробная информация о природной устойчивости доступна на сайте EUCAST в разделе «Expert rules and intrinsic resistance» (https://www.eucast.org/expert_rules_and_intrinsic_resistance).
3. Выбирать один антибиотик в качестве индикаторного.
Объединение антибиотиков в группы и классы основано, прежде всего, на их структурном сходстве и подобном механизме действия. Некоторые классы антибиотиков, такие как макролиды и линкозамиды, несмотря на различную химическую структуру, имеют одну и ту же мишень действия внутри бактериальной клетки и, таким образом, могут рассматриваться в свете интерпретации результатов определения чувствительности in vitro, как единая функциональная группа. Кроме того, в пределах некоторых групп АМП можно выделить подгруппы препаратов, в отношении которых бактерии проявляют полную перекрестную резистентность. Следовательно, результаты определения чувствительности могут и должны интерпретироваться для класса или группы антибиотиков, а не индивидуально для каждого препарата.
В этих случаях на практике достаточно оценивать чувствительность только к одному АМП данной группы. Такой препарат должен наилучшим образом выявлять механизм(ы), определяющий(ие) устойчивость возбудителя ко всем или большинству представителей этой группы, даже если он не применяется в терапевтических целях. Определение чувствительности in vitro к другим представителям данной группы не требуется, и более того, в отдельных случаях может привести к получению недостоверных результатов. Это объясняется плохой воспроизводимостью результатов при исследовании некоторых препаратов in vitro, и/или невозможностью провести надежную корреляцию между результатами определения чувствительности in vitro и клинической эффективностью.
Например, оксациллин и цефокситин, используются для выявления резистентности ко всем β-лактамам у стафилококков (MRSA), при этом для тестирования стафилококков in vitro не используется ни один другой β-лактамный антибиотик; оксациллин используется для скрининга резистентности к пенициллину у пневмококков; для скрининга наличия ESBL у энтеробактерий рекомендуется использовать как минимум два цефалоспорина.
Индикаторный препарат
представитель класса или группы антибиотиков, по результату определения чувствительности к которому можно с высокой степенью вероятности судить о чувствительности (резистентности) не только к данному препарату, но и к родственным антибиотикам.4. Учитывать дополнительную информацию.
При выборе АМП для тестирования необходимо учитывать информацию о типе инфекции (внебольничная или нозокомиальная), распространенности резистентных штаммов в стране, регионе и стационаре. Это позволит составить оптимальную панель АМП для определения чувствительности в каждом конкретном лечебном учреждении.
3.5.3.2 Перечень антибиотиков, рекомендованный для проведения эпидемиологического наблюдения за антибиотикорезистентностью приоритетных возбудителей
Предлагаемый перечень АМП может быть использован как при определении чувствительности с терапевтической целью, так и для проведения эпидемиологического наблюдения за АР. Данный перечень не является исчерпывающим. При появлении новых данных и новых АМП, а также в зависимости от локальных приоритетов, перечень может быть пересмотрен [43] .
Ниже указаны приоритетные для мониторинга АР препараты в зависимости от вида и/или группы микроорганизмов. В первом столбце («Определение чувствительности in vitro к препаратам») указаны приоритетные для мониторинга АР препараты, во втором столбце («Результат «Р» распространяется на препараты») указаны препараты/группы препаратов к которым с высокой степенью вероятности ожидается наличие резистентности (категория Р/R) при резистентности (категория Р/R) к препарату в первом столбце. Таким образом, используя для мониторинга АР указанные препараты из первого столбца можно прогнозировать (экстраполировать) наличие резистентности (категория Р/R) к другим препаратам. Если наименования препаратов в первом и втором столбце полностью совпадают, то наличие резистентности (категория Р/R) распространяется только на данный препарат (препараты из этого же класса следует оценивать отдельно). Следует отметить, что представленные таблицы составлены только для прогнозирования (экстраполяции) категории Р (R), экстраполяция категорий Ч (S) и УР (I) зависит от конкретных механизмов резистентности и их комбинаций, что требует отдельного рассмотрения каждого случая.
Enterobacterales
| Определение чувствительности in vitro: | Результат «Р» распространяется на препараты: |
| Ампициллин или амоксициллин |
Аминопенициллины (ампициллин, амоксициллин), цефалоспорины I поколения
|
|
Амоксициллин/ клавулановая кислота |
Амоксициллин/клавулановая кислота, ампициллин/сульбактам
|
| Пиперациллин-тазобактам | Пиперациллин/тазобактам, амоксициллин/клавулановая кислота, ампициллин/сульбактам, цефоперазон-сульбактам |
| Цефотаксим или цефтриаксон |
Цефалоспорины III поколения (цефотаксим, цефтриаксон)
|
| Цефтазидим |
Цефтазидим
|
| Цефепим |
Цефепим
|
| Цефтазидим-авибактам |
Цефтазидим/авибактам
|
| Гентамицин или тобрамицин | Гентамицин, тобрамицин |
| Амикацин | Амикацин |
| Ципрофлоксацин или левофлоксацин |
Фторхинолоны (ципрофлоксацин, левофлоксацин, моксифлоксацин и др.)
|
| Пефлоксацин (ДДМ) скрининг |
Фторхинолоны (ципрофлоксацин, левофлоксацин, моксифлоксацин и др.)
|
| Имипенем | Имипенем |
| Меропенем |
Меропенем, дорипенем, эртапенем
|
| Эртапенем |
Эртапенем
|
| Колистин |
Полимиксины (колистин, полимиксин В)
|
| Фосфомицин |
Фосфомицин
|
| Нитрофурантоин |
Нитрофурантоин
|
P. aeruginosa
| Определение чувствительности in vitro: | Результат «Р» распространяется на препараты: |
| Пиперациллин-тазобактам | Пиперациллин/тазобактам |
| Цефтазидим | Цефтазидим |
| Цефепим | Цефепим |
| Азтреонам | Азтреонам |
| Цефтолозан-тазобактам |
Цефтолозан/тазобактам
|
| Цефтазидим-авибактам |
Цефтазидим/авибактам
|
| Гентамицин или тобрамицин | Гентамицин, тобрамицин |
| Амикацин | Амикацин |
| Ципрофлоксацин или левофлоксацин | Фторхинолоны (ципрофлоксацин, левофлоксацин) |
| Имипенем | Имипенем |
| Меропенем | Меропенем, дорипенем |
| Колистин |
Полимиксины (колистин, полимиксин В)
|
Acinetobacter spp.
| Определение чувствительности in vitro: | Результат «Р» распространяется на препараты: |
| Гентамицин или тобрамицин | Гентамицин, тобрамицин |
| Амикацин | Амикацин |
| Ципрофлоксацин или левофлоксацин | Фторхинолоны (ципрофлоксацин, левофлоксацин) |
| Имипенем | Имипенем |
| Меропенем | Меропенем |
| Колистин |
Полимиксины (колистин, полимиксин В)
|
|
Триметоприм/ сульфаметоксазол |
Триметоприм/сульфаметоксазол |
S. aureus
| Определение чувствительности in vitro: | Результат «Р» распространяется на препараты: |
|
Цефокситин
(ДДМ) скрининг Оксациллин (МПК) скрининг |
Антистафилококковые бета-лактамы (оксациллин, амоксициллин/клавулановая кислота, ампициллин/сульбактам, цефазолин, цефуроксим, карбапенемы), кроме анти-MRSA цефемов (цефтаролин, цефтобипрол)
|
| Цефтаролин |
Цефтаролин
|
| Ципрофлоксацин или левофлоксацин или офлоксацин | Фторхинолоны (ципрофлоксацин, левофлоксацин, офлоксацин) |
| Норфлоксацин (ДДМ) скрининг | Скрининг резистентности к ципрофлоксацину, левофлоксацину, офлоксацину, моксифлоксацину |
| Ванкомицин |
Ванкомицин
|
| Линезолид | Линезолид |
| Рифампицин | Рифампицин |
| Эритромицин |
Макролиды (эритромицин, азитромицин, кларитромицин, рокситромицин и др.)
|
| Клиндамицин |
Клиндамицин, линкомицин, макролиды
|
| Гентамицин | Аминогликозиды |
S. pneumoniae
| Определение чувствительности in vitro: | Результат «Р» распространяется на препараты: |
|
Оксациллин
(ДДМ) скрининг |
Первичный скрининг для выявления возможной резистентности к другим бета-лактамам (пенициллин, ампициллин, амоксициллина, амоксициллин/клавулановая кислота, ампициллин/сульбактам, пиперациллин, пиперациллин/тазобактам, цефотаксим, цефтриаксон, цефепим, цефтаролин, цефтобипрол, эртапенем, имипенем, меропенем) |
| Пенициллин (МПК) |
Пенициллин
|
| Цефотаксим или цефтриаксон |
Цефотаксим, цефтриаксон
|
| Цефтаролин |
Цефтаролин
|
| Эртапенем |
Эртапенем
|
| Эритромицин |
Макролиды (эритромицин, кларитромицин, азитромицин, рокситромицин и др.)
|
| Клиндамицин |
Клиндамицин, линкомицин, макролиды
|
| Левофлоксацин или моксифлоксацин | Левофлоксацин, моксифлоксацин |
| Норфлоксацин (ДДМ) скрининг | Скрининг резистентности к левофлоксацину, моксифлоксацину |
E. faecalis, E. faecium
| Определение чувствительности in vitro: | Результат «Р» распространяется на препараты: |
| Ампициллин или амоксициллин | Аминопенициллины (ампициллин, амоксициллин) |
| Гентамицин (высокая концентрация) скрининг |
Гентамицин
|
| Ципрофлоксацин или левофлоксацин |
Ципрофлоксацин, левофлоксацин
|
| Норфлоксацин (ДДМ) скрининг |
Скрининг резистентности к ципрофлоксацину, левофлоксацину
|
| Ванкомицин | Ванкомицин |
| Линезолид | Линезолид |
3.5.3.3 Стратегия интерпретации результатов определения чувствительности
Основой стратегии интерпретации результатов определения чувствительности является составление профиля чувствительности исследуемого образца не только на основании результатов изучения отдельных антибиотиков, но и механизмов резистентности, лежащих в его основе.
Стратегия состоит из трех этапов:
- Характеристика фенотипа резистентности на основании анализа полученных результатов исследований выбранного набора антибиотиков;
- Предположение о наличии определенного механизма резистентности на основе фенотипа;
- Прогнозирование фенотипа резистентности к другим антибиотикам, исходя из предполагаемого механизма резистентности.
Оптимальное выявление и характеристика фенотипа резистентности предполагает изучение минимального количества антибиотиков, принадлежащих к одному и тому же классу, в случае различных механизмов резистентности (например, аминогликозидов) или репрезентативных представителей большой группы (β-лактамы).
Наиболее полное и систематизированное описание данной стратегии представлено в документе Европейского комитета по определению чувствительности (EUCAST), известном под названием «Экспертные правила определения чувствительности к антибиотикам EUCAST» [44] .
3.6 Методы выявления механизмов резистентности, имеющих клиническое и эпидемиологическое значение
Выявление отдельных механизмов и детерминант (генетических маркеров) резистентности за исключением редких случаев (например, выявления mecA/mecC у стафилококков [45] ) не заменяет фенотипическое определение чувствительности, поскольку фенотип резистентности может быть связан с наличием множества различных генетических факторов, в том числе, неизвестных. Однако определение механизмов резистентности имеет важное значение, как для выбора и оптимизации АМТ у отдельных пациентов, так и для долгосрочного мониторинга антибиотикорезистентности [46] . Несмотря на кажущиеся различия, эти цели являются тесно взаимосвязанными, поскольку накопление регулярных (рутинных) данных о выявлении микроорганизмов с важными механизмами резистентности у отдельных пациентов со временем формирует пул эпидемиологических данных, которые, в свою очередь, позволяют оценивать риски и вероятность возникновения инфекций, вызванных «проблемными» резистентными возбудителями, и выбирать наиболее эффективные препараты, в том числе для эмпирического применения, у других пациентов (в отделении, стационаре, регионе, и т.д.).
В подавляющем большинстве случаев стандартные фенотипические методы позволяют проводить эффективную и комплексную оценку чувствительности к антибиотикам. Однако в отдельных случаях стандартные методы определения чувствительности к антибиотикам являются недостаточно эффективными или субоптимальными для выявления определенных механизмов и детерминант резистентности вследствие вариабельности их фенотипического проявления in vitro (например, устойчивости к карбапенемам у энтеробактерий, вызванной продукцией OXA-48), или являются длительными, трудоемкими либо недоступными (например, для выявления мутационной устойчивости микобактерий к различным препаратам [47] , или мутационной устойчивости патогенных для человека микоплазм к макролидам и фторхинолонам). В таких случаях выявление генетических маркеров резистентности является более эффективным или более доступным для предсказания АР.
В то же время определение конкретного механизма резистентности часто невозможно только на основании оценки профиля чувствительности, особенно в рутинной практике. Например, дифференциация мутационных (утрата/изменение поринов, активация эффлюкса) и ферментативных (продукция карбапенемаз) механизмов устойчивости к карбапенемам у Pseudomonas aeruginosa или Klebsiella pneumoniae является важной (как для выбора терапии, так и для определения эпидемиологической значимости), но может быть крайне затруднительной или невозможной на основании оценки антибиотикограммы (спектра и уровня устойчивости к антибиотикам). В связи с этим выявление наиболее важных механизмов и детерминант резистентности требует использования дополнительных методов, фенотипических или молекулярно-генетических [48] [49] [50] .
3.6.1 Основные механизмы и детерминанты резистентности подлежащие мониторингу
В настоящее время новые технологии, включая методы амплификации нуклеиновых кислот (МАНК) и секвенирования, становятся более доступными для практического использования в области клинической микробиологии благодаря техническому усовершенствованию, автоматизации и снижению стоимости. Эти методы позволяют осуществлять выявление любых известных детерминант (генов и мутаций), связанных с АР. Важным преимуществом МАНК является возможность быстрого определения детерминант резистентности не только у микробных изолятов, выделенных в чистой культуре, но и в образцах нативного биоматериала или положительных гемокультур. Однако с практической точки зрения наиболее важным является выявление отдельных механизмов, которые имеют особое клиническое и/или эпидемиологическое значение. Такие механизмы и методы их выявления подробно описаны в руководстве «The EUCAST guidelines for detection of resistance mechanisms and specific resistances of clinical and/or epidemiological importance» [51] , а также в руководствах Всемирной организации здравоохранения (ВОЗ) по использованию молекулярно-генетических методов и геномного секвенирования в системе мониторинга антибиотикорезистентности [52] [53] .
Для осуществления эффективного контроля за распространением инфекций и обеспечения рационального применения антибиотиков в стационаре наиболее важным является мониторинг следующих механизмов и детерминант резистентности:
- приобретенных карбапенемаз у Enterobacterales, P. aeruginosa и Acinetobacter;
- β-лактамаз расширенного спектра у Enterobacterales;
- mcr-опосредованной (плазмидно-кодируемой) резистентности к полимиксинам у Грам(-) бактерий;
- mecA/mecC-опосредованной резистентности к беталактамам у S. aureus;
- vanA/vanB-опосредованной резистентности к гликопептидам у E. faecium и E. faecalis.
Спектр выявляемых в каждой лаборатории механизмов и детерминант резистентности может отличаться и должен учитывать локальные, региональные и национальные данные о распространенности фенотипической резистентности и отдельных генетических маркеров. Он может изменяться и дополняться со временем в случае выявления новых значимых механизмов (например, в случае распространения штаммов стафилококков, устойчивых к оксазолидинонам, должен осуществляться мониторинг плазмидных Cfr рРНК метилтрансфераз).
3.6.1.1 Выявление приобретенных карбапенемаз у Enterobacterales, P. aeruginosa и Acinetobacter
Карбапенемазы – крайне разнородная группа бактериальных ферментов, относящихся к трем молекулярным классам A, B и D и множеству генетических групп, которые отличаются от других β-лактамаз способностью расщеплять карбапенемы. Продукция карбапенемаз является наиболее эффективным и эпидемиологически значимым механизмом резистентности к карбапенемам у Грам(-) бактерий [54] [55] . У Pseudomonas и Acinetobacter spp. наличие приобретенных карбапенемаз обычно коррелирует с фенотипической устойчивостью к карбапенемам, однако карбапенемазопродуцирующие энтеробактерии (CPE) могут проявлять «чувствительность» к меропенему и имипенему при тестировании in vitro в соответствии с клиническими пограничными значениями МПК и ДДМ для резистентных штаммов. С другой стороны, резистентность низкого уровня к карбапенемам у энтеробактерий может быть вызвана альтернативными механизмами, которые имеют меньшее клиническое и эпидемиологическое значение. При наличии результатов определения чувствительности выявление продукции карбапенемаз должно проводиться не только для резистентных штаммов, но для всех изолятов Enterobacterales с МПК меропенема >0,125 мг/л или диаметром зоны подавления роста вокруг диска с меропенемом (10 мкг) <28 мм. В стационарах с высоким уровнем распространенности СРЕ выявление продукции карбапенемаз может проводиться для всех изолятов энтеробактерий до получения данных о чувствительности к карбапенемам. При наличии у пациента факторов риска инфицирования СPE выявление генов карбапенемаз может также осуществляться с использованием амплификационных молекулярно-генетических методов (МАНК) в образцах нативного биоматериала или положительных гемокультур до выделения возбудителя в чистой культуре [56] .
Методы выявления карбапенемаз включают [57] :
- Методы определения чувствительности к карбапенемам и их комбинациям с различными ингибиторами («двойные», «комбинированные » диски или определение МПК в присутствии ингибиторов) – оценка синергизма. В связи с необходимостью использования различных ингибиторов и относительно низкой специфичностью данные методы в настоящее время имеют ограниченное применение.
- Иммунохроматографические методы. Позволяют выявлять продукцию и дифференцировать наиболее распространенные типы карбапенемаз у микробных изолятов.
- Методы оценки in vitro гидролиза карбапенемов с использованием индикаторного штамма (CIM, eCIM, mCIM), MALDI-ToF масс-спектрометрии, pH-индикаторов (Carba-NP тест и др.), флуорогенных карбапенемов (СarbaLux).
- МАНК (ПЦР-РВ и др.) и методы секвенирования.
Использование селективных и хромогенных сред позволяет осуществлять предварительный скрининг изолятов энтеробактерий с вероятной продукцией карбапенемаз, но не является специфическим методом выявления их продукции. Рост бактериальных изолятов на селективных средах, содержащих карбапенемы, может быть связан с наличием различных механизмов помимо продукции карбапенемаз. Наличие карбапенемаз должно быть подтверждено с помощью тестов, перечисленных выше. Многие неферментирующие бактерии (Acinetobacter, Pseudomonas и др.) способны расти на селективных средах, предназначенных для выявления CPE, в отсутствии приобретенных механизмов устойчивости к карбапенемам.
При использовании МАНК важно учитывать данные распространенности различных типов карбапенемаз, которые могут существенно отличаться для разных видов бактерий и разных географических регионов (Таблица 3.6.1.).
Таблица 3.6.1.1 Встречаемость различных карбапенемаз у Enterobacterales, P. aeruginosa и Acinetobacter spp. в России [55] [58] [59]
| Молекулярный тип карбапенемаз | Enterobacterales | P. aeruginosa | Acinetobacter spp. |
|---|---|---|---|
| VIM | + | ++++ | + |
| IMP | + | + | + |
| NDM | +++ | + | + |
| OXA-48 | ++++ | - | - |
| OXA-23 | - | - | ++++ |
| OXA-24/40 | - | - | ++++ |
| OXA-58 | - | - | ++ |
| KPC | ++ | - | - |
| GES-5 (G170S) 1 | - | +++ | - |
1 коммерческие тесты для дифференциации ESBL и карбапенемаз группы GES в настоящее время недоступны в России.
- ++++
- наиболее частый тип карбапенемаз
- +++
- широкое распространение
- ++
- распространение в отдельных стационарах и городах
- +
- единичные случаи
3.6.1.2 Выявление ESBL у Enterobacterales
β-Лактамазы расширенного спектра (ESBL) – ферменты, гидролизующие пенициллины, цефалоспорины I-IV поколения и азтреонам, но не гидролизующие цефамицины и карбапенемы. Большинство ESBL относятся к молекулярному классу А и подавляются «классическими» ингибиторами (клавулановой кислотой, сульбактамом, тазобактамом), а также авибактамом. Продукция ESBL встречается у всех клинически значимых видов энтеробактерий, как в стационарах, так и во внебольничной среде.
Методы выявления ESBL включают:
- Методы определения чувствительности к оксииминобеталактамам и их комбинациям с клавулановой кислотой («двойные», «комбинированные» диски или определение МПК в присутствии клавулановой кислоты – оценка синергизма).
- Методы оценки in vitro гидролиза оксииминобеталактамов с использованием индикаторного штамма (модифицированный CIM тест), MALDI-ToF масс-спектрометрии, pH-индикаторов.
- Иммунохроматографические методы. Позволяют выявлять продукцию и наиболее распространенных ESBL группы CTX-M у микробных изолятов.
- МАНК (ПЦР-РВ и др.) и методы секвенирования.
ESBL относятся к множеству различных генетических групп (семейств). Поэтому для универсальной детекции ESBL в основном используются фенотипические тесты, основанные на выявлении синергизма оксииминобеталактамов с клавулановой кислотой. Наиболее распространенными в настоящее время являются ферменты CTX-M-группы, которые, в свою очередь, делятся на генетические кластеры CTX-M-1-, CTX-M-2-, CTX-M-8/25- и CTX-M-9-родственных ферментов (самые частые варианты CTX-M-15, -3, -9 и -14 относятся к кластерам CTX-M-1 и CTX-M-9). Реже встречаются ESBL SHV-типа (SHV-2, -5, -12 и др.) которые представляют собой мутантные производные SHV пенициллиназ (SHV-1, -11 и др.) – видоспецифических ферментов K. pneumoniae. ESBL TEM-типа являются мутантными производными широко распространенных плазмидно кодируемых TEM пенициллиназ. В России ESBL TEM-типа встречаются крайне редко, несмотря на то, что пенициллиназы (TEM-1 и др.) присутствуют у подавляющего большинства клинических штаммов. Доступные в настоящее время в России диагностические тесты на основе МАНК позволяют выявлять только гены CTX-M ESBL, но не позволяют дифференцировать мутантные варианты SHV и TEM с расширенным спектром активности. Поэтому использование молекулярно-генетических методов не заменяет фенотипическое тестирование для обнаружения ESBL. Тем не менее, МАНК, включая ПЦР-РВ, могут применяться для быстрой детекции генов CTX-M β-лактамаз, например, в образцах нативного биоматериала и положительных гемокультур.
3.6.1.3 Выявление mcr-опосредованной резистентности к полимиксинам у Грам(-) бактерий
Гены mcr кодируют фосфоэтаноламин-трансферазы, ферменты, осуществляющие модификацию липида A – основного структурного компонента липополисахарида (ЛПС) и вызывающие устойчивость к полимиксинам. Гены mcr имеют плазмидную локализацию и могут распространяться горизонтально между различными штаммами и видами Грам(-) бактерий. В настоящее время описано множество генетических вариантов mcr, которые относятся как минимум к семи различным генетическим кластерам. Происхождение mcr связано с мобилизацией и переносом на плазмиды хромосомных генов Moraxella (mcr-1,-2), Aeromonas veronii (mcr-3), Shewanella (mcr-4) 23, Raoultella ornithinolytica (mcr-8) и Salmonella enterica ser. Typhimurium (mcr-9). Ген mcr-1, описанный первым, является наиболее распространенным у энтеробактерий, прежде всего, у штаммов E. coli, выделяемых от животных, человека и из окружающей среды. Распространенность плазмидных mcr среди клинических штаммов варьирует в разных странах и регионах, но обычно не превышает распространенность других механизмов резистентности к полимиксинам (модификации ЛПС, эффлюкса), связанных с мутациями в хромосомных генах. В связи с этим детекция mcr имеет в основном эпидемиологическое значение (из-за возможности горизонтального распространения).
Методы выявления mcr включают:
- МАНК (ПЦР-РВ и др.) и методы секвенирования.
- Иммунохроматографические методы. Позволяют выявлять экспрессию mcr-1 у микробных изолятов.
- Методы определения чувствительности к полимиксинам в присутсвии цинк-хелатирующих ингибиторов mcr (ЭДТА, дипиколиновой кислоты).
Другие методы, применяемые для ускоренной диагностики резистентности к полимиксинам, например, оценка роста на селективных средах или в присутствии pH индикаторов (polymyxin NP test), детекция модификаций липида А с помощью MALDI-ToF масс-спектрометрии (MALDIxin тест), не являются специфичными для mcr.
3.6.1.4 Выявление mecA/mecC-опосредованной резистентности к беталактамам у Staphylococcus aureus
Наличие гена mecA, который кодирует дополнительный пенициллин-связывающий белок (ПСБ2а/2’) с низкой аффинностью к беталактамам, является основным механизмом устойчивости Staphylococcus aureus и других видов стафилококков ко всем беталактамам (кроме цефтаролина и цефтобипрола). Помимо mecA штаммы S. aureus, проявляющие фенотип метициллинорезистентности (MRSA), выделяемые от животных и реже от человека, могут нести ген mecC, кодирующий альтернативный вариант ПСБ2а. Гены mecA и mecC входят в состав генетических элементов, известных как стафилококковые хромосомные кассеты mec (SCCmec), и часто ассоциированы с генами резистентности к другим антибиотикам. S. fleurettii и S. xylosus содержат видоспецифические гены ПСБ, гомологичные mecA и mecC. SCCmec элементы, несущие эти гены, также широко распространены у других видов стафилокков.
Методы выявления mecA/mecC-опосредованной резистентности включают:
- Определение МПК оксациллина или цефокситина, скрининг чувствительности к цефокситину с использованием ДДМ.
- МАНК (ПЦР-РВ и др.) и методы секвенирования.
- Латексная агглютинация. Позволяет выявлять ПСБ2a у микробных изолятов, выделенных в чистой культуре.
Выявление mecA/mecC с помощью МАНК является референтным методом детекции MRSA и метициллинорезистентности у других видов стафилококков (MRS) [60] [61] . Несмотря на то, что до настоящего времени в России не были выявлены штаммы, несущие ген mecC, для мониторинга MRSA рекомендуется использовать тесты, обеспечивающие возможность его детекции наряду с mecA. Важным при использовании МАНК для прямого выявления mecA/mecC в клинических образцах является также возможность определения вида возбудителя, который является их источником (S. aureus или другие стафилокки), особенно при анализе колонизации MRSA, поскольку коагулазонегативные стафилококки, колонизирующие кожу и верхние дыхательные пути, являются более частым источником mec генов.
3.6.1.5 Выявление vanA/vanB-опосредованной резистентности к гликопептидам у Enterococcus faecium и E. faecalis
Гены vanA и vanB являются основными детерминантами резистентности к гликопептидам (ванкомицину) у E. faecium и реже у E. faecalis. Они входят в состав мобильных генетических кластеров, которые кодируют синтез измененных мишеней – пептидных остатков (-D-аланил-D-лактат) с низкой аффинностью связывания ванкомицина. Другие редкие приобретенные гены vanD и vanE, а также видоспецифические гены vanC E. gallinarum, E. casseliflavus и E. flavescens имеют меньшее клиническое и эпидемиологическое значение.
Методы выявления vanA/vanB-опосредованной резистентности включают:
- Определение МПК ванкомицина.
- МАНК (ПЦР-РВ и др.) и методы секвенирования.
Распространенность устойчивости к ванкомицину, связанной с vanA/vanB, значительно отличается в разных странах. В России резистентность к ванкомицину в основном встречается у нозокомиальных штаммов E. faecium в стационарах онкологического и гематологического профиля и значительно реже в других стационарах. Мониторинг vanA и vanB является важным в случае увеличения частоты обнаружения ванкомицинорезистентных энтерококков (VRE).
Таблица 3.6.1.2 Основные механизмы устойчивости к антибактериальным препаратам
| Гены | Микроорганизмы | Вероятная устойчивость к… | Комментарий |
|---|---|---|---|
| *blaVIM | Pseudomonas spp., Enterobacterales | Пенициллины, ингибиторозащищенные пенициллины, цефалоспорины I-V, цефоперазон-сульбактам, цефепим-сульбактам, цефтолозан-тазобактам, цефтазидим-авибактам, карбапенемы | Реже могут встречаться у других грамотрицательных бактерий |
| *blaIMP | Pseudomonas spp. | Пенициллины, ингибиторозащищенные пенициллины, цефалоспорины I-V, цефоперазон-сульбактам, цефепим-сульбактам, цефтолозан-тазобактам, цефтазидим-авибактам, карбапенемы | Реже могут встречаться у других грамотрицательных бактерий |
| *blaNDM | Enterobacterales | Пенициллины, ингибиторозащищенные пенициллины, цефалоспорины I-V, цефоперазон-сульбактам, цефепим-сульбактам, цефтолозан-тазобактам, цефтазидим-авибактам, карбапенемы | Реже могут встречаться у других грамотрицательных бактерий |
| *blaOXA-48 | Enterobacterales | Пенициллины, ингибиторозащищенные пенициллины, карбапенемы | Значительно реже могут встречаться у других грамотрицательных бактерий |
| *blaOXA-23 | Acinetobacter spp. | Карбапенемы | В отдельных регионах описано распространение у Proteus spp. |
| *blaOXA-24/40 | Acinetobacter spp. | Карбапенемы | |
| *blaOXA-58 | Acinetobacter spp. | Карбапенемы | В отдельных регионах мира описано распространение у Proteus spp. |
| *blaKPC | Enterobacterales | Пенициллины, ингибиторозащищенные пенициллины, цефалоспорины I-V, цефоперазон-сульбактам, цефепим-сульбактам, цефтолозан-тазобактам, карбапенемы | В отдельных регионах мира описаны случаи распространения у Pseudomonas spp. |
| *blaGESCarb | Pseudomonas spp. | Пенициллины, цефалоспорины III-IV, азтреонам, карбапенемы | Важно! Необходима дифференциация от родственных генов, кодирующих ESBL (blaGESESBL) |
| *blaTEMESBL | Enterobacterales | Пенициллины, цефалоспорины I-V, азтреонам | Важно! Необходима дифференциация от родственных генов, кодирующих пенициллиназы (blaTEMPen ) |
| *blaSHVESBL | Enterobacterales | Пенициллины, цефалоспорины I-V, азтреонам | Важно! Необходима дифференциация от родственных генов, кодирующих пенициллиназы (blaSHVPen) |
| *blaCTX-M-1 | Enterobacterales | Пенициллины, цефалоспорины I-V, азтреонам | |
| *blaCTX-M-2 | Enterobacterales | Пенициллины, цефалоспорины I-V, азтреонам | |
| *blaCTX-M-9 | Enterobacterales | Пенициллины, цефалоспорины I-V, азтреонам | |
| *mcr-1 | Enterobacterales | Полимиксины | Реже могут встречаться у других грамотрицательных бактерий. Другие группы генов mcr распространены в меньшей степени, их регулярный эпидемиологический мониторинг важен только в тех регионах, где выявлены случаи распространения. |
| *armA | Enterobacterales | Все аминогликозиды | |
| *rmtB | Enterobacterales | Все аминогликозиды | |
| *rmtC | Enterobacterales | Все аминогликозиды | |
| *rmtF | Enterobacterales | Все аминогликозиды | |
| *mecA | Staphylococcus aureus* | Все беталактамы, кроме цефтаролина и цефтобипрола | Эпидемиологический мониторинг у других видов рода Staphylococcus не является обязательным |
| *mecC | Staphylococcus aureus | Все беталактамы, кроме цефтаролина и цефтобипрола | Эпидемиологический мониторинг у других видов рода Staphylococcus не является обязательным |
| *vanA | Enterococcus faecium, Enterococcus faecalis | Гликопептиды | Эпидемиологический мониторинг у других видов рода Enterococcus не является обязательным |
| *vanB | Enterococcus faecium, Enterococcus faecalis | Гликопептиды | Эпидемиологический мониторинг у других видов рода Enterococcus не является обязательным |
* Группа, включающая ряд родственных генов
3.6.2 Учет и регистрация данных выявления механизмов и детерминант резистентности
3.6.2.1 Номенклатура и стандартизация
Стандартизация записей результатов исследования является абсолютно необходимой для их обобщения и автоматического анализа. В различных источниках и базах данных для обозначения одних и тех же генетических детерминант резистентности могут использоваться разные названия. Например распространенный ген aadA1, кодирующий аминогликозид-аденилтрансферазу ANT(3’’)-Ia, может также обозначаться как aadA, aadA1-pm, или aad(3’’). В связи с этим для указания генетических маркеров устойчивости к АМП (генов и мутаций) рекомендуется использовать единую международно принятую номенклатуру и базы данных AMRFinderPlus или ResFinder/PointFinder [62] [63] .
Гены и кодируемые ими ферменты, как правило, обозначаются по-разному, например, гены группы blaVIM и кодируемые ими металло-β-лактамазы группы VIM. При учете результатов необходимо соблюдать единообразие записей.
3.6.2.2 Информативность результатов
Разные методы обеспечивают разную точность указания вариантов (типов) детерминант резистентности. В частности, ПЦР обычно позволяет выявить ген и установить его принадлежность к определенной группе, в то время как методы секвенирования позволяют установить точный вариант (например, группа blaOXA-48, тип blaOXA-244). В различных ситуациях точность указания группы или конкретного варианта гена или мутации могут иметь разное значение. Например, результат выявления гена «blaOXA» без указания группы или определенного варианта является неинформативным, поскольку это обозначение является общим для разных генов, кодирующих природные и приобретенные β-лактамазы класса D с разным спектром активности: пенициллиназы, цефалоспориназы и карбапенемазы. В то же время, указание наличия гена группы blaNDM является достаточным для определения металло-β-лактамазы, которая вызывает устойчивость к пенициллинам, цефалоспоринам и карбапенемам.
3.6.2.3 Организация записей
Результаты выявления одних и тех же механизмов или детерминант, полученные с использованием разных методов, например, ПЦР, секвенирования, фенотипических тестов, рекомендуется записывать отдельно (в разных столбцах таблицы или полях базы данных). Примеры учета результатов определения детерминант резистентности представлены в Таблице 3.6.2.
В случае если один тест позволяет выявлять и дифференцировать несколько детерминант (например, мультиплексная ПЦР для детекции генов разных карбапенемаз), рекомендуется учитывать результат обнаружения каждой из них отдельно. Результаты выявления и аннотации множества детерминант, полученные с помощью методов геномного секвенирования (WGS), могут быть записаны в виде одной строки с разделителями между названиями отдельных генов или мутаций.
Анализ механизмов резистентности обычно проводится не для всех, а для определенных или выборочных изолятов. В связи с этим учет отрицательных результатов тестов столь же важен, как и положительных. Учет отрицательных результатов позволяет впоследствии различать случаи, когда соответствующие детерминанты резистентности отсутствовали у микробных изолятов, и когда тестирование не производилось, и, таким образом, правильно оценивать истинную распространенность детерминант.
Выявление с помощью МАНК маркера резистентности непосредственно в клиническом образце из локуса, который в норме не является стерильным, не означает его обязательное наличие у микробного изолята, который может быть выделен из данного локуса. Например, одновременное обнаружение с помощью ПЦР генов группы blaOXA-48 и blaVIM в эндотрахеальном аспирате и последующее выделение в культуре изолятов K. pneumoniae и P. aeruginosa не позволяет констатировать факт наличия у каждого из этих изолятов генов двух карбапенемаз. Каждый выделенный изолят должен быть исследован на наличие карбапенемаз, и результаты представлены отдельно.
Таблица 3.6.2. Примеры учета результатов определения детерминант резистентности
| ID | Вид МО | ПЦР_NDM | ПЦР_VIM | ПЦР_IMP | ПЦР_OXA-48-гр. | ПЦР_KPC | CIM-тест | ESBL_ДД | WGS_генотип |
|---|---|---|---|---|---|---|---|---|---|
| 1 | Klebsiella pneumoniae | Отр. | Отр. | Отр. | Положит. | Отр. | Положит. | Положит. | blaSHV-11|blaTEM-1|blaCTX-M-15|blaOXA-244|fosA|aadA2|qacEdelta1|sul1 |
| 2 | Pseudomonas aeruginosa | Отр. | Положит. | Отр. | Отр. | Отр. | Положит. | Нет данных | Нет данных |
| 3 | Escherichia coli | Отр. | Отр. | Отр. | Отр. | Отр. | Отр. | Положит. | Нет данных |
4. Подготовка данных для эпидемиологического анализа антибиотикорезистентности
4.1 Сценарии потоков данных
В зависимости от оснащения лаборатории, возможны различные способы организации потоков данных. При организации мониторинга АР важным является вопрос, как имеющимися программными средствами получить файл, пригодный для последующей обработки и анализа в системе AMRcloud или других программных комплексах.
Рассмотрим основные способы организации потоков данных в лаборатории:
- В лаборатории используется ЛИС/МИС и все данные регистрируются внутри этих систем;
- В лаборатории установлена программа WHONET, данные находятся в ней;
- В лаборатории отсутствуют какие-либо программные инструменты.
В первом случае можно посоветовать связаться с разработчиками используемой ЛИС/МИС и рассмотреть с ними возможность реализации нового формата отчета (т.е. «выгрузки» исходных данных), отвечающего требованиям к документу из раздела 4.2. Шаблон для сбора и ввода данных.
Если же создание такого отчета не представляется возможным, на помощь могут прийти сторонние инструменты, такие как программа BacLink (входит в состав программы WHONET), которая позволит преобразовать файл отчета в формат WHONET, далее из формата WHONET можно получить отчет в формате AMRcloud. Подробная информация представлена в приложении 6.4. Работа с BACLINK.
Во втором случае, если в лаборатории уже выстроена работа с программой WHONET, советуем обратиться к приложению 6.3. Работа с WHONET, в котором описан необходимый алгоритм получения отчета, пригодного для загрузки в AMRcloud.
Если же у лаборатории отсутствуют какие-либо программные инструменты, а микробиологический журнал ведется в таблице Excel, то настоятельно рекомендуется проверить его соответствие требованиям, предъявляемым к документу из раздела 4.2. Шаблон для сбора и ввода данных. Следование этим требованиям значительно повысит качество вводимых данных и упростит решение задач мониторинга АР.
Кроме того, рекомендуем обратить внимание на бесплатную программу WHONET (http://whonet.org), призванную организовать и упростить в лаборатории ввод, хранение и анализ данных АР. Данная программа имеет большое количество встроенных справочников и средств, упрощающих ввод данных МИ. Модули экспорта и анализа также разработаны с учетом особенностей данных АР и могут помочь в организации процесса мониторинга, а также содержат готовые шаблоны для подготовки файлов в формате международных проектов по мониторингу АР, таких как GLASS и CAESAR. Программа BacLink позволяет перенести накопленные данные в WHONET и продолжить с ними работу. Несомненно, такой переход может потребовать трудовых и временных затрат на освоение новых программ, но это существенно упростит организацию, хранение и анализ данных лаборатории и позволит быстрее выстроить систему мониторинга АР.
4.2 Шаблон для сбора и ввода данных
С целью стандартизации формата хранения данных мониторинга АР и их последующего анализа необходимо разработать шаблон для сбора и ввода данных. Разработка шаблона должна осуществляться одновременно с модификацией (или разработкой) бланка направления на МИ, т.к. последний является основным источником информации для шаблона данных. В данном разделе представлен базовый вариант шаблона данных, а также принципы его заполнения. Полный вариант, а также примеры справочников и словарь наименований антимикробных препаратов представлен в приложении 6.2. Работа с шаблоном, справочниками и словарями.
Таблица 4.2.1. Пример шаблона данных (расшифровка наименований и содержимого столбцов представлена в таблице 4.2.2.)
| Данные изолята | Данные пациента | Маркеры (резистентности/вирулентности) | Результаты чувствительности | ||||||||||||
| Идентификатор изолята | Дата | Вид_МО | Группа_МО | Географич. локализация | Локализация инфекции | Клинический материал | Географич. локализация | Отделение | Возраст пациента | Симптом заболевания | ESBL | MRSA | Antibiotic_mic | Antibiotic_sir | Antibiotic_dd |
| 1AB | 16.12.2018 | Klebsiella pneumoniae | Enterobacterales | Смоленск | Нижние дыхательные пути | Бронхоальвеолярный лаваж | Смоленск | Реанимация | 62 | Нет | CTX-M-15 | 8 | S | 6 | |
| 2CD | 16.12.2018 | Staphylococcus aureus | Staphylococcus | Смоленск | Кожа и мягкие ткани | Раневое отделяемое | Смоленск | Поликлиника | 20 | Нет | Положительный | 16 | R | 8 | |
Таблица 4.2.2. Основные характеристики столбцов и содержащихся в них данных
| Столбец | Примеры значений | Особенности |
| Идентификатор изолята | 1AB | Уникальный идентификатор |
| Дата | 16.12.2018 | Дата взятия материала Единый формат написания |
| Вид МО | Klebsiella pneumoniae | Полное наименование микроорганизма Английский вариант |
| Группа МО | Enterobacterales | Указать род/порядок и т.д. Единое оформление по всему столбцу Английский вариант |
| Antibiotic_mic | 8 |
Указать значения МПК по основному действующему веществу (например, триметоприм/сульфаметоксазол – МПК триметоприма)
Согласно международному соглашению рекомендуется использовать значения, соответствующие двукратным разведениям начиная с 1 мг/л. Для значений менее 0,25 мг/л рекомендуется использовать следующие значения 0.125→ 0.125, 0.0625→0.06, 0.03125→0.03, 0.015625→0.016, 0.0078125→0.008, 0.00390625→0.004 0.001953125→0.002 |
| Antibiotic_sir | S |
Указать один вариант:
|
| Antibiotic_dd | 6 | Указать диаметр зоны задержки роста по данным ДДМ. Целые числа, диапазон от 6 до 50 мм |
| Географическая локализация | Смоленск | Не рекомендуется использовать разное написание для одного географического объекта (если использовалась аббревиатура учреждения, то данное написание должно сохраняться в рамках всей таблицы, но не изменяться на полное название) |
| Локализация инфекции | Нижние дыхательные пути | Единое оформление по всему столбцу Сокращенный или полный вариант |
| Клинический материал | Бронхоальвеолярный лаваж | Единое оформление по всему столбцу Сокращенный или полный вариант |
| Отделение | Реанимация | Единое оформление по всему столбцу Сокращенный или полный вариант Аббревиатуры должны иметь поясняющие сноски |
| Возраст пациента | 62 | Единое оформление по всему столбцу Рекомендуется использовать количество полных лет, либо единые единицы измерения в рамках всей таблицы (годы, месяцы, дни) |
| Маркеры (резистентности/вирулентности) | CTX-M-15 (указать название маркера, см. столбец «Особенности») Положительный Не обнаружено Не определено Пустая ячейка | Единое оформление по всему столбцу Каждая группа маркеров – отдельный столбец (например, карбапенемазы) Если определение маркера неприменимо, т.е. лишено биологического смысла (например, определение ESBL для стафилококков), ячейка должна остаться пустой. Во всех остальных случаях факт отсутствия маркера необходимо фиксировать (например: не определялся, не обнаружено). Примеры наименований различных маркеров резистентности и способы их записи представлены в разделе 3.6. Методы выявления механизмов резистентности, имеющих клиническое и эпидемиологическое значение. Организация записей. |
| Симптом заболевания | Да | Единое оформление по всему столбцу Особые маркеры заболевания, например симптомы, требующие регистрации среди группы пациентов/ отделений (наличие лихорадки, маркеров воспаления и т.д.) |
4.3 Сопроводительная эпидемиологическая, клиническая и демографическая информация
Разработка полноценных локальных систем мониторинга АР невозможна без включения дополнительных данных – метаданных – в шаблон данных (таблицу) [64] . Подробная информация представлена в разделе 4.2. Шаблон для сбора и ввода данных. Анализ связей между метаданными и результатами определения чувствительности позволяет улучшить спектр превентивных мероприятий, подбор АМП в условиях конкретного стационара и/или отделения. Для того, чтобы метаданные были впоследствии проанализированы их следует заранее внести в стандартизованном виде в форму направления на МИ (см. Раздел 6.1. Шаблон направления на микробиологическое исследование) с последующим переносом в шаблон для сбора и ввода данных (см. Раздел 4.2. Шаблон для сбора и ввода данных).
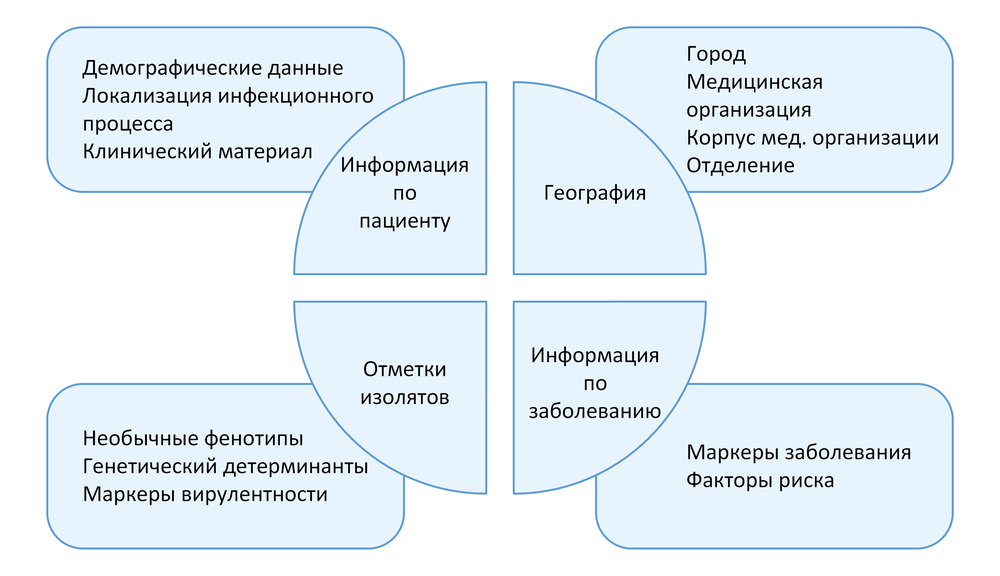
Информация по пациенту включает основные данные, которые могут быть важными для работы локальных систем мониторинга. При этом данные должны подчиняться требованиям универсального варианта оформления, указанным в разделе 4.4. Правила оформления и хранения данных. Присвоение идентификационного номера для каждого пациента может быть полезной практикой для поиска дублей и анализа повторных изолятов (тактика представлена ниже, в пункте Повторные изоляты и дубли). Следует отметить, что такую характеристику, как возраст, необходимо записывать не только как рассчитанное значение, но вносить и подтверждающие данные, т.е. дату рождения.
География может быть представлена в расширенном варианте, если мониторинг охватывает различные медицинские организации (несколько столбцов: регион, город и\или адрес организации) или минимальный вариант – отделения в рамках одной организации (один столбец). В последнем случае отделение выступает в качестве «условной локальной географической единицы». Независимо от объема географических данных важно придерживаться единого оформления ячейки (буквы в одном регистре, одинаковый вариант сокращения названий).
Информация по заболеванию позволяет внести те особенности, которые присущи конкретному заболеванию. Каждый такой показатель (фактор риска, маркер заболевания и т.п.) должен быть представлен в отдельном столбце таблицы, а при загрузке данной таблицы в AMRcloud необходимо выбрать интересующие для дальнейшего анализа показатели (столбцы).
Отметки изолятов (необычные фенотипы, генетические детерминанты, маркеры вирулентности) могут быть различными и не имеют ограничений по количеству столбцов как в исходной таблице пользователя, так и при загрузке в AMRcloud. Оптимальным вариантом является оформление по типу «один маркер – один столбец». Примеры оформления таблиц представлены в разделе 4.2. Шаблон для сбора и ввода данных.
Повторные изоляты и дубли
В реальной клинической практике, особенно у пациентов, долго находящихся на стационарном лечении, зачастую проводят повторные МИ. В результате серии проведенных исследований у конкретного пациента и получении возбудителей одного и того же вида из одного и того же локуса инфекции возникает особенная ситуация – появление повторных изолятов.
Повторные изоляты
это изоляты, выделенные из одного локуса инфекции у одного пациента и принадлежащие к одному виду. Идентификация повторных изолятов важна, т.к. включение всех повторных изолятов в итоговую таблицу для анализа данных приводит к завышению рассчитанной доли резистентных изолятов.От повторных изолятов следует отличать дубли.
Дубли
это записи с единым идентификационным номером изолята, являющиеся копией уже существующей записи (такая ситуация возможна при случайном повторном внесении в таблицу/ЛИС информации по одному и тому же изоляту).Поскольку единого подхода к устранению повторных изолятов не разработано, а бессистемное исключение может маскировать тенденции роста АР у возбудителя инфекции конкретного пациента, к каждому случаю следует подходить индивидуально. Единая таблица (начальный вариант до проведения статистического анализа) с записями по всем выявленным видам микроорганизмов и результатами определения чувствительности может содержать повторные изоляты. При получении изолятов одной видовой принадлежности, полученных из одного локуса инфекции у одного пациента, для исключения повторных изолятов рассматривается возможность применения двух подходов: использование критерия времени и результатов определения чувствительности к АМП.
- Критерий времени заключается в выборе и включении в дальнейший анализ только первого изолята, выделенного от пациента.
- Результаты определения чувствительности к АМП – выбор одной записи с интересующим фенотипом (например, включение в итоговый анализ записи по изоляту, для которой характерен максимальный уровень резистентности по интересующим препаратам).
Оптимальным вариантом, который может быть рекомендован при оценке суммарных статистик по локальной эпидемиологии АР при выделении микроорганизма одного вида, полученного при проведении повторных анализов у пациентов (один локус инфекции), является включение в итоговый анализ первого полученного изолята.
Таким образом, не рекомендуется включать в итоговый набор данных для оценки характеристик по локальной эпидемиологии АР более одного изолята от конкретного пациента. При необходимости учитывать изменения чувствительности к АМП микроорганизмов одного вида, полученных из одного локуса инфекции у одного пациента, следует оформить отдельную таблицу для анализа с обязательным использованием уникального номера пациента.
4.4 Правила оформления и хранения данных
Достижение преемственности в работе с данными, возможность проведения анализа любым заинтересованным специалистом и своевременность внесения исправлений – все это основывается на разработке и соблюдении правил оформления и хранения данных. Ключевые подходы к оформлению данных напрямую связаны с наиболее частыми ошибками, которые возникают в зависимости от типа данных: количественных, качественных (текстовых) и дат. При этом в большинстве случаев ошибки обусловлены отсутствием единообразия записи данных [65] .
4.4.1 Основные требования к таблице
- Таблица плоская (ячейки не объединяются между собой, нет дополнительных подстрок).
- У таблицы должна быть заголовочная строка (строка с названиями столбцов).
- Каждый изолят следует записывать на отдельной строке.
- Формат сохраненной таблицы – .xlsx или .csv. Объединенные требования к оформлению таблицы и записям в ячейках представлены в разделе 4.2. Шаблон для сбора и ввода данных.
4.4.2 Основные требования к записям в ячейке
Согласно концепции AMRcloud данные разделяются на обязательные и дополнительные.
4.4.2.1 Обязательные данные
- Идентификатор изолята (ID_изолята),
- Название вида микроорганизма,
- Название группы микроорганизмов,
- Дата,
- Результат определения чувствительности к антимикробному препарату (АМП).
Рекомендации по оформлению идентификационного номера изолята
В качестве идентификатора изолята необходимо использовать уникальные номера с целью однозначного определения принадлежности изолята. Идентификатор может быть буквенными или числовыми. Следует исключать специальные символы (?,!%$#<>).
Пример
- 2CD
- 69169
- 34ab
Рекомендации по оформлению названий вида микроорганизма
Полный вариант написания вида микроорганизма позволяет автоматически применить критерии интерпретации. Сокращения в названии вида недопустимы.
Пример
- Pseudomonas aeruginosa
- Enterococcus faecium
- Acinetobacter baumannii
Рекомендации по оформлению названия группы микроорганизмов
Оформление этого параметра рекомендуется выполнять с использованием англоязычного варианта. Название группы микроорганизмов может быть сокращено, но сокращения необходимо сделать стандартными (одинаковыми по всему столбцу).
Пример
- Staphylococcus
- Acinetobacter
- Enterobacterales
Рекомендации по оформлению дат
Обязательным «столбцом-датой» является дата взятия материала. Однако представленные правила рекомендуется использовать для всех дат, вносимых в таблицу (дата взятия материала, дата поступления пациента в стационар, дата рождения и т.д.). Ключевым условием является универсальность написания во всех строчках: т.е. если выбран вариант ДД.ММ.ГГГГ, необходимо использовать только его.
Пример: 12.02.2015; 08.01.2015; 29.12.2014
Пример
- 12.02.2015
- 08.01.2015
- 29.12.2014
Рекомендации по оформлению наименований антимикробных препаратов и результатов определения чувствительности:
- результат определения чувствительности к каждому АМП – отдельный столбец;
- алфавит английского языка;
- исключение кириллицы;
- исключение специальных символов (?,!%$#);
- генерическое название АМП (полный вариант написания). Список наименований представлен в разделе 6.2. Работа с шаблоном, справочниками и словарями;
- указание в наименовании АМП методов определения чувствительности с помощью идентифицирующих суффиксов.
Пример
- azithromycin_mic
- ceftazidime-avibactam_mic
- piperacillintazobactam_dd
- meropenem_sir
Таблица 4.4.1. Идентифицирующие суффиксы для обозначения методов определения чувствительности
| Метод тестирования | Суффикс |
|---|---|
| Значения минимальных подавляющих концентраций (МПК) - мг/л | _mic |
| Диаметры зон подавления роста - мм | _dd |
| Категории чувствительности (S/I/R) - значения заранее определяются пользователем | _sir |
4.4.2.2 Количественные (МПК и ДД) и категориальные (S/I/R) данные оценки чувствительности. Важность количественных данных
Данные минимальных подавляющих концентраций
Результаты, которые получены с применением автоматизированных систем, ручных панелей разведений или градиентной диффузии (полоски). Для AMRcloud это столбцы с результатами определения чувствительности вида: antibiotic_mic.
Особенностями столбцов с МПК являются:
- измерения в мг/л;
- для комбинированных препаратов – внесение значений МПК только для первого препарата в комбинации;
- преобразование в AMRcloud значений со знаками: <=, >=, >
Варианты изменений представлены в таблице 4.4.2.
Таблица 4.4.2. Возможные преобразования значений МПК в AMRcloud
| Исходное значение | Результат преобразования в AMRcloud | Пример |
|---|---|---|
| <= число | число | Исходная таблица <=2 В AMRcloud 2 |
| >= число | число | Исходная таблица >=16 В AMRcloud 16 |
| > число | число х2 | Исходная таблица >16 В AMRcloud 32 |
Данные диаметров зон подавления роста
Результаты, которые получены с применением диско-диффузионного метода. Для AMRcloud это столбцы с результатами определения чувствительности вида: antibiotic_dd.
Особенностью столбцов с диаметров зон подавления роста является:
- диапазон целых числовых значений от 6 до 50 мм.
Данные категорий чувствительности
Результаты, которые получены после применения критериев интерпретации с последующим внесением в таблицу (т.е. неколичественные значения). Для AMRcloud это столбцы с результатами определения чувствительности вида: antibiotic_sir.
Особенностями столбцов с категориями чувствительности является:
- указание одного типа чувствительности: S или I или R;
- невозможность использования при изменении критериев интерпретации.
Таким образом, количественные данные категоризируются (определение категории S/I/R) согласно пороговым значениям, указанным в правилах интерпретации. Ежегодно интерпретационные правила пересматриваются и дополняются. Если результаты представлены в виде уже «готовых» категорий (S/I/R), невозможно адекватно сопоставить исторические данные (например, данные прошлого года) с текущими. Поэтому для организации мониторинга настоятельно рекомендуется использовать именно количественные данные, так как к ним всегда можно применить актуальные на данный момент интерпретационные критерии (в том числе к историческим данным). Это обеспечит преемственность и сопоставимость результатов.
4.4.2.3 Дополнительные данные
Любое инфекционное заболевание характеризуется набором отличительных характеристик, которые могут занимать важное место в итоговых эпидемиологических данных. Эти свойства относятся к метаданным (дополнительной информации о содержимом, т.е. изоляте). Включение метаданных позволяет оценить их важность в эпидемиологическом процессе, а также помогает выявлению «скрытых» причинно-следственных связей. Основные группы метаданных представлены в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
Ключевыми требованиями к записи в ячейке являются: исключение специальных символов (?,!%$#<>) и использование единых обозначений. В исходной таблице общее количество столбцов с дополнительными данными может быть любым. При загрузке в AMRcloud установлены следующие требования:
- Отметки изолятов – количество столбцов неограниченно.
- География, информация по заболеванию, информация по пациенту – до 12 показателей + 1 количественный параметр (например, возраст или вес пациента, или длительность госпитализации и т.д.).
Рекомендации по оформлению данных пациента
В группу таких данных могут быть включены различные показатели, например:
- Возраст пациента (количественный параметр);
- Пол пациента (универсальное оформление по всему столбцу);
- Локализация инфекции (универсальное оформление по всему столбцу, сокращенный или полный вариант написания);
- Клинический материал (универсальное оформление по всему столбцу, сокращенный или полный вариант написания) [66] .
В исходной таблице количество таких столбцов с пациент-ориентированной информацией неограниченно. При загрузке в AMRcloud количественный параметр может быть только один. Варианты записи показателей представлены в разделе 4.2. Шаблон для сбора и ввода данных и в разделе 6.2. Работа с шаблоном, справочниками и словарями.
Рекомендации по оформлению географических данных
Группа показателей «География» может включать данные конкретного географического объекта – медицинской организации (название, корпус и/или отделение), а также город [67] . Геокодирование, т.е. определение географических координат, в AMRcloud происходит путем автоматического поиска совпадений «географический объект – широта | долгота». Необходимость определения координат вручную понадобится в нескольких ситуациях:
- Таблица с географическими объектами (название, корпус и/или отделение) загружаются в AMRcloud впервые и/или координаты не сохранялись для повторного использования. Подробнее о сохранении координат и их просмотре в 5.4. Актуализация информации.
- Координаты автоматически проставлены, но выявлены опечатки/неточности.
Ключевым требованием является универсальность написания: если использовалась аббревиатура/сокращение названия географического объекта, то их не следует менять на полное название.
Примеры написания географических объектов представлены в разделе 4.2. Шаблон для сбора и ввода данных.
Рекомендации по оформлению данных «по заболеванию»
Основным требованием является единое оформление всех показателей. Особыми маркерами заболевания могут быть симптомы, требующие регистрации среди группы пациентов (наличие лихорадки, маркеров воспаления, результатов оценки тяжести состояния пациента по соответствующим шкалам – qSOFA, NEWS, APACHE II и т.д.).
Рекомендации по оформлению данных «отметки изолятов»
Для данной группы показателей характерны следующие особенности:
- Общее количество маркеров не ограничено;
- Возможно создание пользователем собственной «группировки маркеров»;
- Указываются генетические маркеры, особые фенотипы, факторы вирулентности.
При работе с такими показателями необходимо придерживаться правил, которые могут быть отражены в справочнике к локальной системе мониторинга. Возможный алгоритм заполнения предложен на рисунке 1.
Вариант заполнения данной группы показателей представлен в разделах 3.7. Методы выявления механизмов резистентности, имеющих клиническое и эпидемиологическое значение и 4.2. Шаблон для сбора и ввода данных.

5. Использование платформы AMRcloud в мониторинге антибиотикорезистентности
AMRcloud – это онлайн-платформа анализа и обмена пользовательскими данными по резистентности к антимикробным препаратам, которая содержит набор инструментов для визуализации результатов определения чувствительности микроорганизмов к антимикробным препаратам и распространенности детерминант устойчивости к антибиотикам [12] .
5.1 Создание проектов и наборов данных
В рамках работы с онлайн-платформой AMRcloud пользователь может создавать различные проекты.
Проект
это структурный элемент приложения, который позволяет группировать несколько наборов с данными (полученных в результате загрузки таблиц с результатами мониторинга в AMRcloud).Если провести аналогию с документами на компьютере, то наборы данных – это файлы, а проект – папка, в которой находятся файлы.
5.1.1 Создание проекта
Для создания проекта пользователю следует нажать кнопку + Новый проект в верхнем левом углу экрана.
Во всплывающем окне необходимо заполнить поля Название нового проекта и Подзаголовок проекта (для каждого поля установлено ограничение в 255 символов). При постоянной работе с различными проектами рекомендуется использовать полностью уникальные или значимо отличающиеся между собой названия. Не стоит ограничиваться вариантами test1 и test2. Подзаголовок проекта может быть представлен ключевыми словами, которые поясняют название, например, Отчет_сентябрь_2020.
5.1.2 Типы проектов
Пользователь может работать с двумя типами проектов:
- Мои проекты
- Командные проекты
Мои проекты – персональные проекты пользователя. Доступ к данным в таком проекте единолично предоставлен создавшему его пользователю. Если пользователь захочет поделиться данными своего проекта с другими людьми, он сможет сделать это с помощью создания общедоступной ссылки.
Командные проекты – проекты, в которых данные доступны всем пользователям, объединенным в одну команду. Подробная информация представлена далее в разделах 5.1.2.2. Команда, создание и назначение на проект и 5.5.1. Участники команды.
5.1.2.1 Инструменты работы с вкладкой «Мои проекты»
В рамках работы с персональными проектами исследователю доступны несколько функциональных возможностей, и для их выбора необходимо воспользоваться панелью инструментов.

5.1.2.2 Команда, создание и назначение на проект
Команда
это несколько пользователей, объединенных для работы над одним проектом.Создатель команды является Администратором, а остальные пользователи, приглашенные администратором команды – Участники команды. Во вкладке «Команды» пользователь может создавать команды, а также просматривать список команд, в которых он включен в качестве приглашенного участника, т.е. в которых он не является администратором.
Рекомендуемые этапы по созданию командных проектов:
- Создать проект.
- Перейти в раздел Команды.
- Создать команду, нажав кнопку + Добавить команду.
- Пригласить участников в команду: нажать кнопку Добавить участника, ввести адрес электронной почты участника (данный участник должен быть зарегистрирован в AMRcloud заранее).
- Перейти в раздел Проекты.
- Выбрать необходимый проект, кликнув по нему мышкой.
- На панеле инструментов нажать кнопку Назначить команду.
- Из предложенного списка выбрать подходящую.
5.1.3 Создание и редактирование набора данных
Набор данных
структурная единица в рамках проекта, основу составляет пользовательская таблица с результатами мониторинга, которую загружают в AMRcloud. Набор данных содержит информацию для непосредственной работы платформы.В рамках проекта пользователь может создавать несколько наборов данных. Для этого следует перейти в выбранный проект, нажав кнопку + Открыть слева от названия проекта, а затем нажать кнопку + Новый набор данных. Во всплывающем окне необходимо заполнить поля Название нового набора и Подзаголовок нового набора (для каждого поля установлено ограничение в 255 символов).
При постоянной работе с несколькими наборами данных в рамках одного проекта рекомендуется использовать понятные, полностью уникальные или значимо отличающиеся между собой названия. Например, не ограничиваться вариантами test1, test2.
После создания набора данных система автоматически переведет пользователя на страницу импорта, где также можно ознакомиться с ключевыми требованиями к загружаемой таблице, списком АМП. Также на страницу импорта можно попасть, выбрав левой кнопкой ранее созданный набор данных и нажав кнопку Редактировать набор данных в панели инструментов для работы с наборами данных.

5.2 Алгоритмы базового анализа данных
Основная информация, которая может быть получена с помощью систем локального мониторинга и использована при составлении протокола АМТ: этиологическая структура возбудителей для различных групп пациентов и спектр активности антибиотиков в отношении ключевых возбудителей. Для получения данной информации в AMRcloud предусмотрен подход раздельного представления и анализа данных с помощью системы вкладок:
- Структура данных;
- Микроорганизмы;
- Антибиотики (все);
- Сравнения.
Работа с AMRcloud предусматривает фильтрацию набора данных. Фильтрация – это выборочное отображение данных по заданным критериям (выбор показателей в поле Параметры основные).
5.2.1 Вкладка «Структура данных»
Предназначена для анализа всего объема данных (собранных в процессе мониторинга АР и загруженных в AMRcloud) в зависимости от выбранных параметров фильтрации и настроек в подвкладках: «Круговая диаграмма», «Группировать по», «Карта».
5.2.1.1 Подвкладка «Круговая диаграмма»
Назначение:
- Оценка распределения долей в наборе данных по установленному параметру (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.). Подробная информация о метаданных представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
- Выявление опечаток в оформлении метаданных.
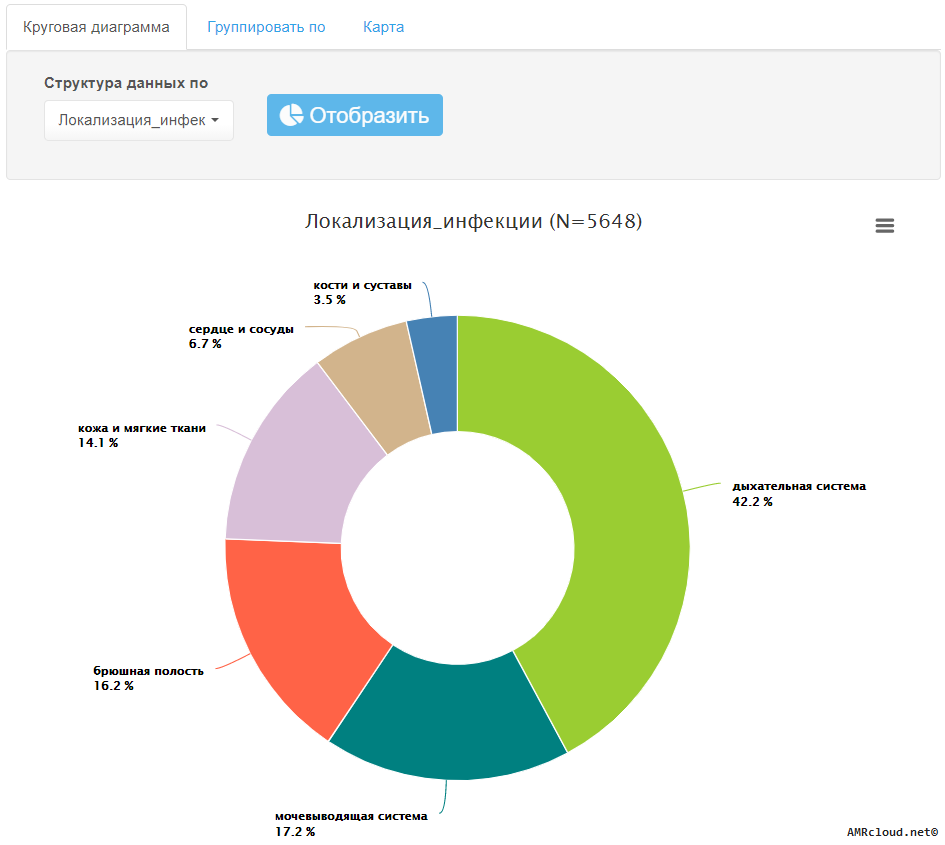
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Структура данных (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Переход в подвкладку Круговая диаграмма.
- Выбор параметра для оценки из выпадающего списка.
- Нажатие кнопки Отобразить.
5.2.1.2 Подвкладка «Группировать по»
Назначение:
- Оценка распределения долей в наборе данных по установленному параметру (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.) в зависимости от другого параметра (даты или метаданных). Подробная информация про метаданные представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
- Выявление опечаток в оформлении метаданных.
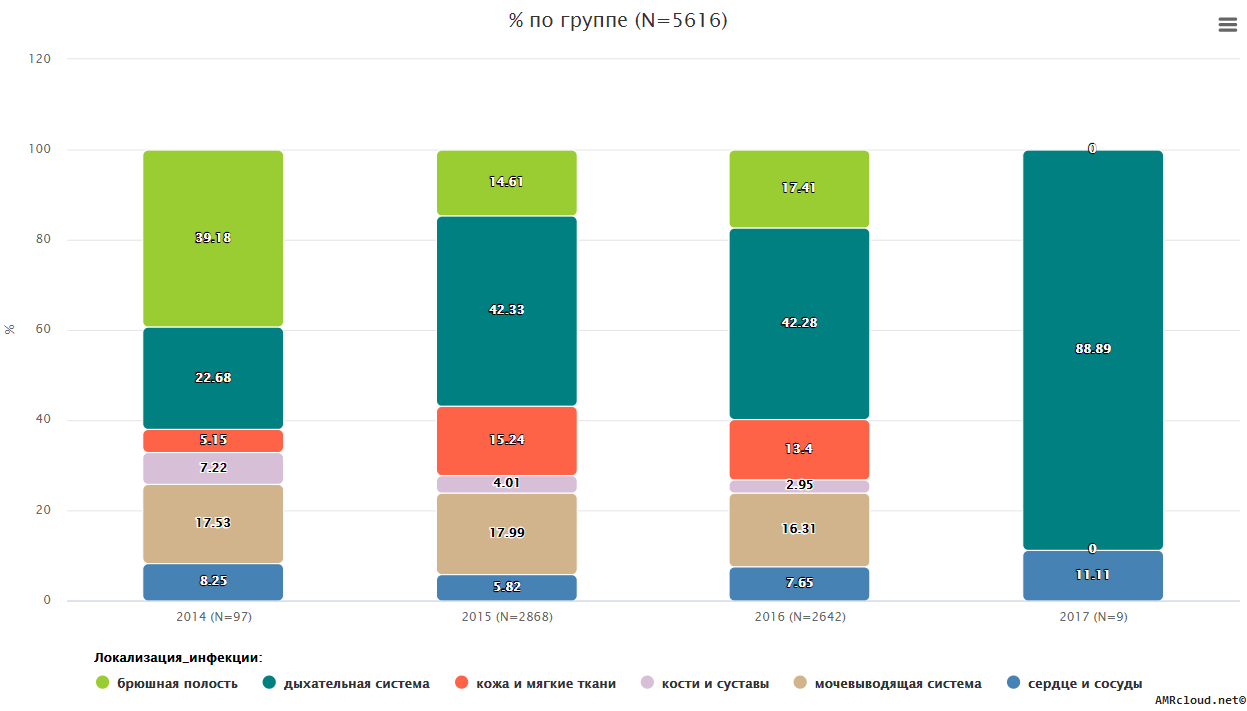
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Структура данных (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Переход в подвкладку Группировать по.
- Выбор параметра для оценки из выпадающего списка.
- Нажатие кнопки Отобразить.
5.2.1.3 Подвкладка «Карта»
Назначение:
- Оценка распределения долей в наборе данных по установленному параметру (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.) в зависимости от географического расположения. Подробная информация про метаданные представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
- Выявление опечаток в оформлении метаданных.
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Структура данных (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Переход в подвкладку Карта.
- Выбор параметра для оценки из выпадающего списка.
- Нажатие кнопки Отобразить.
5.2.2 Вкладка «Микроорганизмы»
Назначение:
- Представление этиологической структуры возбудителей (вид и группа микроорганизмов) в зависимости от выбранных параметров фильтрации.
Особенности:
- Гистограмма – отображение 10 топовых (самых частых) возбудителей.
- Таблица – последовательное представление всех возбудителей в зависимости от частоты встречаемости в выборке (анализируемый набор в зависимости от выбранных фильтров).
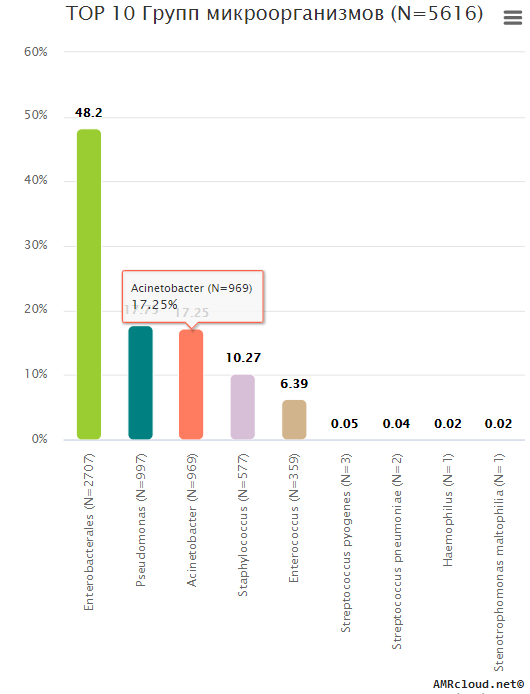
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Микроорганизмы (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Нажатие кнопки Отобразить.
5.2.3 Вкладка «Антибиотики (все)»
Назначение:
- Сводное представление категорий чувствительности микроорганизмов в пределах одной группы микроорганизмов, с учетом выбранных параметров фильтрации и АМП из выпадающего списка. Особенности:
- Выбор источника данных S/I/R:
- объединенные данные S/I/R (столбцы АМП_sir);
- автоинтерпретация из МПК (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_mic);
- автоинтерпретация из диаметров зон подавления роста (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_dd).
- Изменение варианта отображения: S/I/R, S/I+R, S+I/R.
- Представление в виде гистограммы или таблицы.
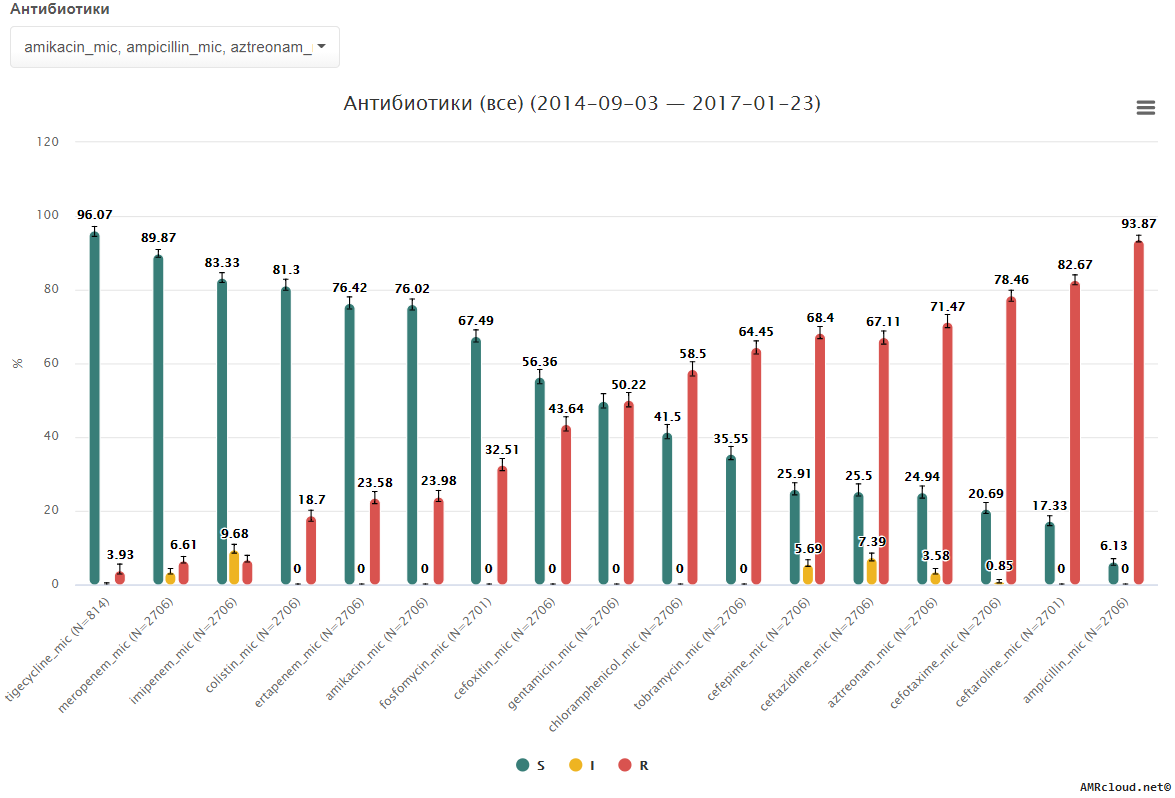
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные с обязательным выбором группы микроорганизмов. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Антибиотики (все) (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R.
- Выбор подходящего варианта отображения.
- Нажатие кнопки Отобразить.
5.2.4 Вкладка «Сравнения»
Предназначена для анализа всего объема данных (собранных в процессе мониторинга АР и загруженных в AMRcloud) в зависимости от выбранных параметров фильтрации в полях («Параметры основные» и «Параметры сравнения»), а также настроек в подвкладках: «Структура данных», «S/I/R», «Детерминанты». В рамках данной вкладки осуществляется сравнение различных параметров (метаданных, чувствительности к АМП, маркеров АР и т.д.) между условными группами «Параметры основные» и «Параметры сравнения», формируемыми на основании произвольной (представляющей интерес для специалиста) фильтрации данных.
5.2.4.1 Подвкладка «Структура данных»
Назначение:
- Сравнение распределения долей в наборе данных по выбранным параметрам фильтрации и установленному параметру (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.) между сформированными группами «Параметры основные» и «Параметры сравнения». Подробная информация про метаданные представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
Особенности:
- Возможность просмотра результатов в виде гистограммы и таблицы.
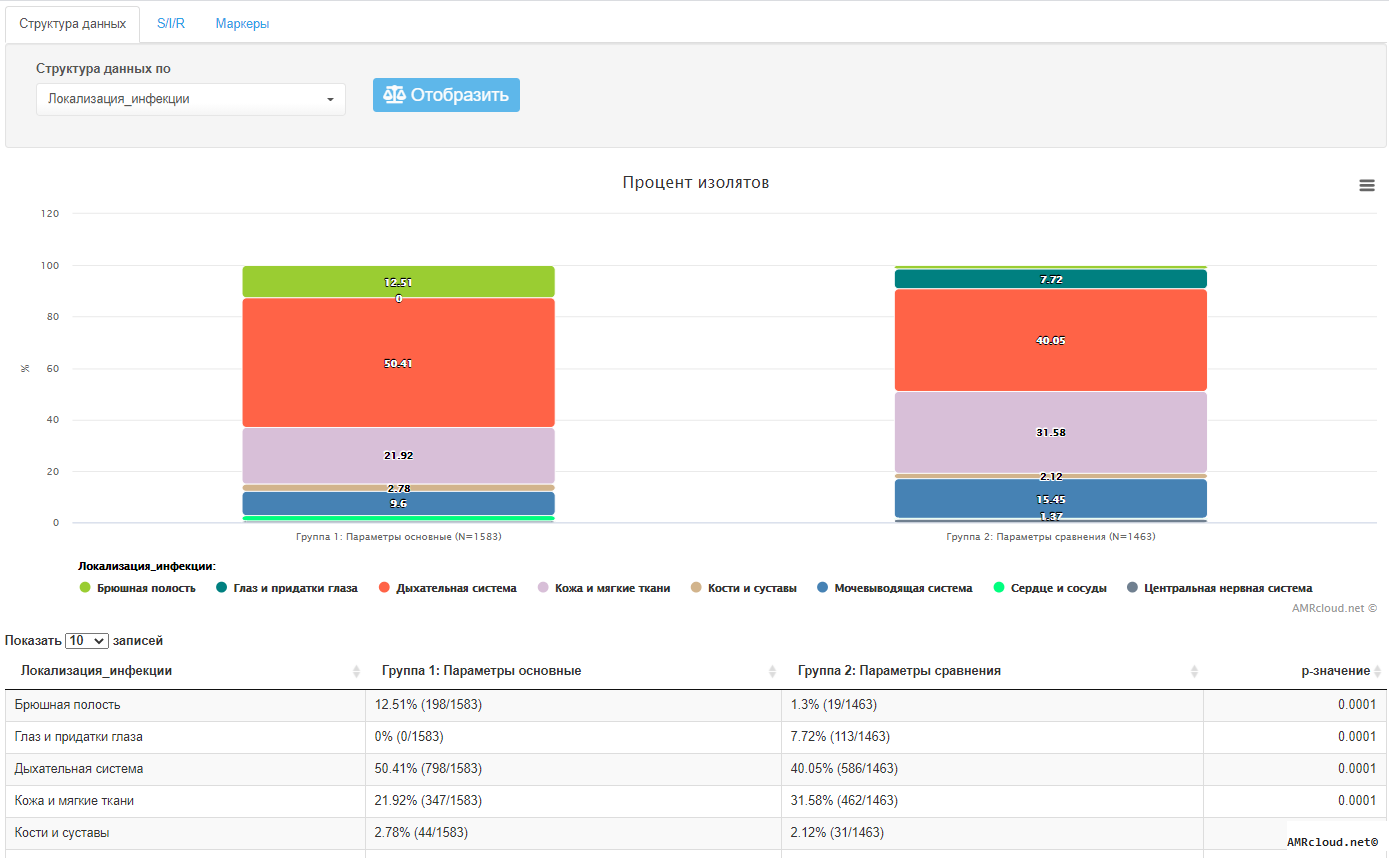
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные.
- Выбор необходимых значений в фильтрах поля Параметры сравнения.
- Переход в подвкладку Структура данных.
- Выбор параметра для оценки из выпадающего списка.
- Нажатие кнопки Отобразить.
5.2.4.2 Подвкладка «S/I/R»
Назначение:
- Сравнение распределения категорий чувствительности и значений МПК, диаметров зон подавления роста выбранной группы микроорганизмов по определенным параметрам (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.) между сформированными группами «Параметры основные» и «Параметры сравнения».
Особенности:
- Выбор источника данных S/I/R:
- объединенные данные S/I/R (столбцы АМП_sir);
- автоинтерпретация из МПК (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_mic);
- автоинтерпретация из диаметров зон подавления роста (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_dd).
- Возможность просмотра результатов в виде гистограммы и таблицы.
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные.
- Выбор необходимых значений в фильтрах поля Параметры сравнения.
- Переход в подвкладку S/I/R.
- Выбор Источника данных S/I/R.
- Выбор антимикробного препарата из выпадающего списка.
- Нажатие кнопки Отобразить.
5.2.4.3 Подвкладка «Детерминанты»
Назначение:
- Сравнение распределения долей маркеров (маркеры – столбцы метаданных, объединенные в группу «Отметки изолятов»: необычные фенотипы, генетические детерминанты, маркеры вирулентности) по определенным параметрам фильтрации (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.) между сформированными группами «Параметры основные» и «Параметры сравнения».
Особенности:
- Возможность просмотра результатов в виде гистограммы и таблицы.
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные.
- Выбор необходимых значений в фильтрах поля Параметры сравнения.
- Переход в подвкладку Детерминанты.
- Выбор группы маркеров и перечня маркеров из выпадающих списков.
- Нажатие кнопки Отобразить.
5.3 Углубленный анализ данных
Расширенный объем информации, который может быть получен с помощью систем локального мониторинга и использован при составлении протокола АМТ: оценка активности определенных АМП к выбранным видам микроорганизмов, активности АМП при устойчивости к другим АМП и определении значимости метаданных. Для получения данной информации в AMRcloud предусмотрен подход раздельного представления и анализа данных с помощью системы вкладок:
- Выбранный антибиотик;
- Ассоциированная устойчивость;
- Маркеры.
5.3.1 Вкладка «Выбранный антибиотик»
Предназначена для анализа данных (собранных в процессе мониторинга АР и загруженных в AMRcloud) в зависимости от нескольких параметров:
- Группа микроорганизмов.
- Источник данных S/I/R.
- Антимикробный препарат.
- Фильтры и настройки в подвкладках.
Вкладка «Выбранный антибиотик» включает подвкладки: «S/I/R графики», «Карта», «МПК», «Диаметры зон».

5.3.1.1 Подвкладка «S/I/R графики»
Назначение:
- Оценка распределения доли выбранной группы микроорганизмов с различной категорией чувствительности (S, I, R) к выбранному АМП в зависимости от установленного параметра (параметр – это дата или любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.). Подробная информация про метаданные представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
- Выявление опечаток в оформлении метаданных.
Особенности:
- Подраздел «Суммарная информация» включает круговую диаграмму и таблицу.
Раздел Суммарная информация - Подраздел «Группировать по» включает настройки: тип графика, сортировка, тип сортировки. А также модуль сравнения нечувствительных изолятов с отображением по типу граф или матрица.
Раздел Группировать по (настройка и пример гистограммы) 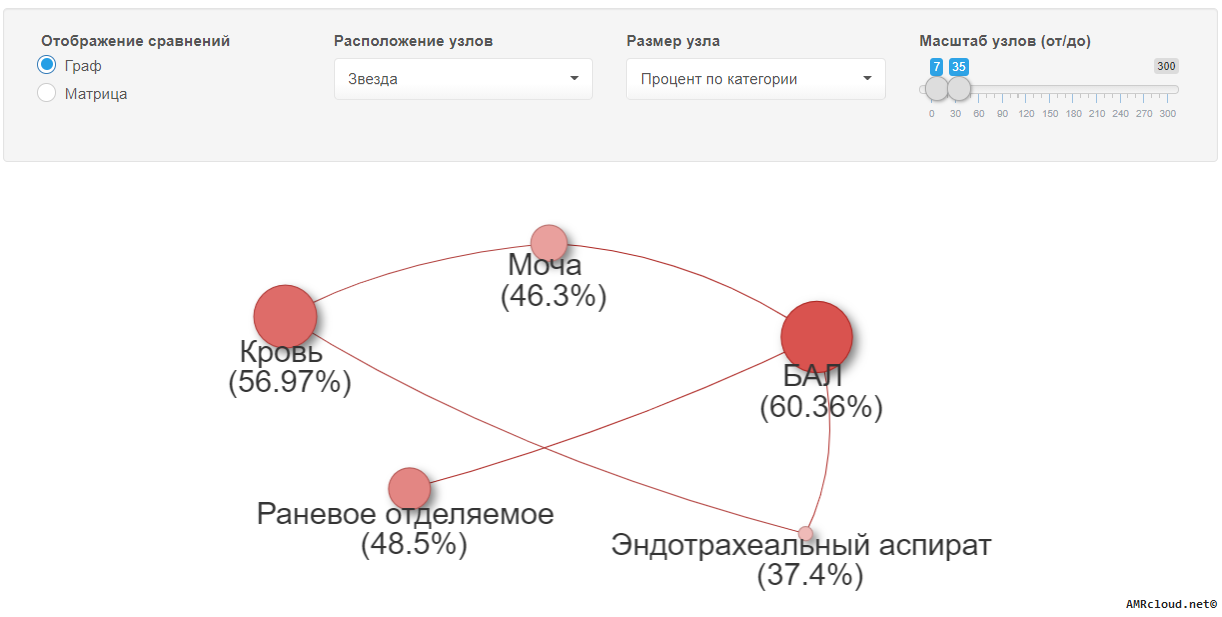
Раздел Группировать по. Отображение сравнений по типу - граф - Подраздел «Таблица» с поиском по столбцам.
Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Выбранный антибиотик (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R.
- Выбор антимикробного препарата (из выпадающего списка).
- Переход в подвкладку S/I/R графики.
- Выбор варианта отображения.
- Выбор способа группировки (параметра из выпадающего списка).
- Настройка ширины 95%ДИ (уменьшение ширины 95% ДИ позволяет выявлять статистически значимые различия, исключает малую выборку).
- Нажатие кнопки Отобразить.
- Переход в подраздел.
- Настройка подраздела.
95% Доверительный Интервал
это интервал, в пределах которого будет находиться истинное значение оцениваемого показателя в 95 случаях из 100, при повторном проведении серии из 100 исследований с таким же дизайном.Таблица 5.3.1. Настройки подраздела «Группировать по»
| Параметр | Варианты |
|---|---|
| Тип графика | Гистограмма + 95%ДИ Гистограмма Тренд Линия Площадь |
| Сортировка | S/S +I R/ I+R Наименование – параметр метаданных N – абсолютное количество |
| Тип сортировки | Нисходящая – от большего к меньшему / в обратном алфавитном порядке Восходящая – от меньшего к большему / в алфавитном порядке |
| Отображение сравнений | Граф – связи между узлами = статистически значимые отличия между долями Матрица – при устойчивости к АМП1 устойчивость к АМП2 будет с вероятностью = пересечение столбцов |
5.3.1.2 Подвкладка «Карта»
Назначение:
- Оценка распределения доли микроорганизмов с различной категорией чувствительности к выбранному АМП в зависимости от географического объекта.
- Выявление опечаток в оформлении метаданных – группа «География ».
Особенности:
- Подраздел «Карта» с настройкой размера точек и порога оповещения (процент устойчивых изолятов, начиная со значений которого отображается %R на карте).
Подвкладка Карта - Подраздел «Рейтинг» – последовательность географических объектов в зависимости от доли устойчивых изолятов, отсортированная от большего значения к меньшему.
Подвкладка Рейтинг - Подраздел «Регрессия» – зависимость доли устойчивых изолятов по различным географическим объектам от года выделения изолята.
Подвкладка Регрессия
Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Выбранный антибиотик (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R.
- Выбор антимикробного препарата (из выпадающего списка).
- Переход в подвкладку Карта.
- Выбор варианта отображения.
- Нажатие кнопки Отобразить.
- Переход в подраздел.
- Настройка подраздела.
5.3.1.3 Подвкладка «МПК»
Назначение:
- Анализ распределения значений МПК выбранного АМП (применяется к столбцам вида АМП_mic).
Особенности:
- Подраздел «МПК тренд» – распределение значений МПК антимикробного препарата в зависимости от параметра группировки (дата или любой столбец из метаданных, за исключением группы «Отметки изолятов») с получением информации по выбранной области значений Получение информации по выбранной области включает распределение МПК, таблицу с данными МПК + метаданные по каждому изоляту.
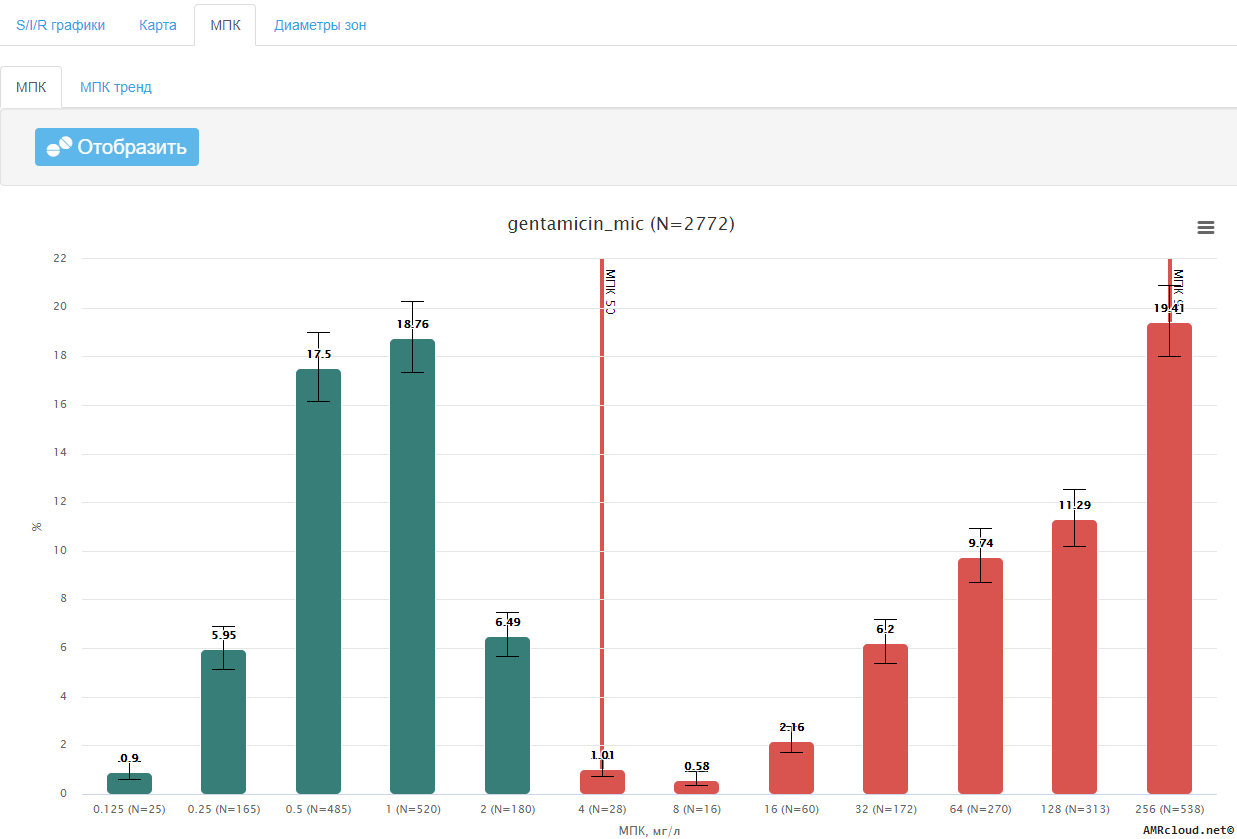
Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Выбранный антибиотик (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R – Автоинтерпретация из МПК.
- Выбор антимикробного препарата (из выпадающего списка)
- Переход в подвкладку МПК.
- Выбор параметра из выпадающего списка Группировать по.
- Нажатие кнопки Отобразить.
- Настройка вкладки (МПК%, размер точек).
- Выделение необходимой области (серии «точек») с помощью левой кнопки мыши.
- Нажатие кнопки Получить информацию.
Пункты 7–9 приведены для подраздела МПК тренд.
5.3.1.4 Подвкладка «Диаметры зон»
Назначение:
- Анализ распределения значений диаметров зон подавления роста выбранного АМП (применяется к столбцам вида АМП_dd).
Особенности:
- Подраздел «Диаметры зон (тренд)» – распределение значений диаметров зон подавления роста АМП в зависимости от параметра группировки (дата или любой столбец из метаданных, за исключением группы «Отметки изолятов») с получением информации по выбранной области значений.
Получение информации по выбранной области включает распределение значений диаметров зон подавления роста, таблицу с данными диаметров зон подавления роста + метаданные по каждому изоляту.
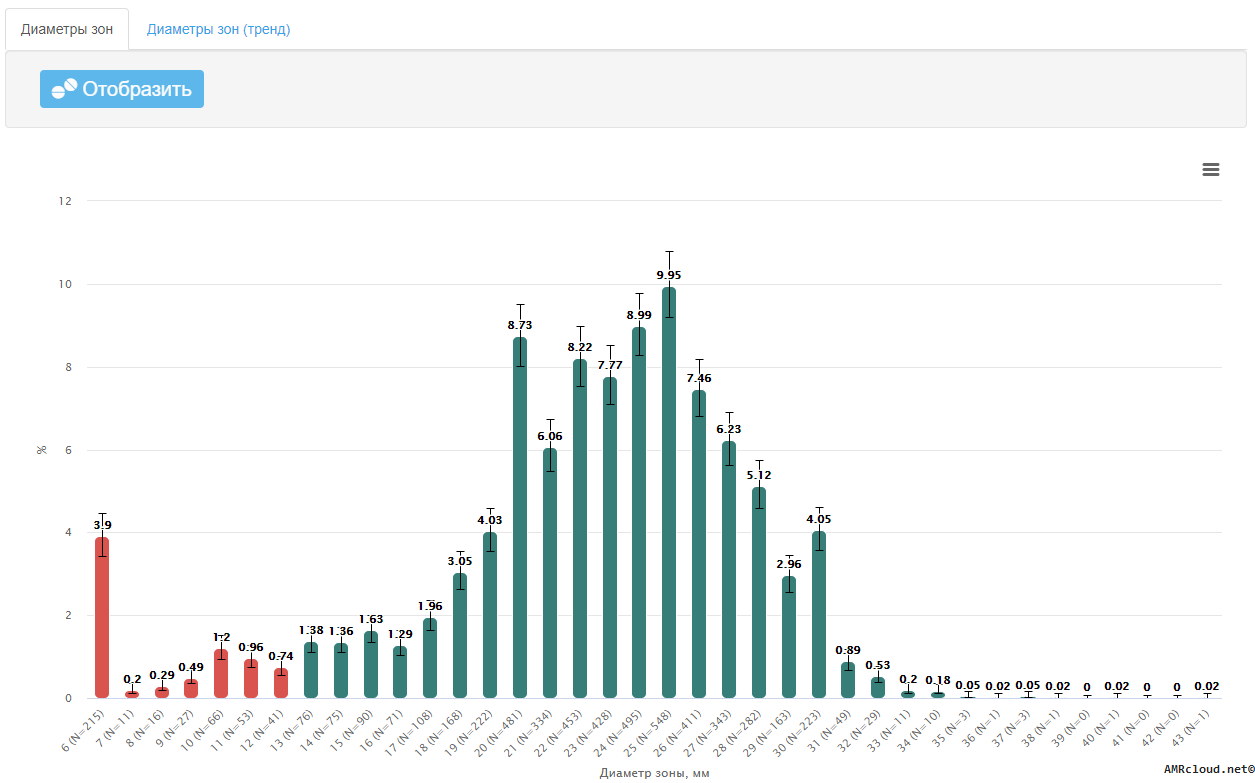
Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Выбранный антибиотик (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R – Автоинтерпретация из диаметров зон.
- Выбор антимикробного препарата (из выпадающего списка).
- Переход в подвкладку Диаметры зон.
- Выбор параметра из выпадающего списка Группировать по.
- Нажатие кнопки Отобразить.
- Настройка вкладки (размер точек).
- Выделение необходимой области (серии «точек») с помощью левой кнопки мыши.
- Нажатие кнопки Получить информацию.
Пункты 7–9 приведены для подраздела Диаметры зон (тренд).
5.3.2 Вкладка «Ассоциированная устойчивость»
Предназначена для анализа у выбранной группы микроорганизмов взаимосвязей:
- Вероятность наличия устойчивости к АМП2 при условии наличия устойчивости к АМП1 (Устойчивость к АМП1 -> Устойчивость к АМП2).
- Устойчивость к нескольким АМП одновременно (Устойчивость к АМП1+Устойчивость к АМП2+Устойчивость к АМП..n = %).
- Выявление значений МПК2 при определенном значении МПК1 (Значение МПК1 -> Значение МПК2).
Вкладка «Ассоциированная устойчивость» включает подвкладки: «Матрица», «Множественная устойчивость», «Диаграмма рассеяния».

5.3.2.1 Подвкладка «Матрица»
Назначение:
- Оценка вероятности появления устойчивости к АМП1 при наличии устойчивости к АМП2.
Особенности:
- Выбор Источника данных S/I/R:
- объединенные данные S/I/R (столбцы АМП_sir);
- автоинтерпретация из МПК (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_mic);
- автоинтерпретация из диаметров зон подавления роста (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_dd).
- Подразделы: матрица, таблица.

Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Ассоциированная устойчивость (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R.
- Нажатие кнопки Отобразить.
- Переход в подраздел.
- Настройка ширины 95% ДИ (уменьшение ширины 95% ДИ позволяет выявлять статистически значимые различия, исключает малую выборку).
- Выбор комбинации АМП.
- Оценка вероятности устойчивости к АМП2, при устойчивости к АМП1 ( АМП1 – вертикальный столбец, АМП2 – горизонтальный столбец).
- Поиск записей с использованием фильтра.
Пункты 6–7 приведены для подраздела Матрица.
Пункт 8 приведен для подраздела Таблица.
5.3.2.2 Подвкладка «Множественная устойчивость»
Назначение:
- Определение процента устойчивых изолятов выбранной группы, исходя из комбинации АМП.
Особенности:
- Выбор Источника данных S/I/R:
- объединенные данные S/I/R (столбцы АМП_sir);
- автоинтерпретация из МПК (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_mic);
- автоинтерпретация из диаметров зон подавления роста (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_dd).
- Различные комбинации АМП.
- Выбор количества АМП, к которым наблюдается устойчивость.

Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Ассоциированная устойчивость (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор Источника данных S/I/R.
- Выбор антимикробных препаратов из выпадающего списка.
- Выбор минимального количества АМП, для которых наблюдается устойчивость (R как минимум к N антибиотикам).
- Нажатие кнопки Отобразить.
5.3.2.3 Подвкладка «Диаграмма рассеяния»
Назначение:
- Соотношение распределений значений МПК, диаметров зон подавления роста и SIR для двух различных АМП. Особенности: - Выбор различных комбинаций АМП.
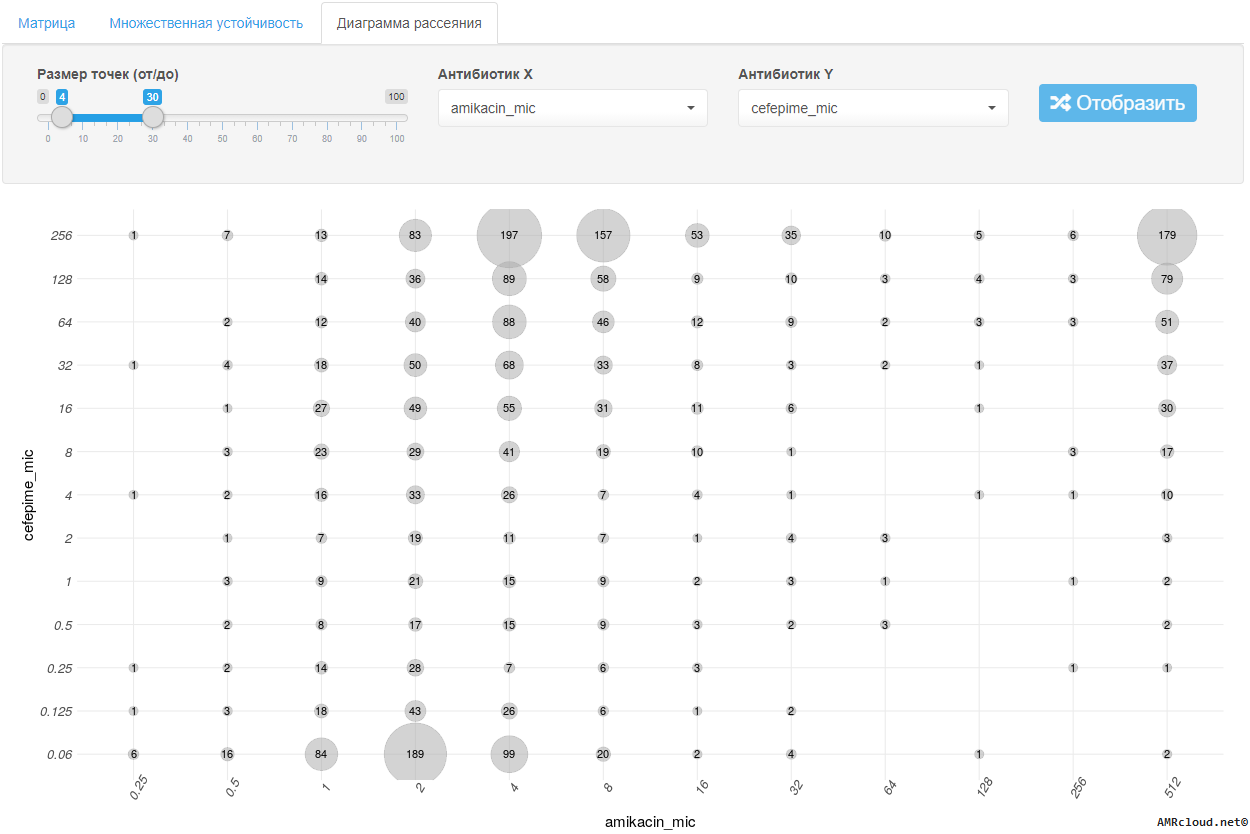
Алгоритм работы:
- Выбор одной группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется выбрать группу микроорганизмов и сразу переходить во вкладку Ассоциированная устойчивость (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор антимикробных препаратов из выпадающих списков.
- Нажатие кнопки Отобразить.
- Настройка размера точек.
5.3.3 Вкладка «Маркеры»
Предназначена для анализа необычных фенотипов, генетических детерминант, маркеров вирулентности в данных (собранных в процессе мониторинга АР и загруженных в AMRcloud).
Вкладка «Маркеры» включает подвкладки: «Маркеры», «Группировать по», «Карта», «Карта х2», «Антибиотики (все)», «МПК», «Диаметры зон», «Время первого обнаружения».
5.3.3.1 Подвкладка «Маркеры»
Назначение:
- Оценка распределения долей маркеров (маркеры – столбцы метаданных, объединенные в группу «Отметки изолятов»: необычные фенотипы, генетические детерминанты, маркеры вирулентности).
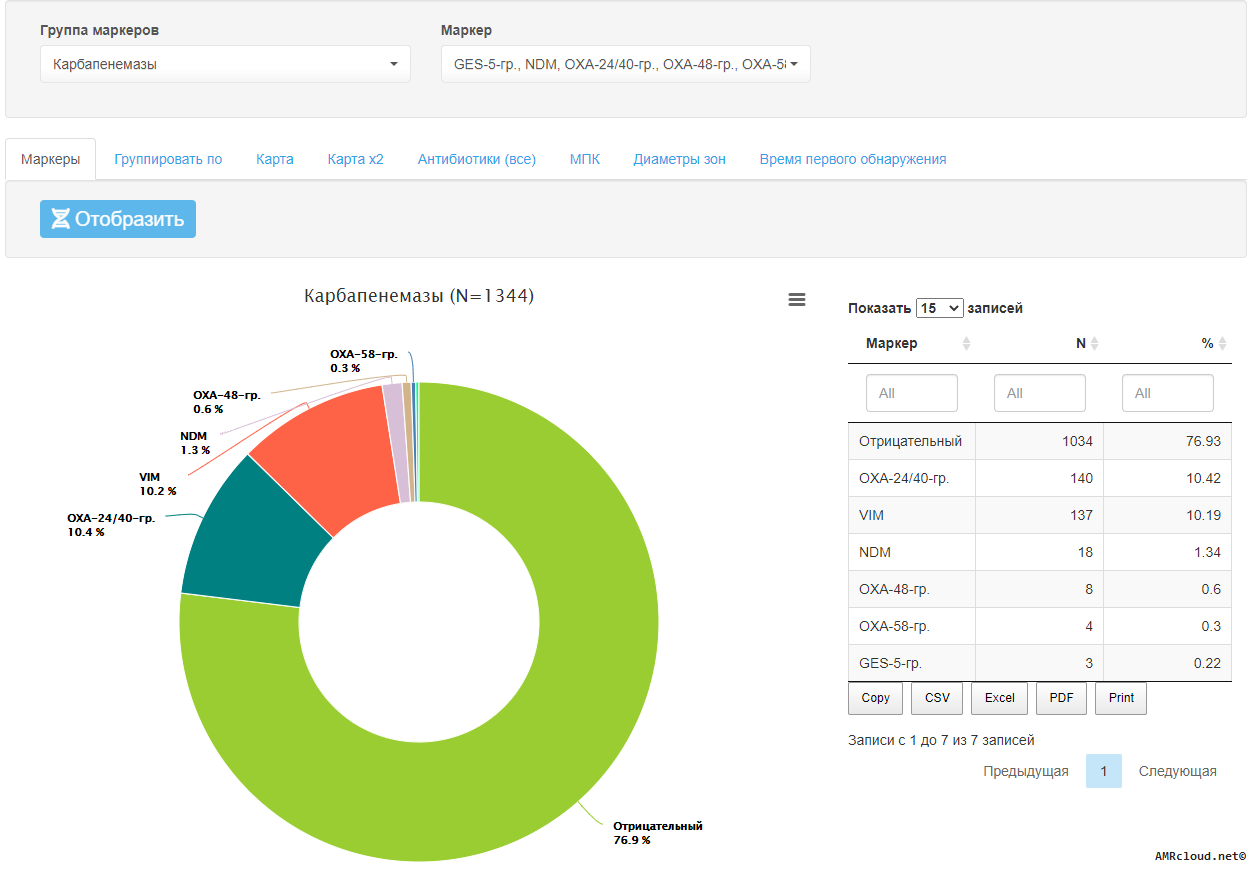
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку Маркеры.
- Нажатие кнопки Отобразить.
5.3.3.2 Подвкладка «Группировать по»
Назначение:
- Оценка распределения долей выбранных маркеров по установленному параметру (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.). Подробная информация про метаданные представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
Особенности:
- Индивидуальная настройка подвкладки: тип графика, сортировка, тип сортировки, вариант расчета.
- Подразделы: график, таблица.
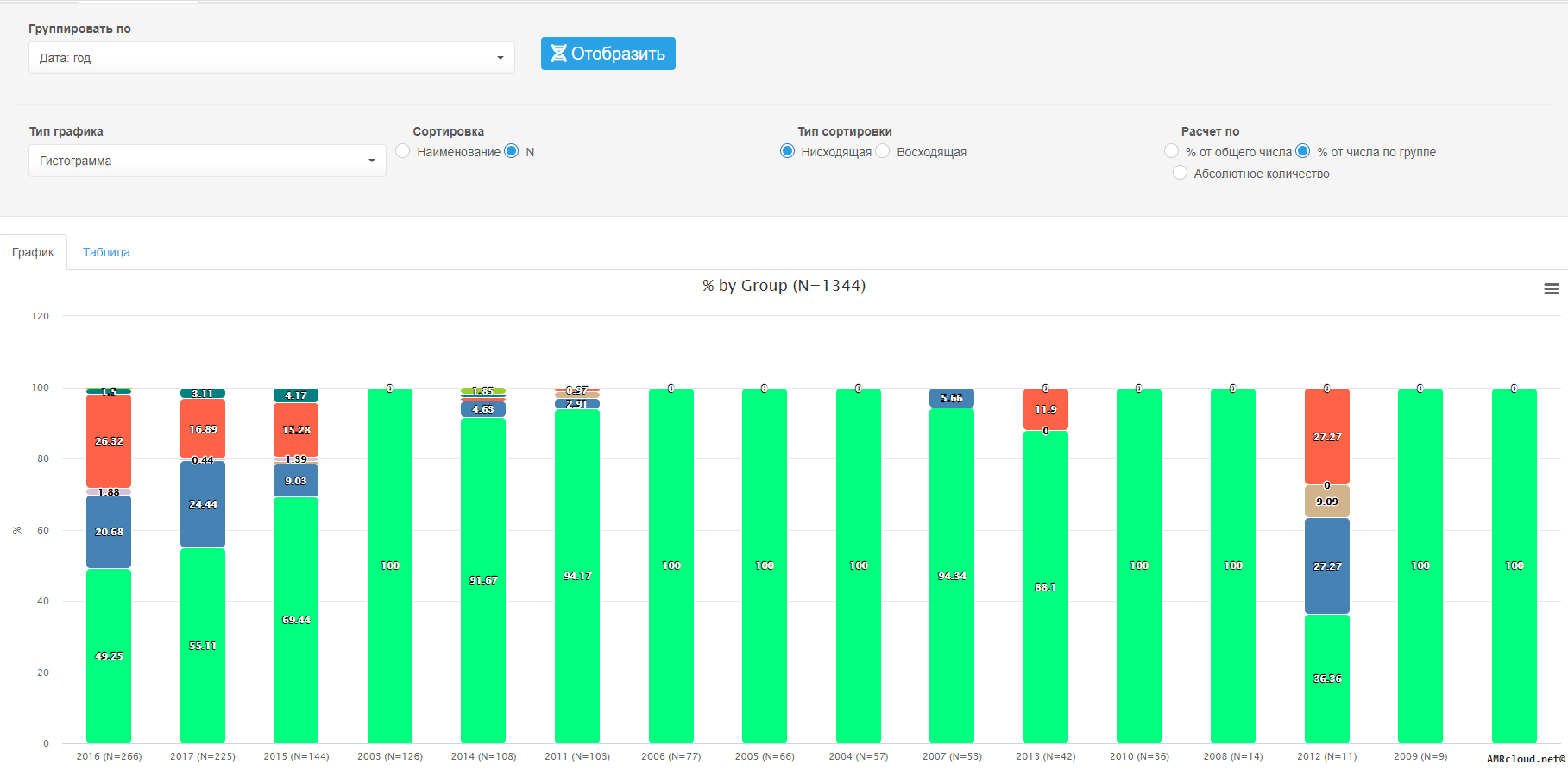
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку Группировать по.
- Выбор параметра для оценки из выпадающего списка.
- Нажатие кнопки Отобразить.
- Настройка подвкладки.
- Выбор подраздела.
5.3.3.3 Подвкладка «Карта»
Назначение:
- Оценка распределения долей маркеров в наборе данных в зависимости от географического расположения.
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку Карта.
- Нажатие кнопки Отобразить.
- Настройка размера точек.
5.3.3.4 Подвкладка «Карта х2»
Назначение:
- Одновременная оценка распределения долей маркеров и результатов определения чувствительности в зависимости от географического расположения.
Особенности:
- Выбор источника данных S/I/R:
- объединенные данные S/I/R (столбцы АМП_sir);
- автоинтерпретация из МПК (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_mic);
- автоинтерпретация из диаметров зон подавления роста (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_dd);
- Выбор АМП;
- Выбор категории: SIR, S+I, R, R+I.
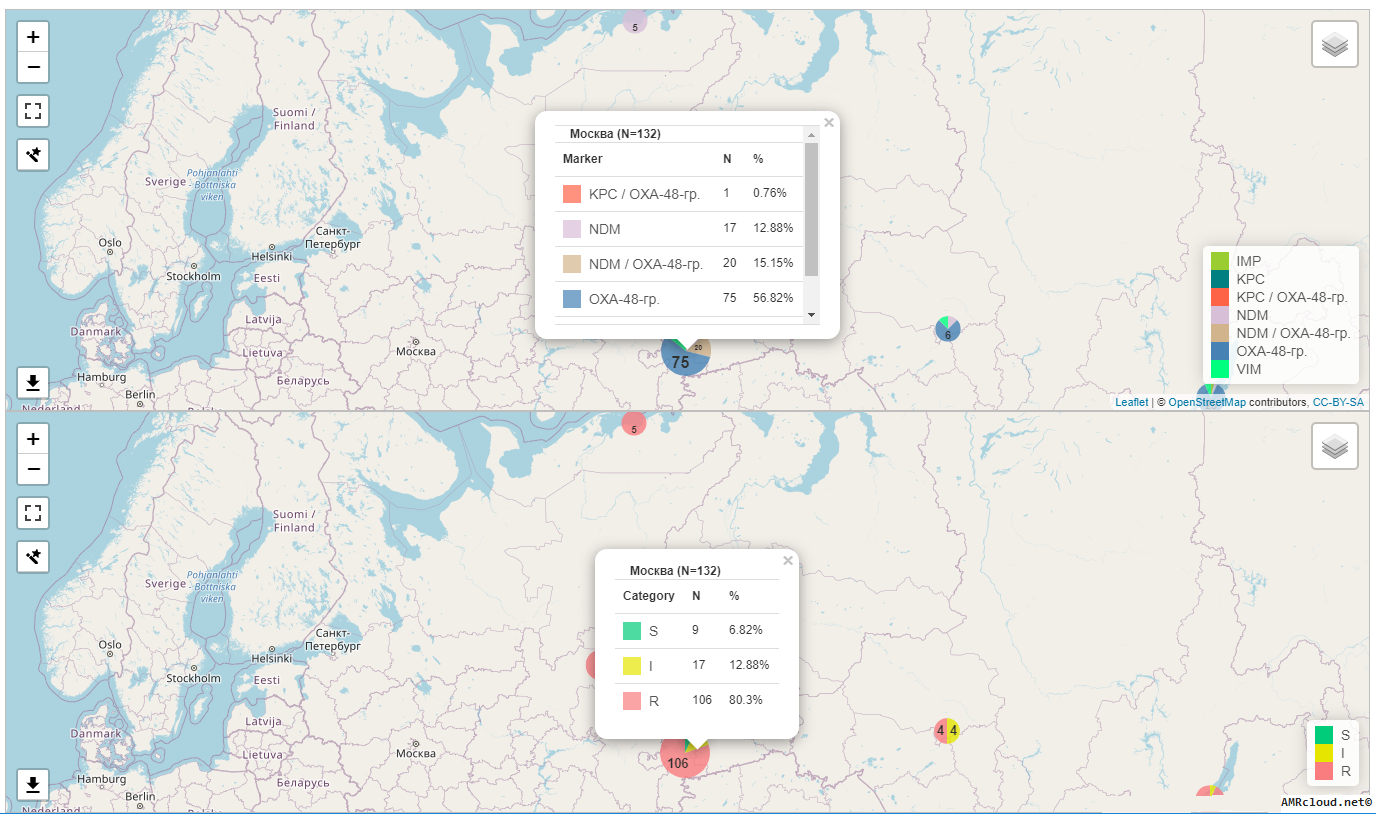
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку Карта х2.
Настройка Подвкладки Карта х2 - Выбор Источника данных S/I/R
- Выбор антимикробного препарата из выпадающего списка.
- Выбор категории.
- Нажатие кнопки .Отобразить.
- Настройка размера точек.
5.3.3.5 Подвкладка «Антибиотики (все)»
Назначение:
- Сводное представление категорий чувствительности микроорганизмов с учетом выбранных маркеров.
Особенности:
- Выбор источника данных S/I/R:
- объединенные данные S/I/R (столбцы АМП_sir);
- автоинтерпретация из МПК (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_mic);
- автоинтерпретация из диаметров зон подавления роста (клинические категории чувствительности, вычисленные AMRcloud в результате применения выбранных критериев интерпретации к столбцам АМП_dd);
- Подразделы: «График», «Таблица».
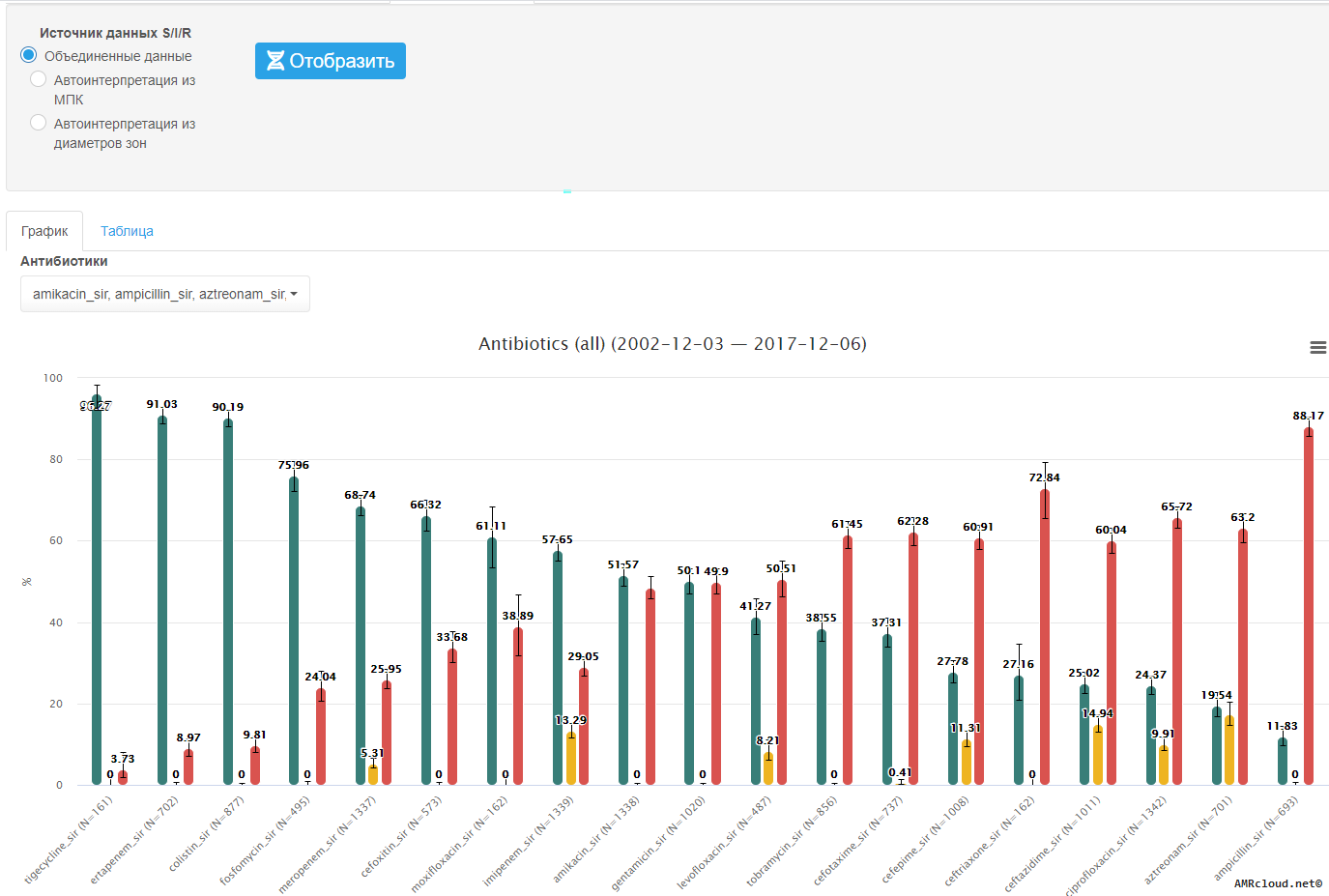
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Выбор Источника данных S/I/R.
- Нажатие кнопки Отобразить.
- Переход в подраздел.
- Выбор списка антимикробных препаратов.
- Поиск записей с использованием фильтра.
Пункт 5 приведен для подраздела График. Пункт 6 приведен для подраздела Таблица.
5.3.3.6 Подвкладка «МПК»
Назначение:
- Анализ распределения маркеров устойчивости в зависимости от значений МПК выбранного антимикробного препарата.
Особенности:
- Подраздел «МПК тренд» – распределение маркеров устойчивости в зависимости от значений МПК выбранного антимикробного препарата с получением информации по выделенной области значений. Получение информации по выделенной области включает распределение МПК, таблицу с данными МПК + метаданные по каждому изоляту с маркерами устойчивости.

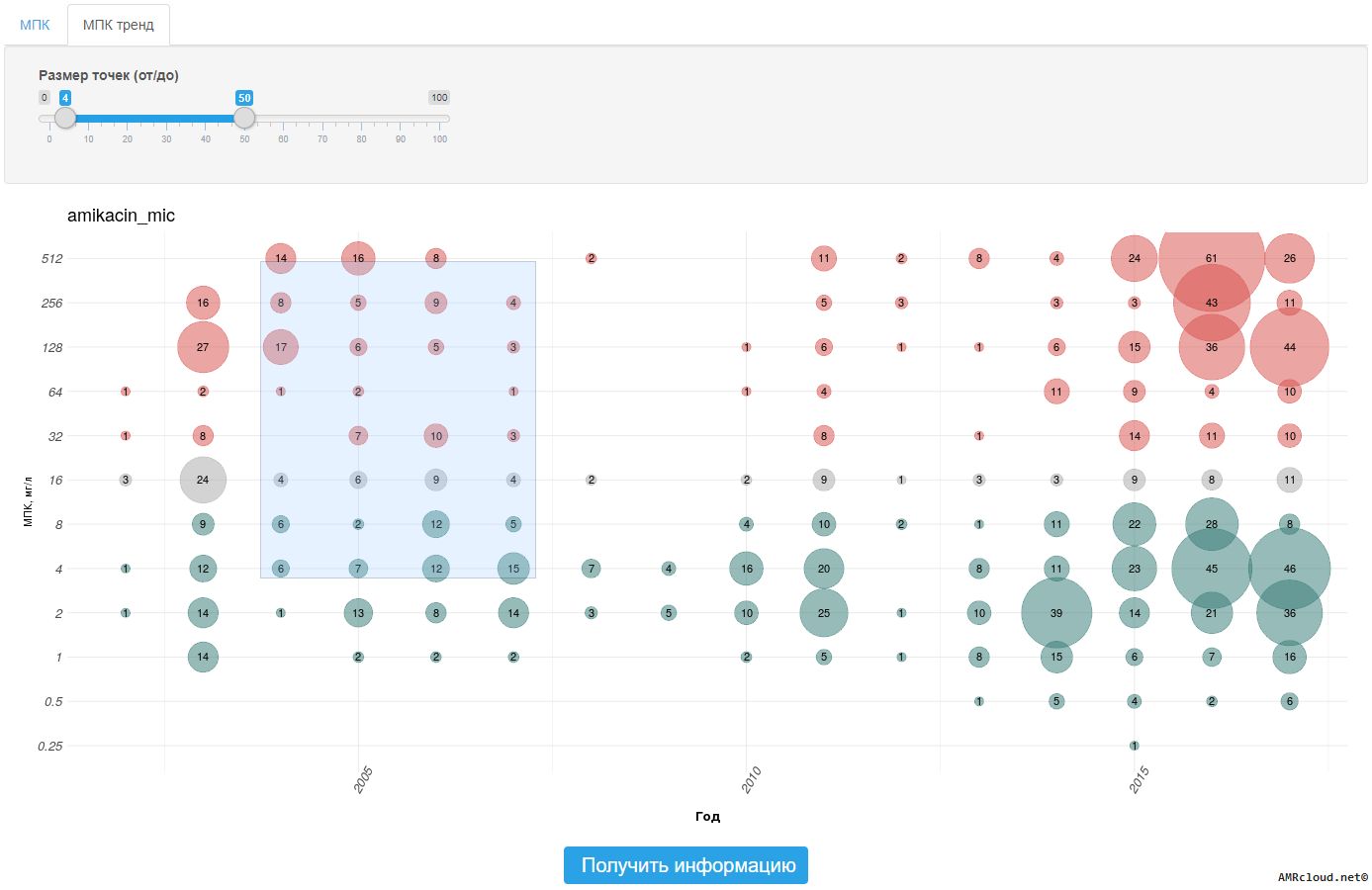
Алгоритм работы:
- Выбор группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку МПК.
- Выбор антимикробного препарата (из выпадающего списка).
- Нажатие кнопки Отобразить.
- Настройка вкладки (размер точек).
- Выделение необходимой области (серии «точек») с помощью левой кнопки мыши.
- Нажатие кнопки Получить информацию.
Пункты 6–8 приведены для подраздела МПК тренд.
5.3.3.7 Подвкладка «Диаметры зон»
Назначение:
- Анализ распределения маркеров устойчивости в зависимости от значений диаметров зон подавления роста выбранного АМП.
Особенности:
- Подраздел «Диаметры зон (тренд)» – распределение маркеров устойчивости в зависимости от значений диаметров зон подавления роста выбранного АМП с получением информации по выделенной области значений. Получение информации по выбранной области включает распределение значений диаметров зон подавления роста, таблицу с данными диаметров зон подавления роста + метаданные по каждому изоляту с маркерами устойчивости.

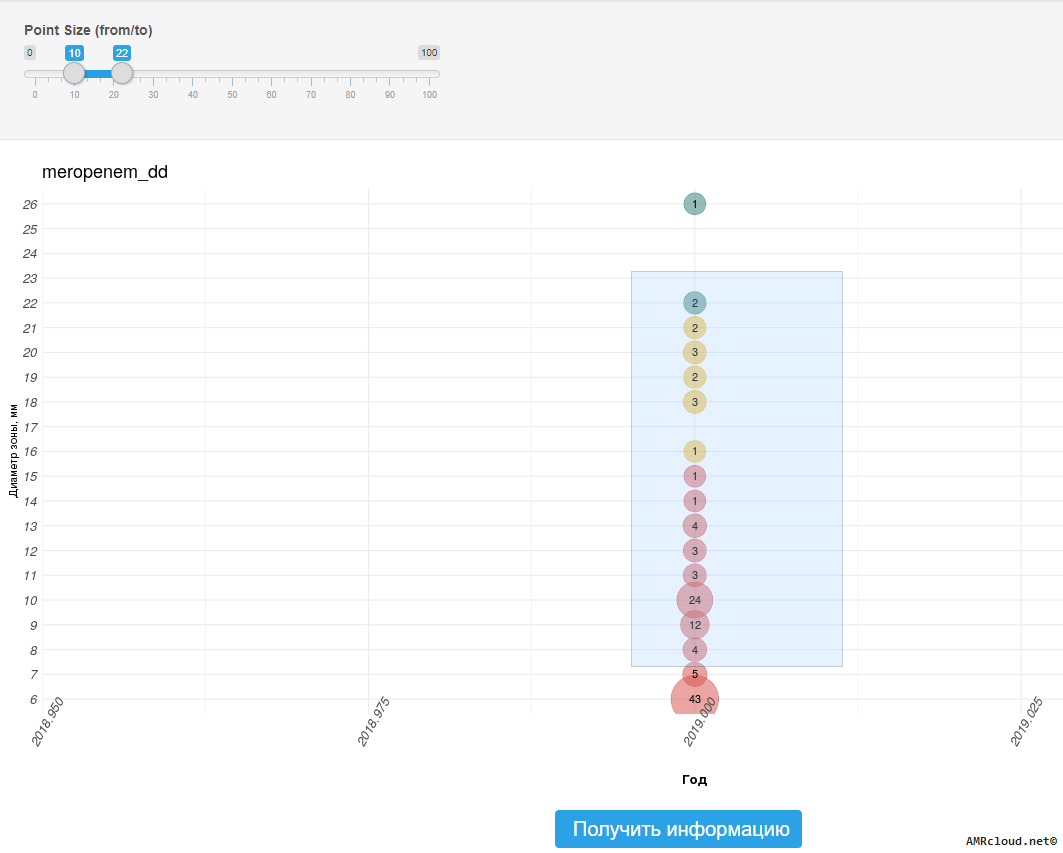
Алгоритм работы:
- Выбор группы микроорганизмов и других значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем для группы микроорганизмов – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку Диаметры зон.
- Выбор антимикробного препарата (из выпадающего списка).
- Нажатие кнопки Отобразить.
- Настройка вкладки (размер точек).
- Выделение необходимой области (серии «точек») с помощью левой кнопки мыши.
- Нажатие кнопки Получить информацию.
Пункты 6–8 приведены для подраздела Диаметры зон (тренд).
5.3.3.8 Подвкладка «Время первого обнаружения»
Назначение:
- Обозначение на временной шкале даты первого выявления маркеров с учетом установленного параметра (параметр – это любой из столбцов метаданных, кроме группы «Отметки изолятов». Например, клинический материал, локализация инфекции и т.д.). Подробная информация про метаданные представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
Алгоритм работы:
- Выбор необходимых значений в фильтрах поля Параметры основные. При первом знакомстве с загруженным набором данных рекомендуется сразу переходить во вкладку Маркеры (т.е. просмотреть данные в общем – не используя фильтры, затем выполнить выбор фильтров в поле Параметры основные).
- Выбор группы маркеров и перечня конкретных маркеров из выпадающих списков.
- Переход в подвкладку Время первого обнаружения.
- Выбор маркера из выпадающего списка (если в пункте 2 выбран более чем 1 маркер).
- Выбор параметра для оценки из выпадающего списка.
- Нажатие кнопки Отобразить.
5.4 Актуализация информации
Одним из основополагающих принципов мониторинга АР является непрерывность. Соблюдение данного принципа невозможно без своевременного внесения новых данных и корректировки уже созданной таблицы с данными – актуализации информации.
Необходимость актуализации информации может быть обусловлена следующими причинами:
- Появление новых данных (новых изолятов) за определенный период времени (добавление новых строк: метаданные + результаты определения чувствительности).
- Новая дополнительная информация (добавление нового столбца).
- Новый АМП (добавление нового столбца).
- Новые особые метки изолятов: генетические маркеры, особые фенотипы, факторы вирулентности (добавление нового столбца).
- Обнаружение опечаток или неточностей в исходной таблице, во время проведения анализа данных в AMRcloud (изменения в ячейках).
- Применение новых критериев интерпретации результатов определения чувствительности к АМП (изменение столбцов с категориями чувствительности).
- Обновление пользовательских критериев интерпретации и сохраненных географических координат (изменение данных в аккаунте AMRcloud).
Для актуализации информации, с учетом возможных причин, предлагаются следующие варианты:
- Внесение исправлений в исходную пользовательскую таблицу.
- Внесение исправлений через AMRcloud (допустимо не во всех случаях).
5.4.1 Внесение исправлений в исходную пользовательскую таблицу
Для включения новой информации (столбцов, строк) и исправления содержимого ячеек необходимо выполнить работу с исходными таблицами. Исходная таблица – это пользовательская таблица, созданная с помощью программы Microsoft Excel (формат .xlsx или .csv), для последующей загрузки в AMRcloud. Данный вариант внесения исправлений является оптимальным.
Чтобы изменить информацию в AMRcloud, нужно пройти следующие этапы:
- Непосредственное внесение изменений в исходную таблицу Excel (формат .xlsx или .csv) или скачивание таблицы из AMRcloud. Для скачивания необходимо выбрать набор данных и нажать кнопку на панели инструментов Редактировать набор данных. Затем перейти во вкладку Скачать и нажать кнопку Скачать исходные данные. Во избежание возможных трудностей при последующей работе с Excel не рекомендуется менять кодировку (оставить по умолчанию Windows (кириллица)).
- После внесения исправлений в исходную таблицу с помощью Excel заново создать набор данных в AMRcloud и пройти шаги импорта.
- Выбрать набор данных, который будет изменен (т.к. он является устаревшим и есть более актуальный импортированный набор данных). На панели инструментов нажать кнопку Удалить.
Название наборов данных в рамках одного проекта должны различаться – с данной целью необходимо использовать уточняющее наименование, например, «Отчет_сентябрь_2020» и «Отчет2_сентябрь_2020». Необходимо отметить, что в подзаголовке названия набора данных можно кратко отражать важные изменения, например, уточнить, что данный набор данных является неактуальным ввиду появления новой версии данных. Несмотря на то, что создание нового набора является более простым вариантом, необходимо учитывать пользовательские ограничения в количестве наборов данных.
5.4.2 Внесение исправлений через AMRcloud
5.4.2.1 Применение новых/других критериев для интерпретации результатов определения чувствительности
- Выбрать набор данных, который необходимо изменить, кликнуть по нему левой клавишей мыши.
- На панели инструментов для редактирования набора данных нажать кнопку Редактировать набор данных.
- Перейти на Шаг 4: Категоризация S/I/R, сбросить текущий шаг.
- Выбрать из выпадающего списка подходящие критерии интерпретации.
- Нажать кнопку Завершить все шаги.
5.4.2.2 Внесение изменений с помощью раздела «Мой профиль»
В данном разделе можно редактировать пользовательские критерии интерпретации, а также сохраненные географические координаты.
Во вкладке Мои правила интерпретации доступна возможность изменить или удалить пользовательские правила.
Для географических объектов во вкладке Мои геообъекты с помощью панели инструментов можно добавлять или удалять сохраненные ранее географические координаты. При непосредственном нажатии на ячейку таблицы географические координаты становятся доступны для редактирования.
5.4.2.3 Информирование об актуализации данных
При внесении исправлений в набор данных пользователи AMRcloud (в том числе участники команды) могут отправлять ссылки всем заинтересованным специалистам. Подробная информация о генерации ссылок, их видах и настройках представлена в разделе 5.5. Взаимодействие участников.
5.5 Взаимодействие участников
Своевременное получение информации в рамках реализации локального мониторинга АР требует включение в процесс многих специалистов, наряду с доступностью важной информации. Решением данных вопросов в рамках рабоы с платформой AMRcloud является создание команды проекта и использование генератора ссылок.
5.5.1 Участники команды
Различные специалисты могут быть вовлечены в работу с платформой AMRcloud:
- Микробиологи;
- Эпидемиологи;
- Клинические фармакологи;
- Лечащие врачи;
- Представители администрации.
Подробная информация по созданию команды в AMRcloud представлена в разделе 5.1. Создание проектов и наборов данных. Команда, создание и назначение на проект. В зависимости от организации процесса работы с микробиологическими данными в медицинской организации степень участия каждого члена команды будет различна.
Таблица 5.5.1. Роли участников команды
| Роль | Участники |
| Формирование объединенной таблицы с результатами микробиологического исследования и дополнительными данными с учетом требований AMRcloud | Микробиолог Эпидемиолог Клинический фармаколог |
| Создание набора данных и проекта в AMRcloud (все шаги импорта данных) | Любой специалист – пользователь AMRcloud |
| Анализ сформированного набора данных в AMRcloud | Любой специалист – пользователь AMRcloud |
| Создание локальных протоколов АМП по результатам анализа в AMRcloud | Клинический фармаколог |
| Разработка и утверждение формулярного списка ЛС по результатам анализа в AMRcloud | Клинический фармаколог Администратор |
| Совершенствование комплекса профилактических мероприятий по результатам анализа в AMRcloud | Эпидемиолог Лечащий врач Администратор |
| Внесение исправлений в объединенную таблицу (опечатки, новые данные) | Любой специалист – пользователь AMRcloud |
| Обновление набора данных в AMRcloud (загрузка новой таблицы) | Любой специалист – пользователь AMRcloud |
| Индивидуальная настройка работы с AMRcloud (cоставление персональных критериев интерпретации, сохранение географических координат, создание команды) | Любой специалист – пользователь AMRcloud |
Алгоритм работы с AMRcloud может изменяться в зависимости от наличия в структуре медицинской организации микробиологической лаборатории и с учетом ролей участников локального мониторинга. Примеры функционирования локального мониторинга для создания протокола АМТ представлены на Рисунках 1-2.
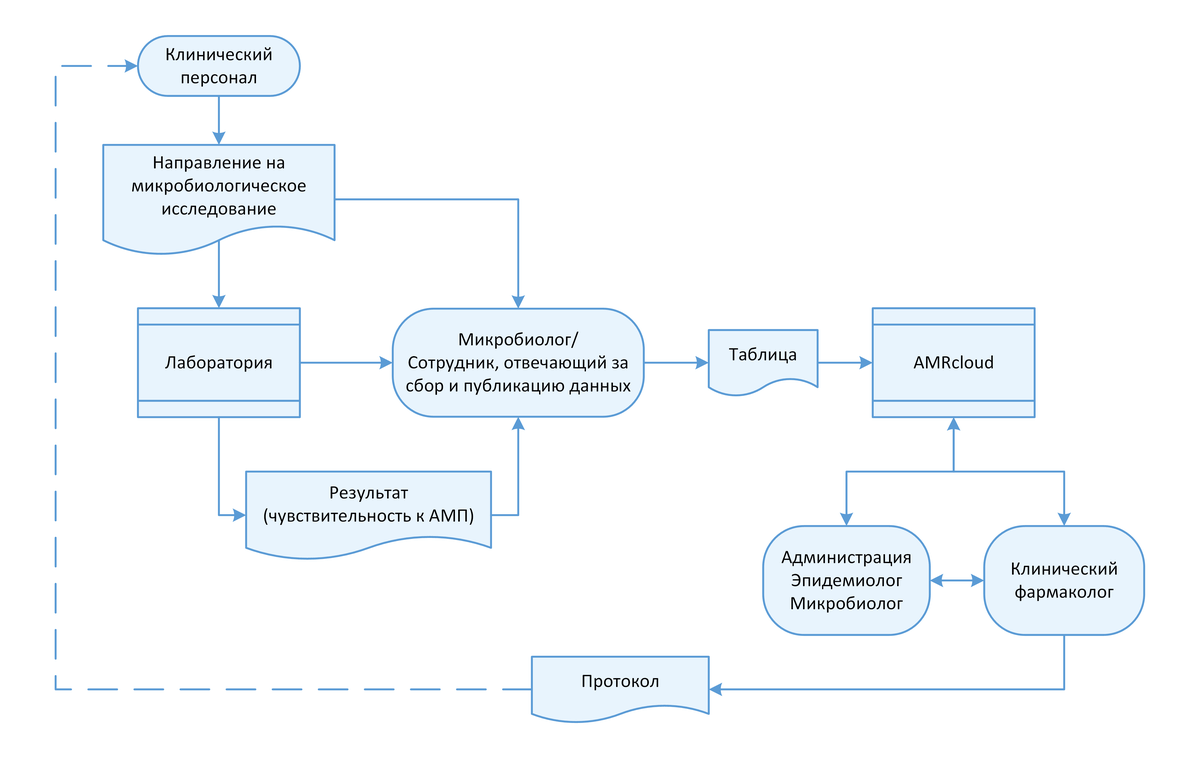
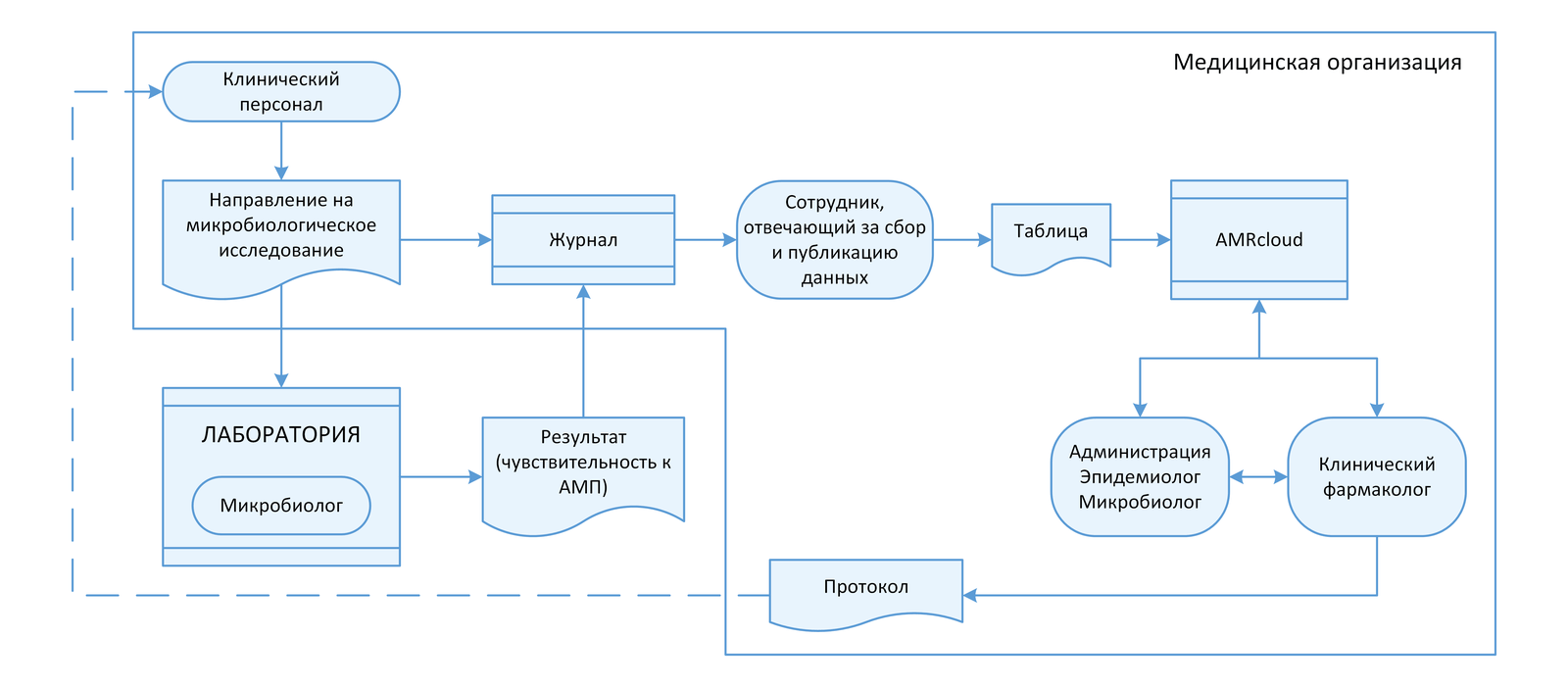
Платформа AMRcloud помогает выстроить локальную систему мониторинга. При этом универсальные требования, которые применяются в отношении исходной таблицы, а также доступность широкого круга возможностей по анализу данных в AMRcloud позволяют привлечь пользователей различных специальностей на любом из этапов работы.
5.5.2 Создание ссылок
Результатами анализа данных (таблицами, графиками и т.д.) можно поделиться не только среди участников команды. Для этого, не переходя со страницы полученных результатов (при запущенном для анализа наборе данных), следует воспользоваться генератором ссылок (кнопка расположена в правом верхнем углу экрана). Сгенерированную ссылку можно скопировать и отправить в сообщении (например, e-mail) всем заинтересованным специалистам, однако для полноценного взаимодействия в рамках локального мониторинга рекомендуется использовать функционал командного взаимодействия. Подробная информация по командному взаимодействию представлена в разделе 5.1. Создание проектов и наборов данных.Команда, создание и назначение на проект.
Варианты ссылок
По наличию защиты:
- Открытые (без установленного пароля),
- Закрытые (с установленным паролем).
По уровню доступа:
- Ограниченные (разрешается просматривать только фрагмент набора данных, при изменении параметров фильтра или вкладок, данные отображаться не будут);
- Полные (при переходе по ссылке можно изменять фильтры и выбирать вкладки для анализа).
Выбор варианта (настройки) ссылки представлены на странице Генератор ссылок.
Просмотр созданных ссылок возможен в разделе Мой профиль → вкладка Ссылки. Здесь представлены все ссылки вне зависимости от набора данных и проекта.
Для просмотра, редактирования и удаления ссылки следует выбрать нужный набор данных, воспользоваться кнопкой Редактировать набор данных на панели инструментов и перейти во вкладку Ссылки.
5.6 Использование AMRcloud при формировании локальных протоколов антимикробной терапии
Результаты работы локальной системы мониторинга могут быть использованы при создании рекомендаций, протоколов и формуляров, а также других вариантов необходимой медицинской документации. Для описания всего комплекса мероприятий требуется оценить широкий объем информации, в том числе метаданные. Метаданные или дополнительные данные могут быть связаны с изменениями результатов чувствительности микроорганизмов к различным АМП, поэтому их включение в систему мониторинга имеет особое значение. Подробная информация по метаданным представлена в разделе 4.3. Сопроводительная эпидемиологическая, клиническая и демографическая информация.
В рамках создания протоколов АМП могут быть достигнуты следующие цели:
- Разработка общих подходов к АМТ.
- Создание дифференцированных схем терапии.
- Разработка схем терапии проблемных возбудителей.
Таким образом, применение AMRcloud обеспечивает доказательную базу разработанным протоколам АМП за счет широкого объема аналитических возможностей и сохранения полученных результатов.
Таблица 5.6.1. Решение поставленных задач с помощью AMRcloud
| Задачи | Применение AMRcloud | |
| Сбор данных по единому стандарту (с соблюдением разработанных локальных требований), с включением записей за период не менее 1 года | Проверка записей на основе требований подготовки данных для AMRcloud | |
| Анализ данных | Выявление наиболее распространенных возбудителей в отделении/стационаре | Вкладка «Микроорганизмы» |
| Определение спектра активности АМП в отношении выбранной группы микроорганизмов | Вкладка «Антибиотики (все)» | |
| Анализ активности АМП в отношении определенного возбудителя с учетом различных стратификаций | Вкладка «Выбранный антибиотик» | |
| Анализ активности АМП при наличии устойчивости возбудителя к другому антимикробному препарату | Вкладка «Ассоциированная устойчивость» | |
| Оценка распространенности механизмов устойчивости возбудителя в зависимости от различных факторов риска | Вкладка «Маркеры» | |
| Сравнение данных (результатов определения чувствительности, метаданных) в различных отделениях или между группами пациентов | Вкладка «Сравнение» | |
| Внесение полученных результатов анализа данных – графиков и таблиц в созданный протокол | Скачивание диаграмм и таблиц при работе с любой вкладкой | |
6. Приложения
Основные схемы и таблицы, представленные в руководстве, можно скачать по адресу или QR-code:
https://monitoring.amrcloud.net/files/cheatsheet.pdf

6.1 Шаблон направления на микробиологическое исследование

Ссылка для скачивания шаблона: https://monitoring.amrcloud.net/files/appointment.docx
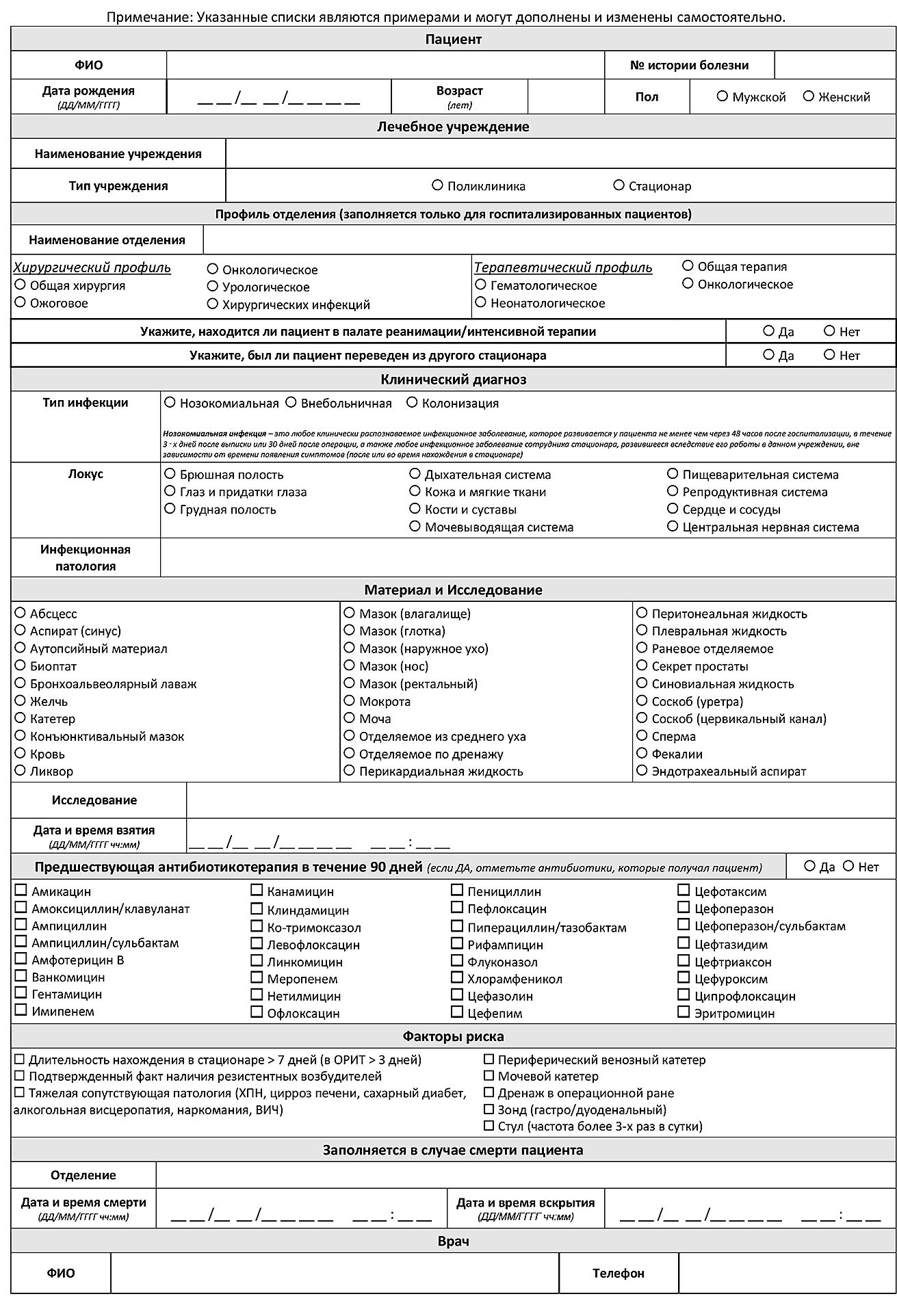
6.2 Работа с шаблоном, справочниками и словарями
6.2.1 Шаблон данных
Шаблон данных является базовым элементом для организации мониторинга АР и представляет файл формата .xlsx и включает себя следующие страницы:
- «ИРК_1» – пример таблицы для ввода данных.
- «Словари» – наборы справочных наименований (микроорганизмы и группы микроорганизмов, профиль отделения, локализация инфекций, клинический материал).
- «Антибиотики справка» – наименования АМП с учетом метода определения чувствительности (суффиксы формата «_mic», «_dd», «_sir», подробнее см. раздел 4.4. Правила оформления и хранения данных.
Шаблон данных может быть скачан по ссылке https://monitoring.amrcloud.net/files/template.xlsx и адаптирован с учетом специфики конкретного учреждения.

6.2.2 Добавление новых строк в шаблон
При внесении данных в шаблон может возникнуть ситуация, когда подстановки будут активны только для окрашенных ячеек, а если продолжать заполнять таблицу, то выпадающие списки пропадут. Для того, чтобы выпадающие списки с подстановками продолжали формироваться и для новых строк, необходимо добавлять такие строки следующим образом:
- Выделить первую свободную строку после заполненнных. На панели инструментов, на вкладке Главная, в разделе Ячейки, нажать кнопку Вставить, выбрать пункт Вставить строки на лист.
6.2.3 Добавление новых значений в существующие словари/создание новых словарей
Допустим, в файл Шаблон_AMRcloud.xlsx потребовалось добавить новые значения во встроенные справочники. Например, в список отделений необходимо внести новое отделение – Нейрохирургия. Для этого необходимо сделать следующее:
- Перейти на лист Словари, найти столбец с нужным справочником и добавить недостающее значение (напечатав значение в ячейку).
- На листе ИРК_1 выделить диапазон ячеек, в котором содержатся значения отделений. На панели инструментов во вкладке Данные, в разделе Работа с данными нажать на кнопку Проверка данных и выбрать пункт Проверка данных.
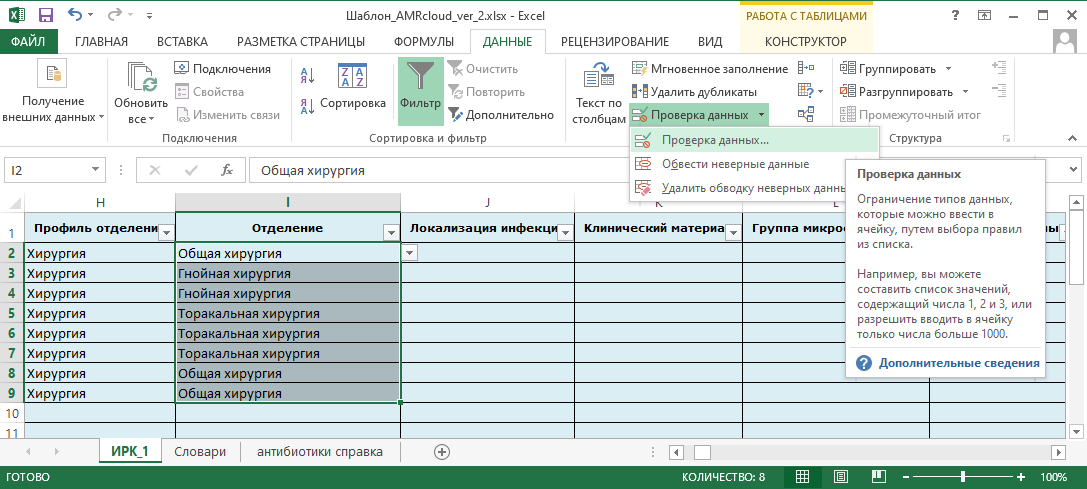
- В открывшемся диалоговом окне Проверка вводимых значений выбрать Тип данных: Список, отметить галочку Список допустимых значений, и после клика в поле Источник выделить рамкой диапазон ячеек на листе Словари, в которых будут храниться значения подстановки. Нажать ОК.
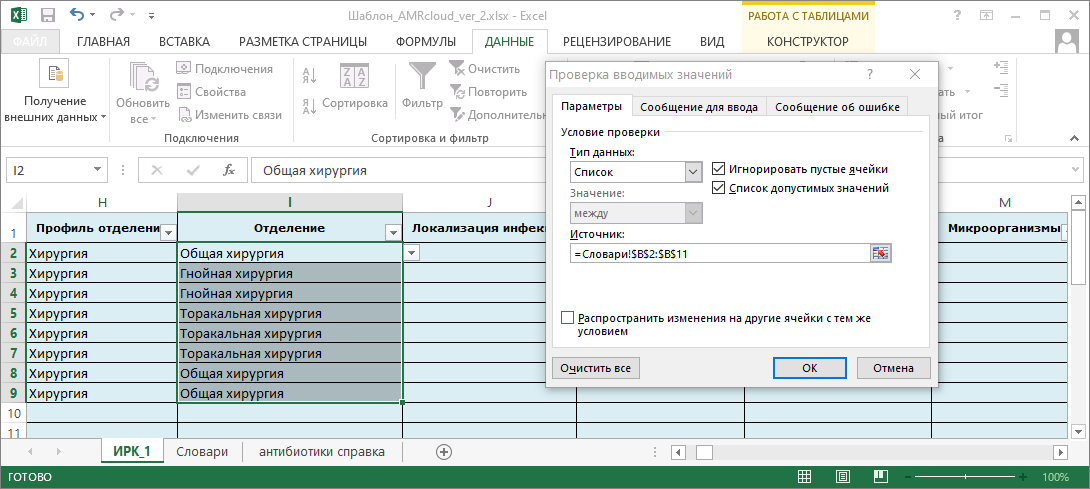
6.2.4 Замена существующих значений
Допустим, в таблице имеется заполненный столбец с кодами отделений и его требуется перекодировать в полное наименование. Например, закодированы они как ХИР1, ХИР2, ХИР3, ХИР4. Необходимо их автоматически переименовать в «Общая хирургия», «Урология», «Гнойная хирургия», «Торакальная хирургия».
Для этого следует использовать встроенную в Excel функцию ВПР().
Синтаксис функции следующий:
ВПР(искомое_значение; таблица; номер_столбца; интервальный_ просмотр)
- искомое значение – значение, которое необходимо заменить;
- таблица – ссылка на диапазон ячеек со словарем, который будет использоваться при замене;
- номер_столбца – номер столбца из таблицы, значения которого будут использоваться в качестве замены;
- интервальный_просмотр – может принимать 2 значения: ИСТИНА (поиск значения ближайшего к критерию или совпадающее с ним) и ЛОЖЬ (поиск значения в точности совпадающее с критерием).
Общий порядок действий для осуществления такой замены следующий.
- Создать словарь замен на новом листе.
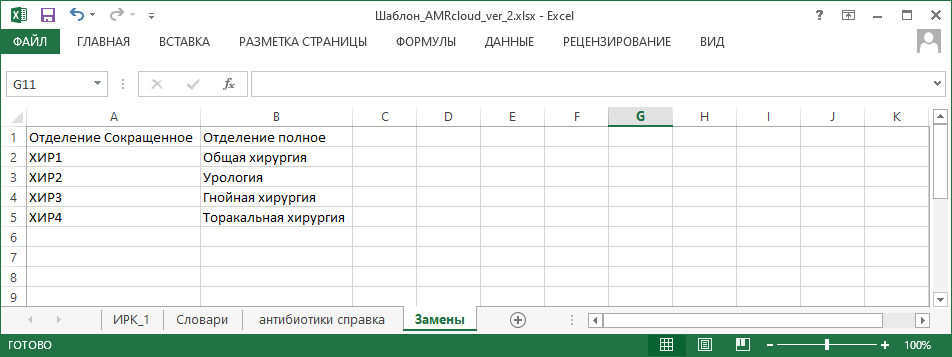
- На листе ИРК, рядом со столбцом с сокращенными названиями Отделение Сокр, добавить два новых пустых столбца – Отделение Полное 1 и Отделение Полное 2. В первый столбец вставить функцию =ВПР(I2; Замены!$A$2:$B$5;2;ЛОЖЬ), где:
- I2 – ссылка на значение, которое предполагается заменить (в примере это столбец I, строка 2);
- Замены!$A$2:$B$5 – ссылка на диапазон ячеек, в котором содержатся данные для замены;
- 2 – это номер столбца в диапазоне ячеек для замен, откуда будут браться полные значения;
- ЛОЖЬ – мы сообщаем функции, что нужно использовать точное совпадение.
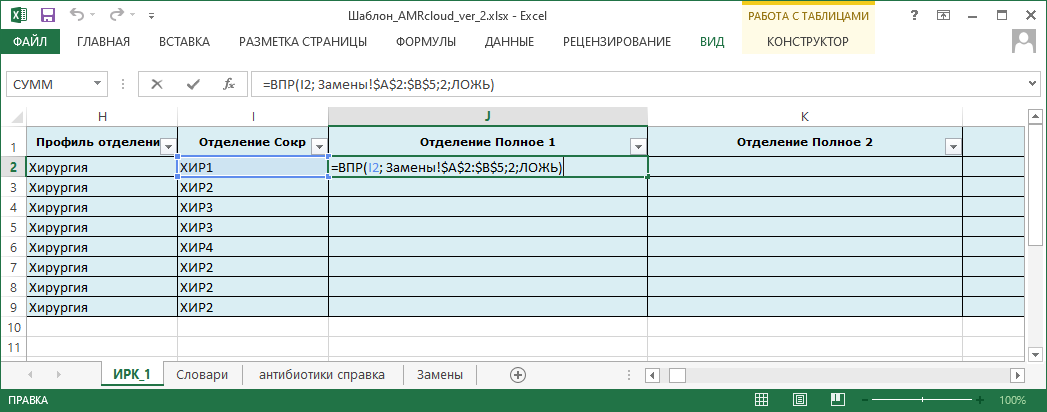
- После растягивания функции на все ячейки скопировать результат и вставить в столбец Отделение Полное 2 в режиме Значения. Столбец (Отделение Полное 1) с функцией удалить.
6.2.5 «Разворачивание» таблицы
Возможна ситуация, когда ЛИС либо иная информационная система выдает отчет в формате, где информация по одному изоляту содержится более чем в одной строке. Например, результаты тестирования для каждого из изолятов написаны в строках, при этом идентификатор изолята повторяется.
Для приведения такой таблицы к формату, пригодному для загрузки в AMRcloud (где требуется, АМП находились в столбцах, а запись для каждого изолята была представлена единственной строкой) следует выполнить ряд действий.
- Добавить новый столбец и пронумеровать все имеющиеся строки.
- На вкладке Вставка панели инструментов нажать кнопку Сводная таблица. В диалоговом окне выбрать таблицу с учетом добавленного столбца с номерами строк и отметить, что отчет сводной таблицы следует поместить на новый лист.
- Excel предложит заполнить поля сводной таблицы, перетаскивая поля мышкой в соответствующие области. В качестве строк необходимо выбрать идентификатор изолята (RecordID), в качестве столбцов – АМП (Antibiotic), а для значений выбрать номера строк (Номер строки). Сводная таблица в Excel может работать только с числовыми значениями, поэтому и был дополнительно создан суррогатный столбец (Номер строки). Агрегирующая функция для значений не играет роли, так как все значения уникальны. После «разворота» таблица примет вид, показанный на рисунке ниже.
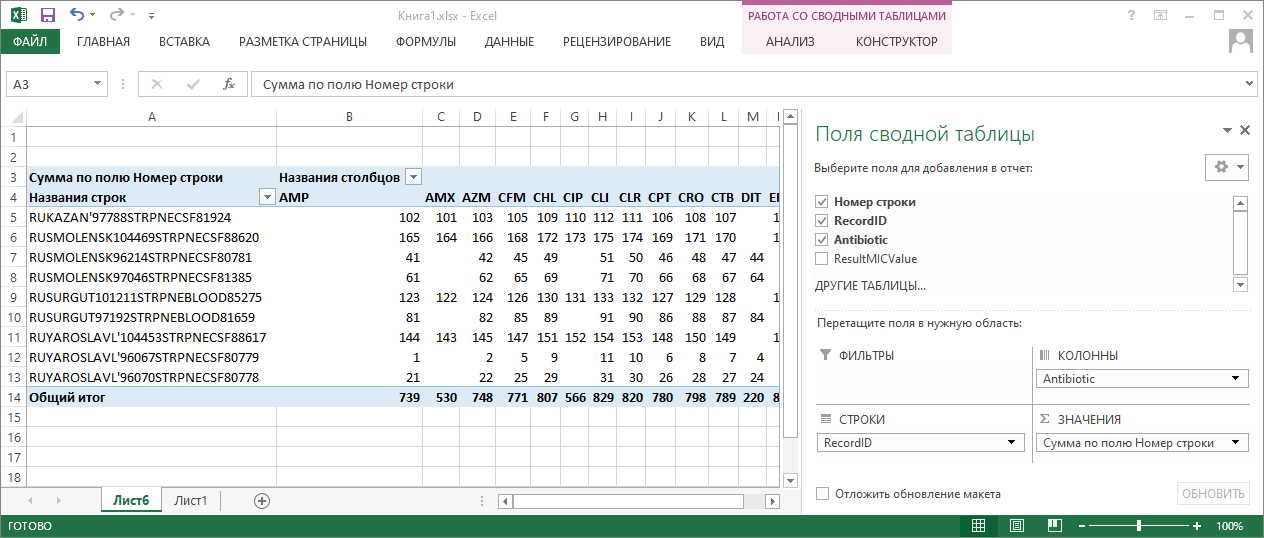
- Скопировать таблицу без итогов и вставить как значения на новый лист.
- Продублировать имена столбцов АМП для вставленной таблицы и добавить к ним коды («_sir», «_mic» или «_dd»), в представленном примере – _mic.
-
В новых столбцах с кодом «_mic» необходимо вставить значения МПК из исходного варанта таблицы (т.е. до разворота) с помощью функции ВПР(). Подробнее работа с функцией ВПР() описана выше. В качестве кодов выступает столбец Номера строк, в качестве замен – четвертый столбец ResultMICValue оригинальной таблицы.
В пустые значения необходимо вставить функцию для замены номеров строк на МПК =ЕСЛИ(ЕПУСТО(B2); «»;ВПР(B2; Лист1!$A$1:$D$189; 4; ЛОЖЬ))
В данной формуле проверяется, пустая ли ячейка, и если не пустая, то значение номера строки заменяется на МПК.
- B2 – ячейка, содержащая номер строки, который подлежит замене
- Лист1!$A$1:$D$189 – диапазон ячеек с данными исходной таблицы из пункта 1. Из этой таблицы мы и будем брать значения МПК для вставки вместо номеров ячеек
- 4 – для замены необходимо использовать четвертый столбец
- ЛОЖЬ – задается использование точного совпадения
Результат работы этой функции показан на рисунке ниже.
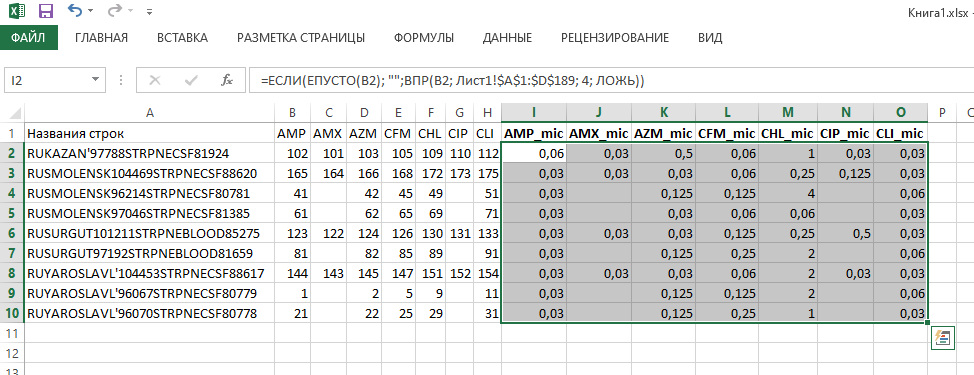
- Скопировать полученные данные (столбцы с кодами АМП и значениями МПК) и вставить как значения на место столбцов с АМП и номерами строк. Ячейки с формулами удалить.
6.3 Работа с WHONET
WHONET – это бесплатное настольное приложение Windows для управления и анализа данных микробиологических лабораторий с особым упором на наблюдение за устойчивостью к АМП. Программа разработана и поддержана Сотрудничающим центром ВОЗ по надзору за устойчивостью к АМП [68] .
Официальный сайт программы, где она доступна для загрузки – https://whonet.org.
Помимо встроенных справочников материалов, организмов и антибиотиков, программа имеет уже настроенные критерии интерпретации чувствительности EUCAST и CLSI (M100, M45, M60, M61, доступ к беcплатным ресурсам, в том числе ветеринарным VET03, VET04, VET06 и VET08), настроенные панели тестирования для каждого вида микроорганизмов и встроенную экспертную систему.
WHONET также включает модуль импорта данных под названием BacLink для сбора и стандартизации данных из существующих настольных приложений, лабораторных приборов и лабораторных информационных систем [69] . Подробнее о работе с данным модулем можно прочитать на официальном сайте программы, а также в разделе, посвященном работе с BacLink.
Механизмы построения отчетов предоставляют широкие возможности для анализа данных чувствительности. Данное руководство не ставит себе целью описывать все возможности программы WHONET, для этого лучше обратиться к официальной документации программы [70] , либо воспользоваться справкой в самой программе.
6.3.1 Создание новой лаборатории
Для начала работы с WHONET необходимо создать и сконфигурировать новую лабораторию [71] . При первом запуске программы предлагается выбрать лабораторию с демонстрационными данными.
Чтобы создать новую лабораторию, в стартовом окне программы необходимо нажать кнопку Новая лаборатория, в открывшемся окне выбрать страну Российская Федерация, ввести произвольное название лаборатории и указать её код. Код следует указывать латинскими символами, так как он будет использоваться в качестве расширения по умолчанию для всех файлов, связанных с созданной лабораторией. В дальнейшем при создании новых лабораторий можно копировать конфигурации лабораторий с помощью кнопки Скопировать лабораторию.
После этого необходимо пройти дополнительные шаги настройки, связанные с непосредственной деятельностью лаборатории, поэтому требуется заранее собрать информацию о АМП и методах тестирования, используемых в лаборатории, а также о стационарах, с которыми взаимодействует лаборатория и откуда поступают образцы.
6.3.1.1 Настройка антибиотиков (обязательно)
Для открытия окна настройки антибиотиков, методов и контрольных точек нужно нажать кнопку Антибиотики. Появится окно со списком антибиотиков, доступных в WHONET, (слева) и списков антибиотиков для работы в лаборатории.
Порядок добавления антибиотиков в лабораторию следующий:
- Выбрать надлежащие рекомендации (рекомендуется EUCAST).
- Кликнуть на нужный метод тестирования (DDM, MIC, Е-тест).
- Выбрать подходящий антибиотик (и нагрузку диска, если речь идет о диско-диффузионном методе).
Чтобы выбрать антибиотик, нужно дважды кликнуть по нему или же кликнуть по нему один раз, а затем нажать на кнопку со стрелкой вправо –>. Для удаления выбранного антибиотика нужно нажать кнопку со стрелкой влево <–.
Каждому антибиотику присваивается код (до девяти знаков), который включает в себя:
- трехбуквенный код антибиотика;
- однобуквенный код, отсылающий к типу используемых критериев интерпретации (например, E=EUCAST);
- однобуквенный код, обозначающий метод тестирования (D=DDM, M=MIC, E=ETest);
- нагрузка диска (при тестировании диско-диффузионным методом).
В отношении МПК или ETest необходимо выбрать только нужный антибиотик и соответствующие критерии интерпретации. Например, код ATM_ED30 расшифровывается следующим образом: азтреонам, EUCAST, DDM, 30 μg, тогда как ATM_EM означает: азтреонам, EUCAST, MIC.
Дальнейшие настройки антиботиков необязательны, но тем не менее могут быть полезны.
6.3.1.2 Пограничные значения для антибиотиков (Breakpoints) (необязательно)
При вводе данных чувствительности программа автоматически интерпретирует их, исходя из установленных пограничных значений. В большинстве случаев их менять не требуется, но для особых случаев предусмотрена такая возможность. Чтобы открыть окно редактирования пограничных значений, требуется нажать кнопку Breakpoints.
Доступны следующие опции:
- Общие – используются для установки пограничных значений для всех видов.
- Видоспецифические – используются для установки пограничных значений для конкретных видов, по приоритету стоят выше общих (для EUCAST заполняются только видоспецифические пограничных значения). Все изменения необходимо осуществлять именно здесь.
Квалифицированные правила интерпретации служат для создания экспертных правил интерпретации.
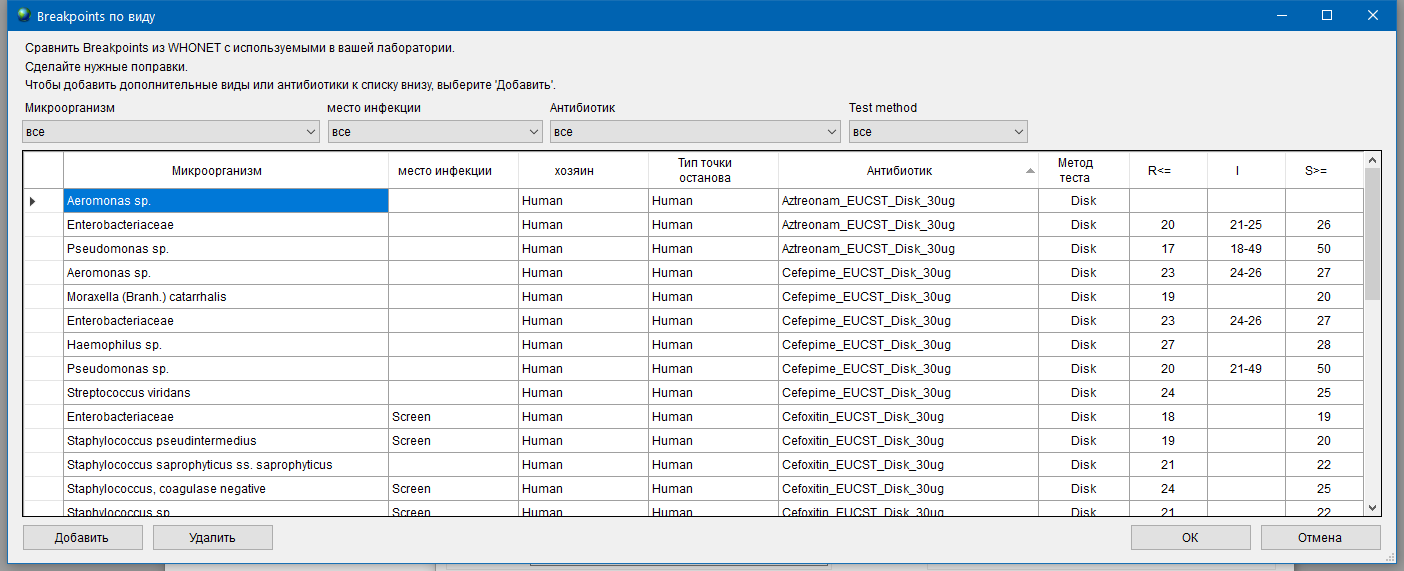
6.3.1.3 Наборы антибиотиков (Панели) (необязательно)
В целях упрощения ввода данных присутствует возможность настроить панели тестируемых антибиотиков для каждой группы микроорганизмов с помощью кнопки Наборы. В открывшемся окне можно указать, какие антибиотики тестируются по каждому виду микроорганизмов. Однако в момент ввода данных допускается добавление результатов для антибиотиков, не предусмотренных в панели.
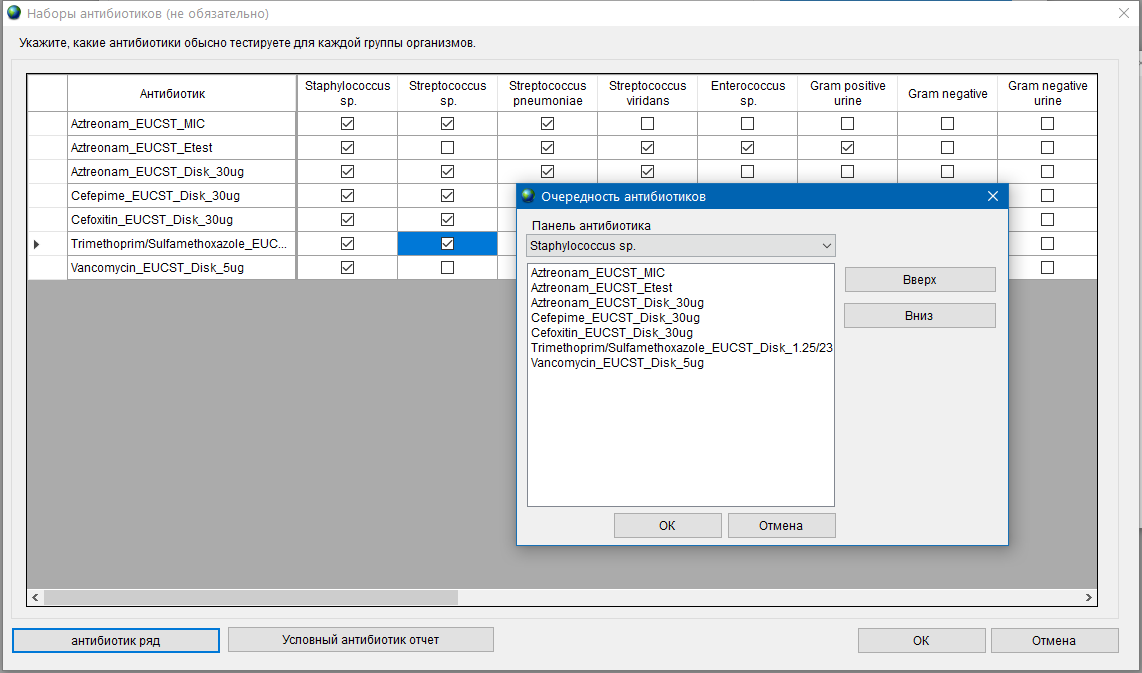
6.3.1.4 Отделение
Данная опция является необязательной с точки зрения работы программы WHONET, но обязательна в рамках организации мониторинга АР.
В рамках данного справочника можно завести несколько отделений из разных учреждений. При этом используется трехуровневая иерархическая структура: Учреждение – Отделение – Тип отделения. Справочники учреждений и отделений при этом могут быть отредактированы самим пользователем.
6.3.1.5 Поля данных
Программа WHONET автоматически определяет набор стандартных полей данных. К стандартным полям относится такая информация, как уникальный идентификатор пациента, местонахождение, дата получения образца, тип образца, микроорганизм/возбудитель, панель антибиотиков.
Для организации полноценного мониторинга АР может потребоваться внесение дополнительных полей, таких как диагноз пациента, локализация инфекции, другие значимые данные. Чтобы добавить такие поля, требуется нажать кнопку Поля данных в окне настройки лаборатории. Откроется окно Поля данных, в нем необходимо нажать кнопку Изменить список.
Слева находятся списки категорий данных WHONET (клинические данные, инфекционный контроль и т.п.) и полей данных (диагноз, дата поступления пациента и т.п.), из которых пользователь может выбирать нужные ему категории и поля. Справа – поля, которые доступны в конфигурации текущей лаборатории. Поля добавляются двойным нажатием либо кнопкой –>.
Для того, чтобы добавить поле, которое отсутствует в списке, нужно дважды кликнуть по пункту (Определенный для пользователя…) и в появившемся окне указать название, тип и описание нового поля.
Для полей можно определить справочники, которые будут использоваться для заполнения. Это позволит снизить количество ошибок, а также увеличит скорость ввода за счет кодов. Чтобы определить такие справочники, необходимо выбрать поле и нажать кнопку Код список.
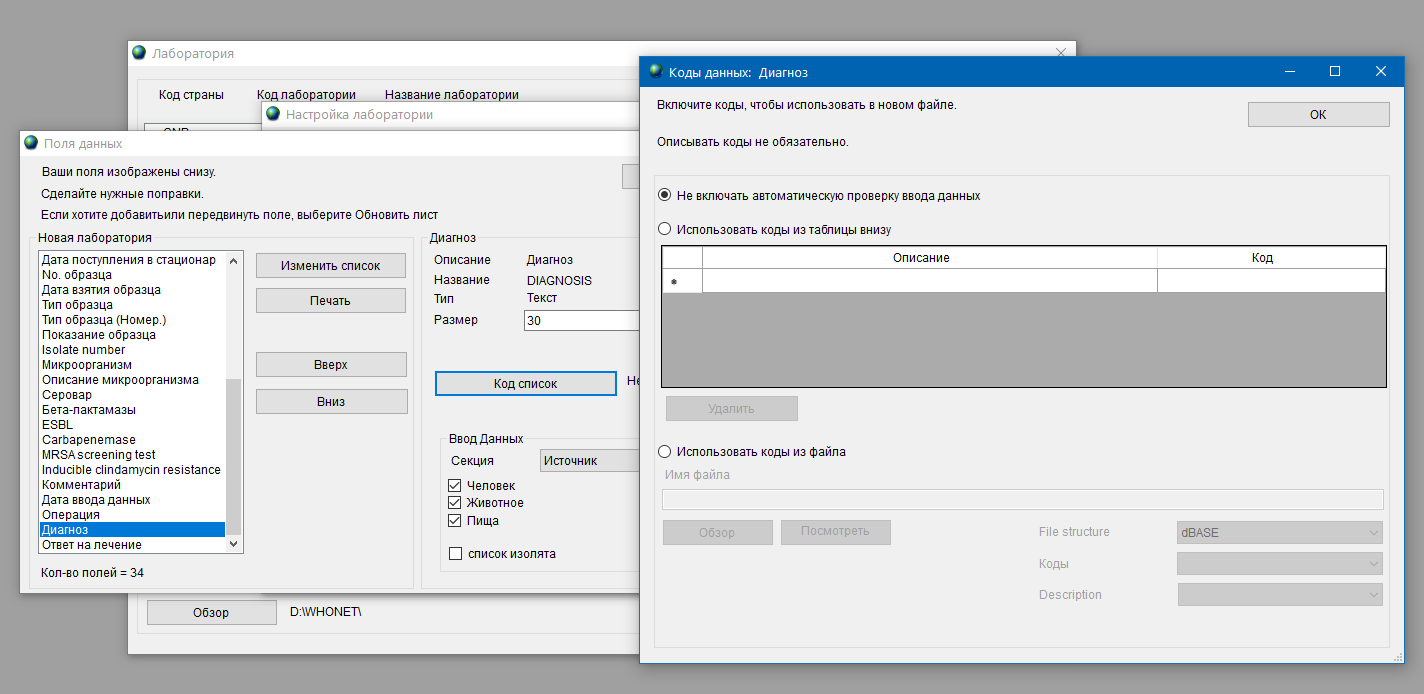
На этом настройку лаборатории можно считать завершенной. После сохранения конфигурации можно приступать к вводу данных. Остальные разделы настройки являются более специфичными и не относятся к теме данного руководства. Работа с ними подробно описана в официальной документации к программе.
6.3.2 Ввод данных
Для организации хранения и формирования интерфейса для ввода информации WHONET использует структуру данных, определенную текущими настройками лаборатории. Структура данных – это своего рода инструкции, как должен быть устроен файл с данными. На основании структуры данных для лаборатории можно создать один или несколько файлов с данными. Это может быть полезно в случаях, когда одной конфигурацией пользуется несколько лабораторий одновременно, либо одна лаборатория хранит данные за разные периоды в разных файлах на диске.
Чтобы начать ввод, необходимо создать новый или выбрать существующий файл данных.
Откроется окно ввода данных.
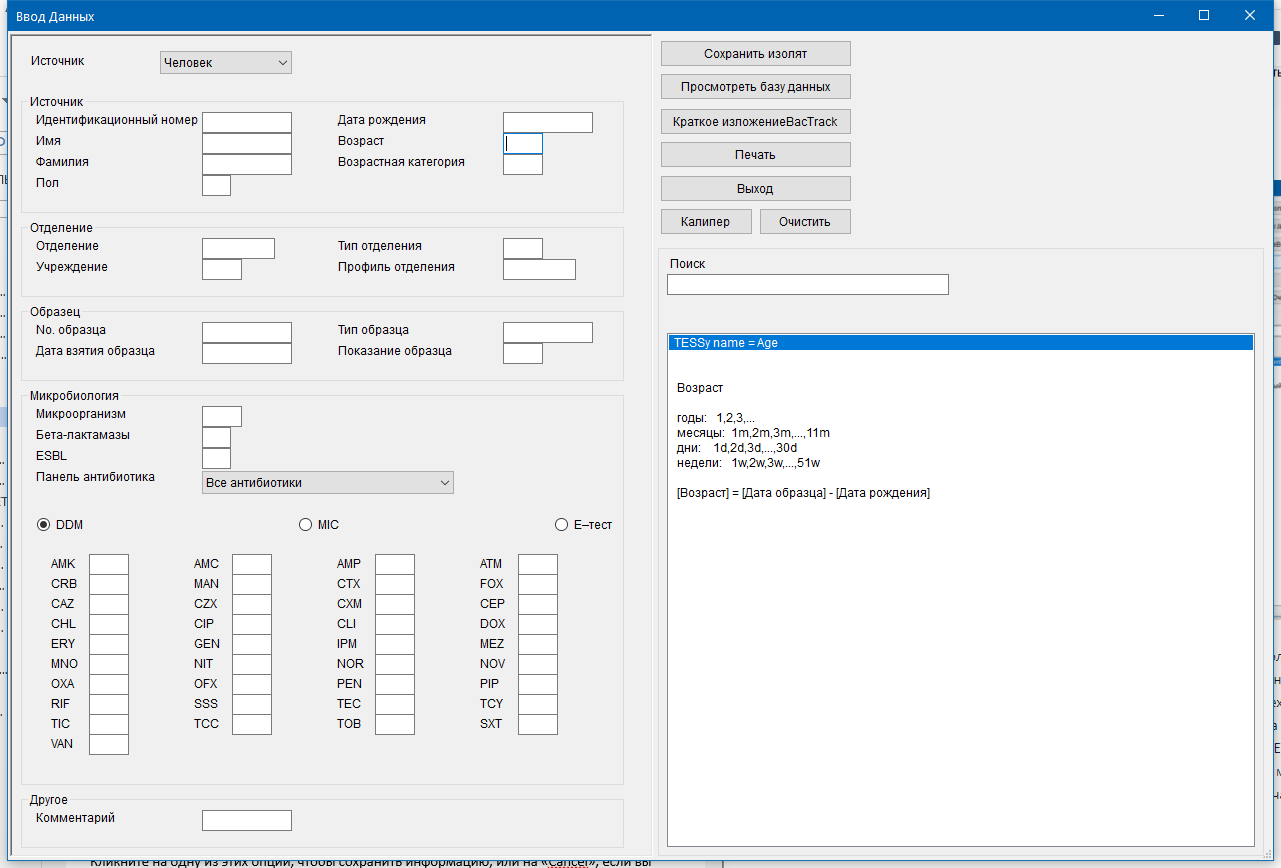
Данные необходимо вводить в поля, расположенные в левой половине экрана. Когда курсор переключается на то или иное поле для ввода данных, в правом нижнем углу экрана появляются краткие инструкции и рекомендуемые коды данных для этого поля. Переходить по полям можно с помощью кнопок Enter, Tab, стрелками. После ввода даты и перехода к следующему полю пользователю следует убедиться в том, что дата была прочитана верно: WHONET автоматически преобразует числовое обозначение месяца в название месяца. При введении даты год может быть введен как в виде двузначной, так и в виде четырехзначной цифры. Числа, обозначающие день, месяц и год, должны быть разделены косой чертой /, дефисом или пробелом.
После того как будут введены все данные об изоляте, нужно нажать кнопку Сохранить изолят (или комбинация клавиш Alt+С). WHONET предложит три варианта сохранения записи:
- сохранить изолят,
- сохранить и продолжить работу с тем же изолятом,
- сохранить и продолжить работу с тем же пациентом.
Выбор одной из этих опций сохраняет информацию и очищает экран Ввод данных для ввода данных следующего изолята (полностью или частично).
6.3.3 Просмотр данных
При открытии файла данных всегда по умолчанию открывается форма для ввода нового образца. Чтобы посмотреть информацию по всем образцам в файле, нужно нажать на этой форме кнопку Просмотреть базу данных.
По данной таблице можно осуществлять поиск, удаление записей, вывод результатов на печать и даже осуществлять быстрое редактирование для некоторых полей. Чтобы перейти к полноценной форме редактирования, требуется выделить интересующую строку и нажать кнопку Редактировать.
6.3.4 Объединение данных
К преимуществам WHONET можно отнести концепцию разделения конфигурации лаборатории и ее файла данных. Благодаря этому на основе одной конфигурации можно создавать несколько файлов данных с одинаковой структурой и «протоколом» хранения для разных лабораторий, а затем объединять и агрегировать эти данные. На этом принципе построена работа в таких междунароных системах эпиднадзора за устойчивостью к АМП, как GLASS и CAESAR.
Порядок действий для объединения файлов WHONET, полученных от разных центров/лабораторий/стационаров:
- Выбрать лабораторию с требуемой конфигурацией, нажать кнопку Открыть лабораторию.
- В меню Ввод данных выбрать пункт Объединять, экспортировать или шифровать файлы данных.
- В появившемся окне выбрать файлы, которые требуется объединить с помощью кнопки Файлы данных, указать, куда сохранить объединенный файл данных. Если данный файл предполагается передавать в сторонние центральные лаборатории или другие организации, необходимо отметить галочку Зашифровать информацию о пациентах. В этом случае персональные данные в объединенном файле будут удалены. Для начала процесса объединения необходимо нажать кнопку Совместить.
6.3.5 Экспорт данных
Одно из главных преимуществ WHONET состоит в возможности обмениваться стандартизированными данными с другими лабораториями, например, в национальной сети эпиднадзора.
Однако текущая версия WHONET не имеет достаточно гибких возможностей для углубленного анализа и представления данных, а также существенно ограничена при использовании мультидисциплинарной командой с целью мониторинга АР. Получаемые отчеты лишены интерактивности и удобных возможностей быстрого переключения параметров и сравнения полученных результатов. Для этого гораздо удобнее использовать AMRcloud. Существует возможность подготовить данные, хранящиеся в датафайлах программы WHONET, к загрузке в AMRcloud.
В общем случае алгоритм следующий:
- Открыть в программе лабораторию, из которой нужно получить данные, выбрать в меню Анализ данных пункт Анализ данных.
- Выбрать Тип анализа – Список изолята и сводка. В разделе Формат отчетов выбрать пункт Результат загрузки.
- Выбрать Микроорганизмы, которые требуется включить в анализ.
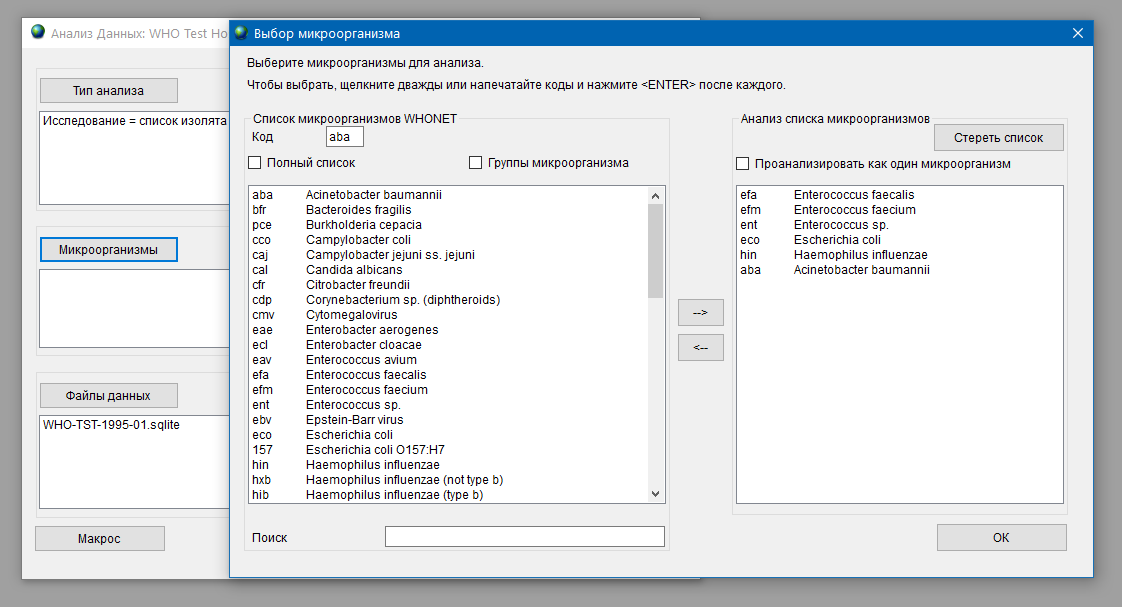
- Выбрать файл данных.
- Установить вывод результата в формате Текст (WHONET).
- Указать, куда сохранить файл экспорта.
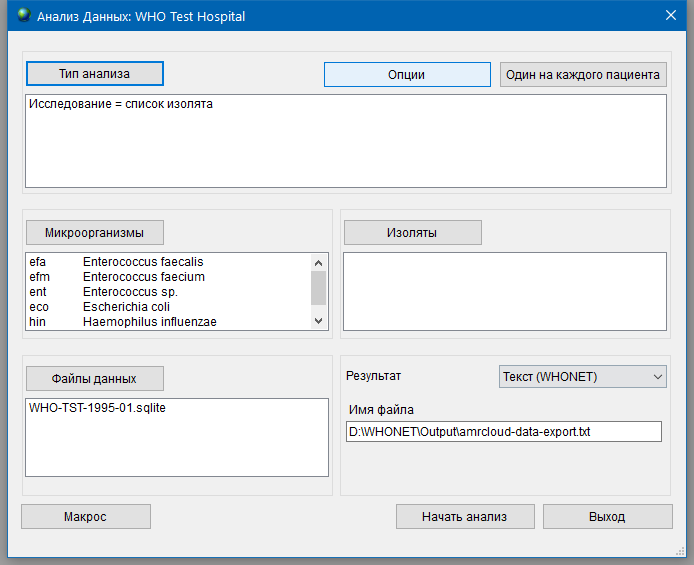
Нажать кнопку Начать анализ. Программа сформирует текстовый файл, который необходимо загрузить в онлайн-конвертер, доступный по ссылке https://public.amrcloud.net/whonet. После прохождения всех шагов работы конвертера появится файл, пригодный для загрузки в AMRcloud.
Более подробное описание процедуры экспорта и работы с конвертером можно найти в официальном руководстве по ссылке https://amrcloud.net/ru/tutorials/whonet-converter.
6.4 Работа с BACLINK
Программа BacLink входит в состав программы WHONET и позволяет преобразовывать табличные файлы различных форматов, чтобы затем работать с ними уже в рамках программы WHONET [69] . Если решено отказаться от накопления данных в Excel и начать работать в WHONET, то программа BacLink максимально упростит этот переход и перенесет накопленные за долгое время данные [72] .
Официальное руководство по работе с программой доступно на сайте https://whonet.org/documentation.html , а также в самой программе.
В данном руководстве представлен алгоритм работы с программой на примере табличного файла с результатами тестирования.
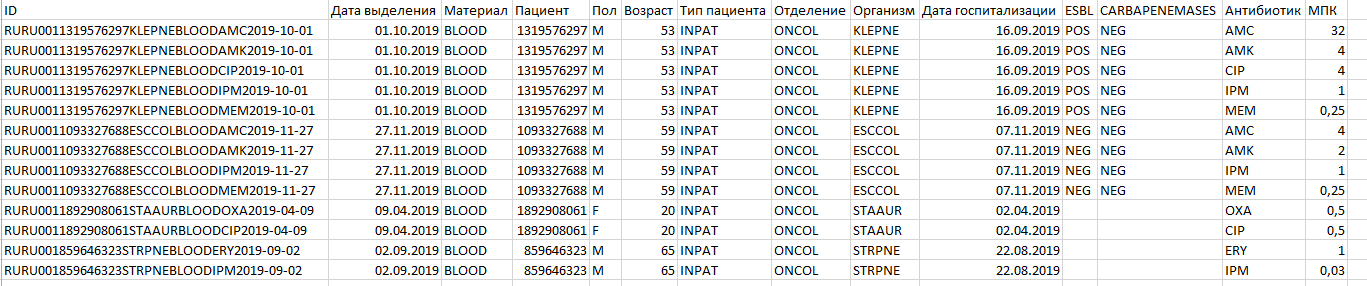
Данный файл не удовлетворяет условию одна строка – один изолят. В каждой строке содержатся данные по чувствительности к отдельному антибиотику, при этом данные самого изолята повторяются. Мало того, что такой способ хранения неэффективно расходует место на диске, это затрудняет анализ полученных данных. Требуется преобразовать такой файл с помощью программы BacLink. Лучше всего программа работает с текстовыми файлами, поэтому, если данные хранятся в форматах Excel (.xls, .xlsx), необходимо их сохранение в формат CSV. Рекомендуется использовать латинские символы в именах полей.
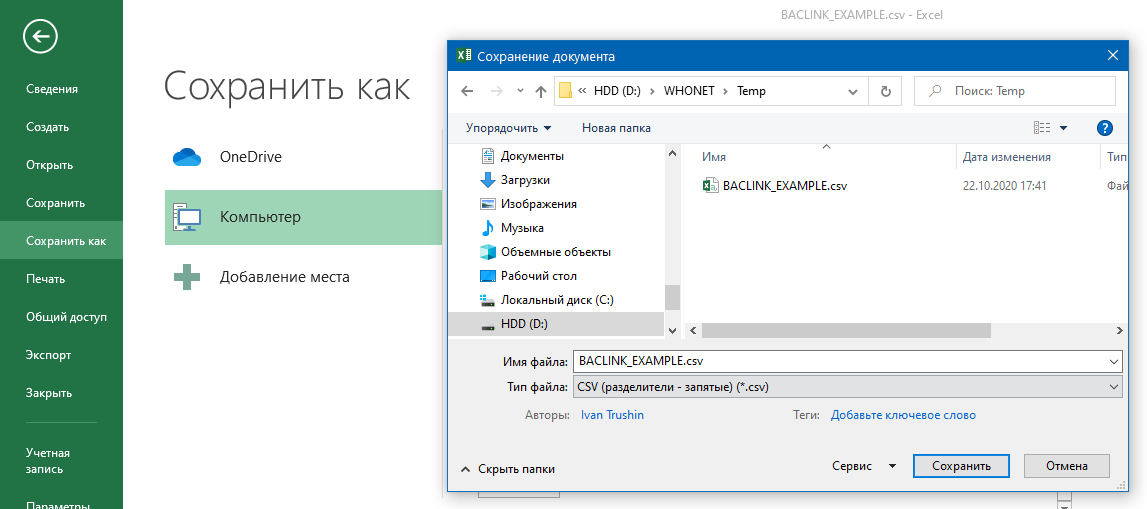
Окно программы BacLink после запуска показано на рисунке.
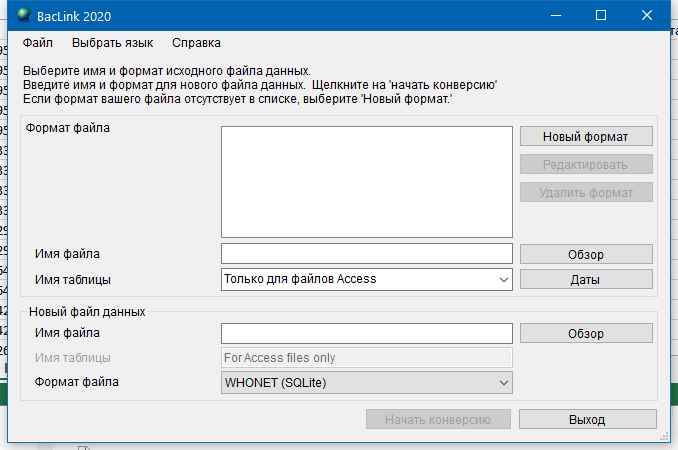
Необходимо определить параметры файла с помощью кнопки Новый формат. Регистрационные данные лаборатории можно заполнить произвольно.
Затем нужно сопоставить поля файла с внутренними полями WHONET с помощью кнопки Структура файла.
Файл сохранен в формате CSV, значит, это текстовый файл с разбиением по полям, при этом разделителем полей служит символ точки с запятой ( ; ) и первая строка содержит заголовки полей. Если файл получен с помощью Excel, то по умолчанию используется кодировка Windows (ANSI).
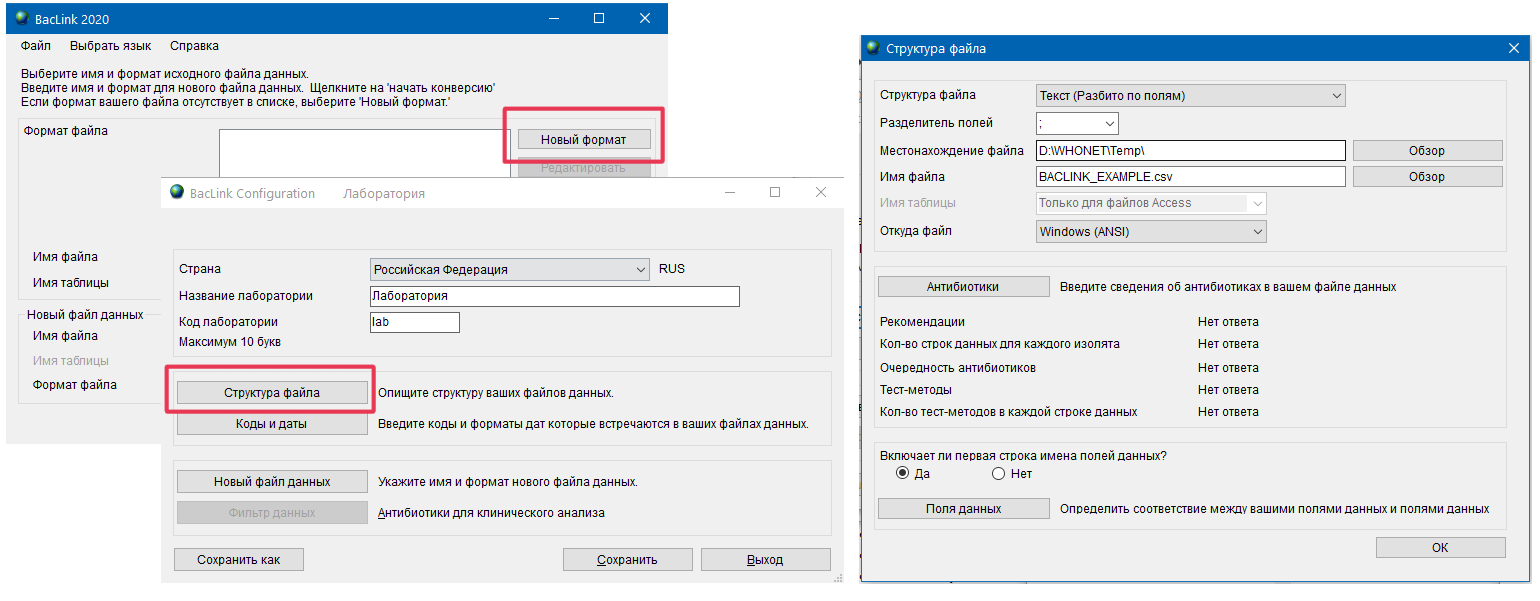
Необходимо заполнить сведения об антибиотиках – нажать кнопку ****Антибиотики. В файле на изолят приходится несколько строк, при этом каждый изолят может тестироваться разными антибиотиками. Результаты чувствительности представлены МПК. Интерпретировать полученные результаты необходимо согласно EUCAST.
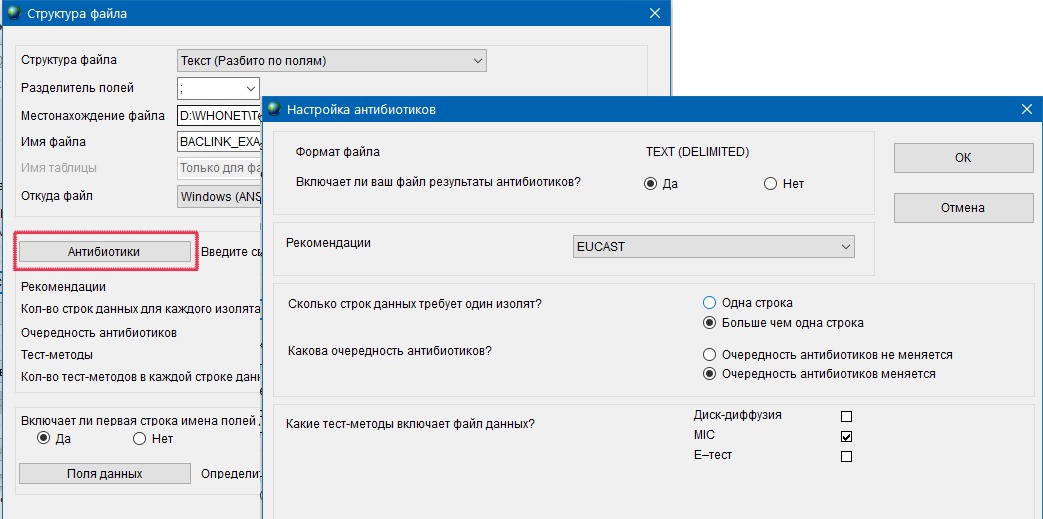
Один из самых важных этапов – сопоставление имеющихся полей данных с полями данных программы WHONET – кнопка Поля данных. Сначала необходимо получить список полей файла: выбрать файл с помощью кнопки Выбрать образцовый файл данных. В результате в правой части окна появятся заголовки столбцов импортируемой таблицы с примерами данных. Для сопоставления необходимо последовательно выбирать поле из левой колонки, затем из правой и нажимать кнопку [=], чтобы создать связь.
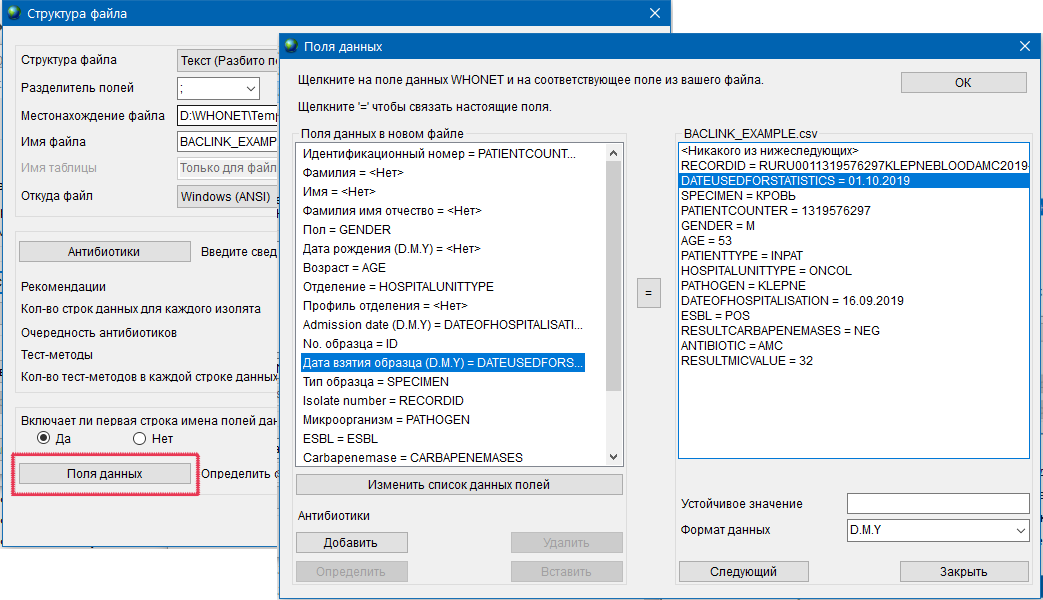
В файле используется формат даты ДД.ММ.ГГГГ, поэтому нужно установить формат данных D.M.Y. Часть полей отсутствует в списке Поля данных в новом файле, чтобы добавить новые поля, требуется нажать кнопку Изменить список данных полей и выбрать по очереди поля ESBL, CARBAPENEMASE и ADMISSION DATE, отметить галочку Демонстрировать в поле списка и нажать кнопку ОК.
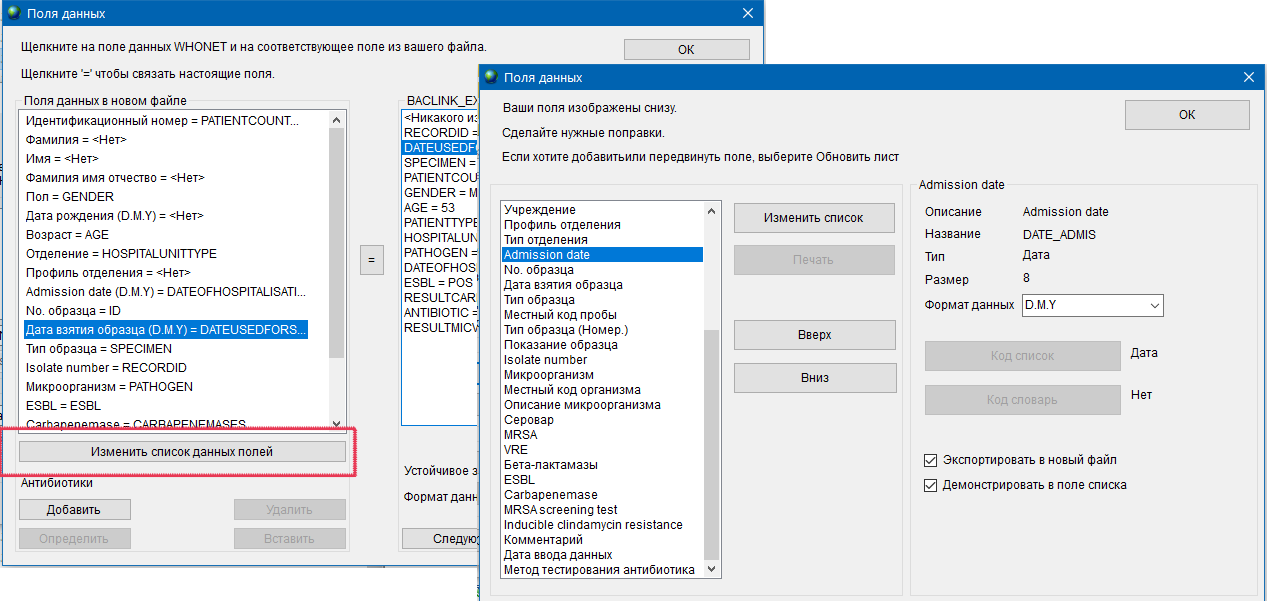
По окончании сопоставления всех полей нужно нажать кнопку ОК. На этом определение структуры файла окончено, можно закрыть окно Структура файла с помощью кнопки ОК. Во время конверсии файлов программа BacLink пытается сопоставить значения полей файла со встроенными справочниками программы WHONET. Если нажать кнопку Коды и даты в окне конфигурации лаборатории, можно увидеть данные настройки, а также пути сохранения файлов с правилами сопоставлений. На данном этапе можно не заполнять данные настройки целиком, а лишь определить формат дат и назначить коды для определения пола. Также стоит обратить внимание на место сохранения файлов-словарей и при необходимости изменить его. Это может быть полезным в дальнейшем.
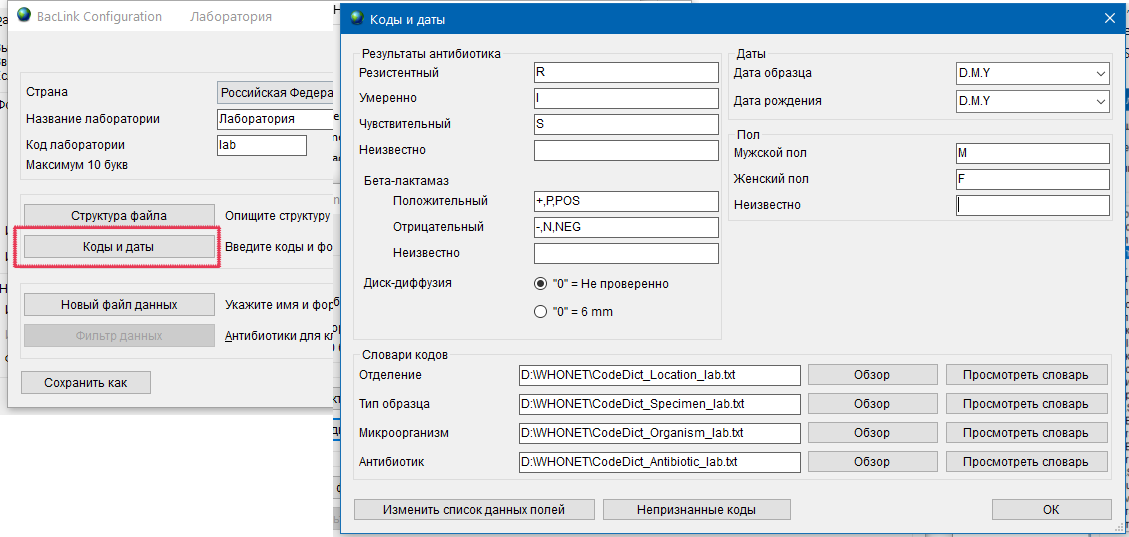
Далее необходимо определить, куда будет сохранен преобразованный файл – для этого нужно нажать кнопку Новый файл данных и указать путь.
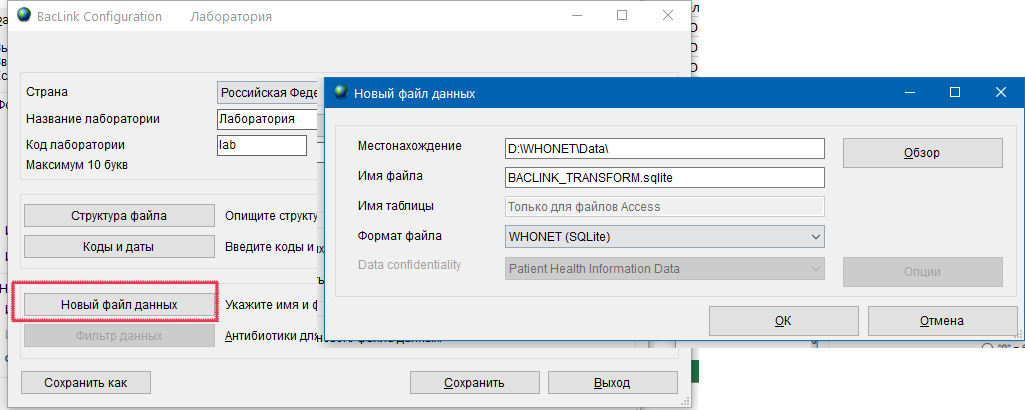
C помощью кнопки Сохранить необходимо сохранить конфигурацию лаборатории и структуры файла. Эти настройки можно использовать повторно, если потребуется преобразовать еще несколько файлов с такой же структурой. В таком случае не придется заново задавать все параметры, достаточно будет указать файл конфигурации. После этого можно нажать кнопку Выход.
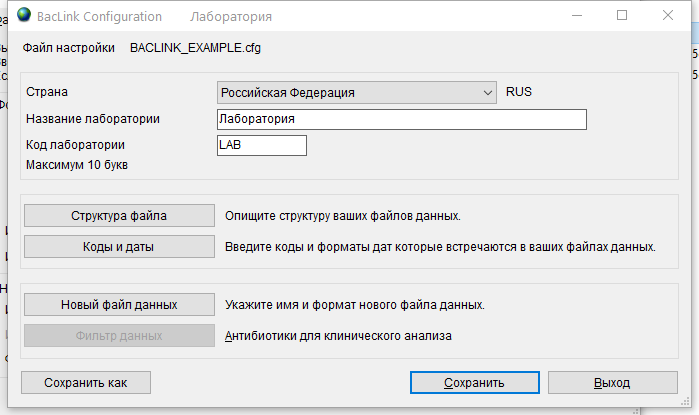
Теперь все готово к преобразованию и можно нажать кнопку Начать конверсию.
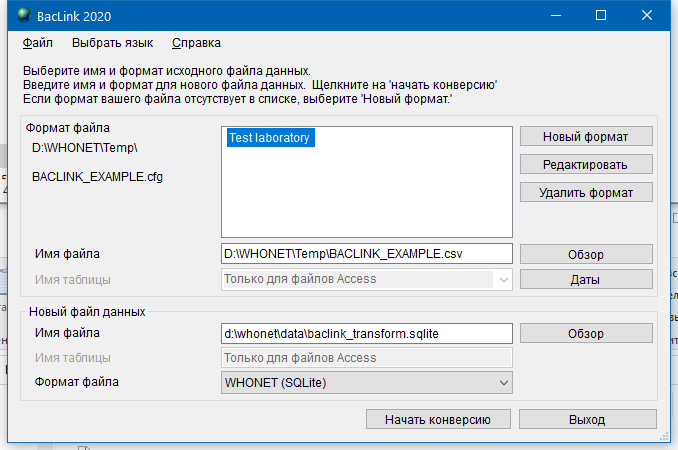
В процессе работы программа покажет несколько изолятов для демонстрации преобразования и по окончании выведет отчет о количестве обработанных изолятов. Так как при определении структуры файла не заполнялись словари замен, будут выведены коды, которые BacLink не смог интерпретировать. Их необходимо интерпретировать вручную.
Окно Непризнанные коды показывает, какие коды для каких полей остались нераспознанными. Необходимо последовательно выбрать каждое поле данных, нажать кнопку Определить коды и указать правильную интерпретацию.
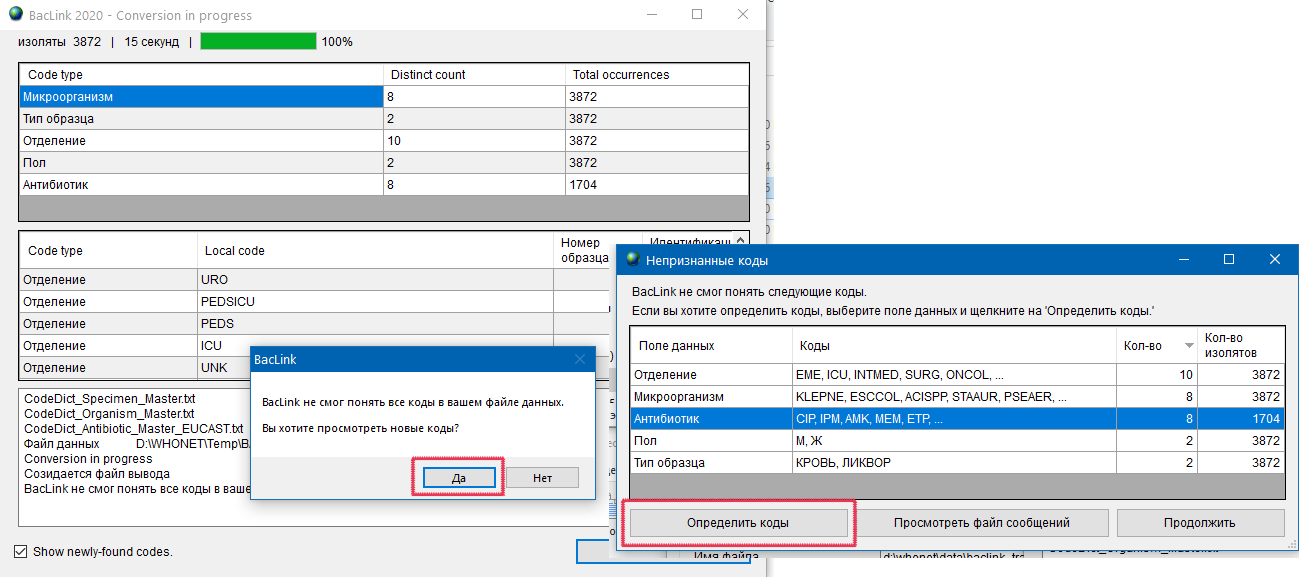
Для примера можно взять поле Антибиотик: выделить поле данных Антибиотик и нажать кнопку Определить код.
В нижней части окна предлагаются возможные варианты замен. Для антибиотиков рекомендуется выбрать значения с пометкой EUCAST. После выбора замены необходимо нажать кнопку ОК. По мере сопоставления замен они будут исчезать из списка.
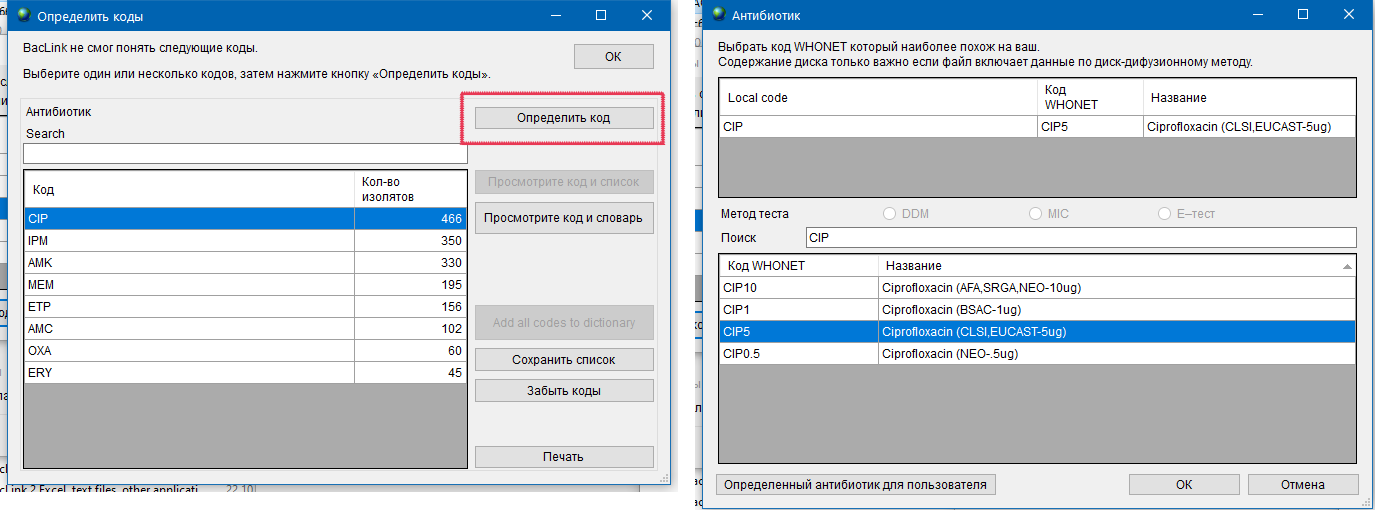
Возможна ситуация, когда программа сама не сможет предложить замену, например, при определении кодов организмов. В таком случае необходимо ввести в поле Поиск нужную замену и тем самым указать программе интерпретацию.
После определения всех кодов окно Непризнанные коды должно остаться пустым. Теперь можно нажать кнопку Продолжить.
Далее нужно повторить процедуру конвертации, но уже с определенными заменами для кодов, так как при первоначальной конверсии поля с нераспознанными кодами оставались пустыми. При дальнейшей конвертации подобных файлов коды заново определять не потребуется, так как они сохраняются в словарях. Для повторного запуска процедуры конвертации нужно нажать кнопку Начать конверсию повторно и согласиться на перезапись файла данных.
Теперь созданный файл можно использовать в программе WHONET и работать с ним как с обычным датафайлом лаборатории. Для этого нужно запустить программу и выбрать лабораторию.
Если ее нет, можно создать новую лабораторию на основе сконвертированного файла. Для этого нужно закрыть окно выбора лаборатории, выбрать в меню Файл пункт Создать лабораторию с файла данных. В открывшемся окне необходимо заполнить данные лаборатории и выбрать сконвертированный файл данных.
На основании этих данных программа WHONET создаст новую конфигурацию лаборатории, импортирует существующие настройки, добавит антибиотики, отделения и поля из датафайла и предложит дополнить информацию. Для лаборатории можно указать, с какими антибиотиками она работает, из каких отделений могут поступать пациенты, какие поля данных стоит заводить на изолят. Более подробно о том, как сконфигурировать лабораторию, написано в официальном руководстве программы WHONET, а также краткий обзор дается в приложении (6.3. Работа с WHONET](https://monitoring.amrcloud.net/docs/06/03/). Чтобы продолжить работу с конвертированным датафайлом, достаточно нажать кнопку Сохранить.
Для начала ввода необходимо выбрать в меню Ввод данных пункт Открыть файл данных и указать файл, который был получен после конвертации программой BacLink.
Откроется окно ввода новых данных. Для просмотра списка изолятов нужно нажать кнопку Посмотреть базу данных. Откроется таблица со всеми изолятами и результатами определения чувствительности.
6.5 Стандартные эпидемиологические определения случая инфекции
Определения случая инфекции адаптированы из следующих источников:
- Pan American Health Organization. Epidemiological Surveillance of Healthcare-Associated Infections [73] ;
- CDC. Procedure-associated Module. Surgical Site Infection (SSI) Event [74] ;
- IDSA. Clinical Practice Guideline for the Management of Asymptomatic Bacteriuria: 2019 [75] .
6.5.1 Пневмония
Пневмония должна удовлетворять одному из следующих критериев:
Критерий 1:
- один и более из следующих признаков, сохраняющихся на двух или более последовательных рентгеновских снимках грудной клетки:
- новый или прогрессирующий и стойкий инфильтрат;
- консолидация;
- кавитация.
Примечание. У пациентов без основной легочной патологии и/или патологии сердца (например, в отсутствии респираторного дистресс-синдрома, бронхолегочной дисплазии, отека легких, хронической обструктивной болезни легких) допускается однократное рентгенологическое исследование.
И
- один и более из следующих признаков:
- температура тела ≥38 °С или ≤36 °С без других известных причин;
- лейкоциты крови >12 × 109/мл или <4 × 109/мл, или незрелых форм >10%;
- для взрослых >70 лет измененное психическое состояние без какой-либо другой установленной причины.
И
- два и более из следующих признаков:
- вновь появившаяся гнойная мокрота, или изменение характера мокроты, или усиление дыхательной секреции, или повышенная потребность в аспирации отделяемого трахебронхиального дерева;
- появление или ухудшение кашля и/или одышки и/или тахипноэ;
- наличие хрипов или бронхиального дыхания;
- ухудшение показателей газообмена [например, десатурация O2 (например, PaO2 /FiO2 <240), повышенная потребность в кислороде или потребность в аппарате ИВЛ].
Критерий 2:
- один и более из следующих признаков, сохраняющихся на двух или более последовательных рентгеновских снимках грудной клетки:
- новый или прогрессирующий и стойкий инфильтрат;
- консолидация;
- кавитация.
Примечание. У пациентов без основной легочной патологии и/или патологии сердца (например, в отсутствии респираторного дистресссиндрома, бронхолегочной дисплазии, отека легких, хронической обструктивной болезни легких) допускается однократное рентгенологическое исследование.
И
- один и более из следующих признаков:
- температура тела ≥38 °С или ≤36 °С без других известных причин;
- лейкоциты крови >12 × 109/мл или <4 × 109/мл, или незрелых форм >10%;
- для взрослых >70 лет измененное психическое состояние без какой-либо другой установленной причины.
И
- один и более из следующих признаков:
- вновь появившаяся гнойная мокрота, или изменение характера мокроты, или усиление дыхательной секреции, или повышенная потребность в аспирации отделяемого трахебронхиального дерева;
- появление или ухудшение кашля и/или одышки и/или тахипноэ;
- наличие хрипов или бронхиального дыхания;
- ухудшение показателей газообмена [например, десатурация O2 (например, PaO2 /FiO2 <240), повышенная потребность в кислороде или потребность в аппарате ИВЛ].
И
- один и более из следующих признаков:
- положительная гемокультура (выявление микроорганизмов в культуре крови), не связанная с другим источником инфекции;
- выявление микроорганизмов в культуре плевральной жидкости;
- положительный количественный посев материала из нижних дыхательных путей из образцов, полученных с минимальной вероятностью контаминации (например, с помощью бронхоальвеолярного лаважа (>104 КОЕ/мл), или метода защищенных щеток (>103 КОЕ/мл), или аспирате из трахеи (>103 КОЕ/мл);
- ≥5% клеток, полученных в бронхоальвеолярном лаваже, содержат внутриклеточно расположенные бактерии при прямом микроскопическом исследовании (например, при окрашивании по Граму);
- один и более из следующих признаков:
- образование абсцесса или очаги уплотнения с интенсивным накоплением полиморфноядерных лейкоцитов в бронхиолах и альвеолах;
- положительный количественный посев паренхимы легкого;
- свидетельства инвазии паренхимы легкого грибковыми гифами или псевдогифами.
6.5.2 Лабораторно подтвержденная инфекция кровотока
Лабораторно подтвержденная инфекция кровотока должна удовлетворять одному из следующих критериев:
Критерий 1:
- положительная гемокультура (выявление микроорганизмов в культуре крови) в одном или нескольких образцах (не относится к контаминирующим кожные покровы микроорганизмам, см. Критерий 2).
И
- выделенный из гемокультуры микроорганизм не связан с другим источником инфекции.
Критерий 2:
- один и более из следующих признаков без другой установленной причины:
- лихорадка (>38 °C);
- озноб;
- гипотензия.
И
- обнаружение условно-патогенного микроорганизма из числа обычно наблюдаемых на поверхности кожного покрова (наример дифтероиды [Corynebacterium spp.], Bacillus [за исключением B. anthracis] spp., Propionibacterium spp., коагулазонегативные стафилококки [включая S. epidermidis], зеленящие Streptococci, Aerococcus spp., Micrococcus spp.) в двух или более гемокультурах, взятых в разные сроки;
И / ИЛИ
- обнаружение микроорганизма из числа обычно наблюдаемых на поверхности кожного покрова хотя бы в одном посеве крови, взятой у пациента с внутривенным катетером и назначение врачом по этому поводу антимикробной терапии.
6.5.3 Инфекция мочевыводящих путей
Клинически выраженная инфекция мочевыводящих путей должна удовлетворять одному из следующих критериев:
Критерий 1:
- один и более из следующих признаков без другой установленной причины:
- лихорадка (≥38 °С);
- резкие позывы к мочеиспусканию;
- учащенное мочеиспускание;
- дизурия или надлобковая болезненность.
И
- положительный количественный посев мочи (>105 КОЕ/мл мочи).
Критерий 2:
- два и более из следующих признаков без другой установленной причины:
- лихорадка (≥38 °С);
- резкие позывы к мочеиспусканию;
- учащенное мочеиспускание;
- дизурия или надлобковая болезненность.
И
- один и более из следующих признаков:
- положительный тест на лейкоцитарную эстеразу или нитрат;
- пиурия (>10 лейкоцитов/мл мочи или >3 лейкоцитов в поле зрения микроскопии мочи высокого разрешения);
- обнаружение микроорганизмов при окраске по Граму нецентрифугированной мочи;
- выделение одного уропатогенного микроорганизма в количестве ≤105 КОЕ/мл у пациента, получающего антимикробную терапию по поводу уроинфекции;
- двукратно микробиологически подтвержденная бактериурия (>105 КОЕ/мл мочи).
6.5.4 Бессимптомная бактериурия
Бессимптомная бактериурия должна удовлетворять следующим критериям:
Критерий:
- микробиологически подтвержденная бактериурия (≥105 КОЕ/ мл мочи) вне зависимости от наличия пиурии;
И
- температура тела <37,5 °С, отсутствие резких позывов к мочеиспусканию, учащенного мочеиспускания, дизурии, надлобковой болезненности.
6.5.5 Инфекция области хирургического вмешательства
Поверхностная инфекция разреза возникает не позднее 30 дней после операции и вовлекает только кожу и подкожные ткани в области разреза.
Критерий:
как минимум одно из перечисленного- гнойное отделяемое из поверхностного разреза;
- выделение микроорганизмов из жидкости или ткани, полученной асептически пункцией области поверхностного разреза или из мазка из раны при наличии микроскопических признаков гнойного воспаления;
- имеется не менее двух из следующих симптомов:
- боль или болезненность;
- ограниченный отек;
- гиперемия;
- местное повышение температуры.
- назначение лечащим врачом антимикробной терапии по поводу нагноения послеоперационной раны.
Глубокая инфекция области хирургического вмешательства возникает не позднее 30 дней после операции при отсутствии имплантата или не позднее одного года при наличии имплантата в месте операции и вовлекает глубокие мягкие ткани (например, фасциальный и мышечный слой) в области разреза.
Критерий:
как минимум одно из перечисленного- гнойное отделяемое из глубины разреза в месте данного хирургического вмешательства, но не из органа/полости;
- выделение микроорганизмов из жидкости или ткани, полученное асептически пункцией области глубокого разреза или из мазка из глубины раны при наличии микроскопических признаков гнойного воспаления;
- спонтанное расхождение краев раны или намеренное ее открытие хирургом, когда у пациента имеются следующие признаки и симптомы:
- температура тела >37,5 °C;
- локализованная боль и/или отек.
- при непосредственном осмотре, во время повторной операции, при гистологическом или рентгенологическом исследовании обнаружен абсцесс или иные признаки инфекции в области глубоких слоев операционной раны;
- назначение лечащим врачом антимикробной терапии по поводу абсцесса, флегмоны в месте операции и пр.
Инфекция полости/органа возникает не позднее 30 дней после операции при отсутствии имплантата или не позднее одного года при наличии имплантата в месте операции, с вовлечением органа или полости.
Критерий:
как минимум одно из перечисленного- гнойное отделяемое из дренажа, установленного в органе/полости через специальный разрез;
- выделение микроорганизмов из жидкости или ткани, полученной асептически из органа/полости и лихорадка;
- при непосредственном осмотре, во время повторной операции, при гистологическом или рентгенологическом исследовании обнаружен абсцесс или иные признаки инфекции, вовлекающие орган/полость;
- назначение лечащим врачом антимикробной терапии по поводу перитонита, остеомиелита, эмпиемы плевры, пиелонефрита, медиастенита, эндометрита и другие, возникшие после операции на соответствующем органе.
Литература
1. Tillotson G.S., Zinner S.H. Burden of antimicrobial resistance in an era of decreasing susceptibility. Expert Rev Anti Infect Ther. 2017;15(7):663–676.
https://www.tandfonline.com/doi/abs/10.1080/14787210.2017.1337508?journalCode=ierz20
2. Козлов Р.С., Голуб А.В. Остановить темпы роста антибиотикорезистентности микроорганизмов сегодня – дать шанс на выживание человечества завтра. Клиническая микробиология и антимикробная химиотерапия. 2019;21(4):310–315.
https://cmac-journal.ru/publication/2019/4/cmac-2019-t21-n4-p310/
3. Всемирная организация здравоохранения. Глобальный план действий по борьбе с устойчивостью к противомикробным препаратам. Geneva, Switzerland; 2016.
https://apps.who.int/iris/bitstream/handle/10665/254884/9789244509760-rus.pdf?sequence=1
4. Распоряжение правительства Российской Федерации от 25 Сентября 2017 г. № 2045-р «Стратегия предупреждения распространения антимикробной резистентности в Российской Федерации на период до 2030 года».
http://static.government.ru/media/files/onJ3GY3ObDGqLDvrED7AhpLF3ywRRFpp.pdf
5. Michael C.A., Dominey-Howes D., Labbate M. The antimicrobial resistance crisis: causes, consequences, and management. Front public Heal. 2014;2:145.
https://www.frontiersin.org/articles/10.3389/fpubh.2014.00145/full
6. Tacconelli E., Sifakis F., Harbarth S., Schrijver R., van Mourik M., Voss A., et al. Surveillance for control of antimicrobial resistance. Lancet Infect Dis. 2018;18(3):e99–106.
https://www.thelancet.com/journals/laninf/article/PIIS1473-3099(17)30485-1/fulltext
7. Stratchounski L.S., Dekhnich A.V., Kretchikov V.A., Edelstain I.A., Narezkina A.D., Afinogenov G.E., et al. Antimicrobial resistance of nosocomial strains of Staphylococcus aureus in Russia: results of a prospective study. J Chemother. 2005;17(1):54–60.
https://www.tandfonline.com/doi/abs/10.1179/joc.2005.17.1.54
8. Stratchounski L.S., Kozlov R.S., Appelbaum P.C., Kretchikova O.I., KosowskaShick K. Antimicrobial resistance of nasopharyngeal pneumococci from children from day-care centres and orphanages in Russia: results of a unique prospective multicentre study. Clin Microbiol Infect. 2006;12(9):853–866.
https://www.clinicalmicrobiologyandinfection.com/article/S1198-743X(14)64303-2/fulltext
9. Stratchounski L.S., Kretchikova O.I., Kozlov R.S., Reshedko G.K., Stetsiouk O.U., Tarasova G.D., et al. Antimicrobial resistance of Streptococcus pneumoniae isolated from healthy children in day-care centers: results of a multicenter study in Russia. Pediatr Infect Dis J. 2000;19(3):196–200.
https://journals.lww.com/pidj/Abstract/2000/03000/Antimicrobial_resistance_ofStreptococcus.4.aspx
10. Кузьменков А.Ю., Трушин И.В., Авраменко А.А., Эйдельштейн М.В., Дехнич А.В., Козлов Р.С. AMRmap: Интернет-платформа мониторинга антибиотикорезистентности. Клиническая микробиология и антимикробная химиотерапия. 2017;19(2):84–90.
https://cmac-journal.ru/publication/2017/2/cmac-2017-t19-n2-p084/
11. Виноградова А.Г., Кузьменков А.Ю. Организация данных как основа локального мониторинга антибиотикорезистентности. Клиническая микробиология и антимикробная химиотерапия. 2020;22(2):137–141.
https://cmac-journal.ru/publication/2020/2/cmac-2020-t22-n2-p137/
12. Кузьменков А.Ю., Виноградова А.Г., Трушин И.В., Авраменко А.А., Эйдельштейн М.В., Дехнич А.В., и соавт. AMRcloud: новая парадигма мониторинга антибиотикорезистентности. Клиническая микробиология и антимикробная химиотерапия. 2019;21(2):119–124.
https://cmac-journal.ru/publication/2019/2/cmac-2019-t21-n2-p119/
13. Lushniak B.D. Antibiotic resistance: a public health crisis. Public Health Rep. 2014;129(4):314–316.
https://journals.sagepub.com/doi/10.1177/003335491412900402
14. Ferri M., Ranucci E., Romagnoli P., Giaccone V. Antimicrobial resistance: a global emerging threat to public health systems. Crit Rev Food Sci Nutr. 2017;57(13):2857–2876.
https://www.tandfonline.com/doi/abs/10.1080/10408398.2015.1077192?journalCode=bfsn20
15. Núñez-Núñez M., Navarro M.D., Palomo V., Rajendran N.B., del Toro M.D., Voss A., et al. The methodology of surveillance for antimicrobial resistance and healthcare-associated infections in Europe (SUSPIRE): a systematic review of publicly available information. Clinical Microbiology and Infection. 2018;24(2):105–109.
https://www.clinicalmicrobiologyandinfection.com/article/S1198-743X(17)30385-3/fulltext
16. Masterton R. The Importance and Future of Antimicrobial Surveillance Studies. Clin Infect Dis. 2008;47(S1):S21–31.
https://academic.oup.com/cid/article/47/Supplement_1/S21/304789
17. Coulehan M.B., Wells J.F. Guidelines for responsible data management in scientific research. 46 p.
https://ori.hhs.gov/guidelines-responsible-datamanagementscientific-research
18. Кузьменков А.Ю., Виноградова А.Г. Мониторинг антибиотикорезистентности: обзор информационных ресурсов. Бюллетень сибирской медицины. 2020;19(2):163–170.
https://bulletin.tomsk.ru/jour/article/view/2873/1753
19. Perez F., Villegas M.V. The role of surveillance systems in confronting the global crisis of antibiotic-resistant bacteria. Curr Opin Infect Dis. 2015;28(4): 375–383.
20. Яковлев С.В., Брико Н.И., Сидоренко С.В., Проценко Д.Н. Программа СКАТ (Стратегия Контроля Антимикробной Терапии) при оказании стационарной медицинской помощи. 2018; 156 с. Доступно по адресу: http://nasci. ru/?id=2880
21. CDDEP. ResistanceMap: Antibiotic Resistance. 2020.
https://resistancemap.cddep.org/AntibioticResistance.php
22. CDC. Surveillance Reports.
http://www.cdc.gov/abcs/reports-findings/surv-reports.html
23. The British Society for Antimicrobial Chemotherapy. Resistance Surveillance Project.
24. Edwards J.R., Peterson K.D., Mu Y., Banerjee S., Allen-Bridson K., Morrell G., et al. National Healthcare Safety Network (NHSN) report: Data summary for 2006 through 2008, issued December 2009. Am J Infect Control. 2009;37(10):783–805.
https://www.ajicjournal.org/article/S0196-6553(09)00839-6/fulltext
25. Antimicrobial Resistance Standing Committee. National Surveillance and Reporting of Antimicrobial Resistance and Antibiotic Usage for Human Health in Australia (Project AMRAU). 2013; 152 p.
26. Fuhrmeister A.S., Jones R.N. The Importance of antimicrobial resistance monitoring worldwide and the origins of SENTRY antimicrobial surveillance program. Open Forum Infect Dis. 2019;6(Suppl.1):S1–4.
https://academic.oup.com/ofid/article/6/Supplement_1/S1/5381628
27. European Centre for Disease Prevention and Control. Surveillance of antimicrobial resistance in Europe 2018.
28. Виноградова А.Г., Кузьменков А.Ю. Практическое применение AMRmap: элементы подхода «от общего к частному» на примере Klebsiella pneumoniae. Клиническая микробиология и антимикробная химиотерапия. 2019;21(2):181–186.
https://cmac-journal.ru/publication/2019/2/cmac-2019-t21-n2-p181/
29. ГОСТ Р 53079.2-2008 Технологии лабораторные клинические. Обеспечение качества клинических лабораторных исследований. Часть 2. Руководство по управлению качеством в клинико-диагностической лаборатории. Типовая модель. 2010.
http://docs.cntd.ru/document/1200073591
30. МУ 4.2.2039-05 Техника сбора и транспортирования биоматериалов в микробиологические лаборатории. 2006; 23 с.
http://docs.cntd.ru/document/1200044664
31. Культуры крови (гемокультуры). Ключевое исследование для диагностики инфекций кровотока.
32. Решедько Г.К. Определение чувствительности к антибиотикам: методы, результаты, оценка.
http://www.antibiotic.ru/rus/all/articles/absens.shtml
33. Тапальский Д.В., Бильский И.А. Определение чувствительности к антибиотикам методом микроразведений в бульоне: модификация, доступная для всех. Клиническая микробиология и антимикробная химиотерапия. 2018;20(1):62–67.
https://cmac-journal.ru/publication/2018/1/cmac-2018-t20-n1-p062/
34. Шамина О.В., Крыжановская О.А., Лазарева А.В., Поликарпова С.В., Карасева О.В., Чеботарь И.В. и соавт. Сравнение методов определения устойчивости к колистину у карбапенемрезистентных штаммов Klebsiella pneumoniae. Клиническая лабораторная диагностика. 2018;63(10):646–650.
35. ISO 20776-1:2019. Susceptibility testing of infectious agents and evaluation of performance of antimicrobial susceptibility test devices – Part 1: Broth micro-dilution reference method for testing the in vitro activity of antimicrobial agents against rapidly growing aerobic bacteria involved in infectious diseases. 2019.
https://www.iso.org/standard/70464.html
36. Приказ Федерального агентства по техническому регулированию и метрологии Российской Федерации от 23 ноября 2010 г. N 499-ст.
http://docs.cntd.ru/document/902264310
37. Jorgensen J.H., Ferraro M.J. Antimicrobial Susceptibility Testing: A review of general principles and contemporary practices. Clin Infect Dis. 2009;49(11):1749– 1755.
http://www.ncbi.nlm.nih.gov/pubmed/19857164
38. Kahlmeter G., Giske C.G., Kirn T.J., Sharp S.E. Point-Counterpoint: differences between the European Committee on Antimicrobial Susceptibility Testing and Clinical and Laboratory Standards Institute Recommendations for reporting antimicrobial susceptibility results. J Clin Microbiol. 2019;57(9): e01129-19.
https://jcm.asm.org/content/57/9/e01129-19
39. Pantozzi F.L., Ibar M.P., Nievas V.F., Vigo G.B., Moredo F.A., Giacoboni G.I. Wild-type minimal inhibitory concentration distributions in bacteria of animal origin in Argentina. Rev Argent Microbiol. 2014;46(1):34–40.
https://www.sciencedirect.com/science/article/pii/S0325754114700458?via%3Dihub
40. Kronvall G., Giske C.G., Kahlmeter G. Setting interpretive breakpoints for antimicrobial susceptibility testing using disk diffusion. Int J Antimicrob Agents. 2011;38(4):281–290.
https://www.sciencedirect.com/science/article/abs/pii/S0924857911001968?via%3Dihub
41. Всемирная организация здравоохранения. Эпиднадзор за устойчивостью к противомикробным препаратам в Центральной Азии и Восточной Европе. Ежегодный доклад. Женева; 2019; 167 с.
http://www.euro.who.int/PubRequest?language=Russian
42. Сухорукова М.В. Определение чувствительности микроорганизмов к антибиотикам: что стоит за результатом. Клиническая микробиология и антимикробная химиотерапия. 2013;15(3):219–229.
https://cmac-journal.ru/publication/2013/3/cmac-2013-t15-n3-p219/
43. Кафтырева Л.А., Зуева Л.П., Колосовская Е.Н., Егорова С.А., Макарова М.А., Светличная Ю.С., Кузин А.А., Свистунов С.А. Принципы организации мониторирования устойчивости ведущих возбудителей инфекций, связанных с оказанием медицинской помощи, к антимикробным препаратам к антимикробным препаратам в лечебно-профилактических медицинских организациях здравоохранения. 2014; 137 с.
44. EUCAST. Экспертные правила определения чувствительности к антибиотикам EUCAST.
https://www.eucast.org/expert_rules_ and_intrinsic_resistance/
45. Полонская А.В., Корниенко М.А., Манолов А.И., Купцов Н.С., Смирнов Г.Б., Любасовская Л.А., Припутневич Т.В., Шитиков Е.А., Ильина Е.Н. Вариабельность генов рекомбиназ и mecA стафилококковой хромосомной кассеты Staphylococcus haemolyticus. Антибиотики и Химиотерапия. 2018;63(7–8):33–40.
https://www.antibiotics-chemotherapy.ru/jour/article/view/94/94
46. Тимофеева О.Г., Поликарпова С.В. Локальный микробиологический мониторинг штаммов Enterobacterales, продуцирующих карбапенемазы. Лабораторная служба. 2019;8(3):14.
https://www.mediasphera.ru/issues/laboratornaya-sluzhba/2019/3/1230521982019031014
47. Kumari R., Banerjee T., Anupurba S. Molecular detection of drug resistance to ofloxacin and kanamycin in Mycobacterium tuberculosis by using multiplex allelespecific PCR. J Infect Public Health. 2018;11(1):54–58.
https://www.sciencedirect.com/science/article/pii/S1876034117300990?via%3Dihub
48. Malik T., Naim A., Saeed A. Molecular Detection of TEM, SHV and CTX-M Genes among Gram-negative Klebsiella Isolates. Curr Drug Deliv. 2018;15(3):417– 423.
https://pubmed.ncbi.nlm.nih.gov/29295691/
49. Thirumalmuthu K., Devarajan B., Prajna L., Mohankumar V. Mechanisms of fluoroquinolone and aminoglycoside resistance in keratitis-associated Pseudomonas aeruginosa. Microb Drug Resist. 2019;25(6):813–823.
https://www.liebertpub.com/doi/10.1089/mdr.2018.0218
50. Shaaban M., Al-Qahtani A., Al-Ahdal M., Barwa R. Molecular characterization of resistance mechanisms in Pseudomonas aeruginosa isolates resistant to carbapenems. J Infect Dev Ctries. 2018;11(12):935–943.
https://jidc.org/index.php/journal/article/view/9501
51. EUCAST guidelines for detection of resistance mechanisms and specific resistances of clinical and/or epidemiological importance. 2017.
52. Global Antimicrobial Resistance Surveillance System. Molecular methods for antimicrobial resistance (AMR) diagnostics to enhance the Global Antimicrobial Resistance Surveillance System. 2019.
https://www.who.int/glass/resources/publications/molecular-methods-for-amr-diagnostics/en/
53. Global Antimicrobial Resistance Surveillance System. GLASS whole-genome sequencing for surveillance of antimicrobial resistance. 2020.
https://www.who.int/publications/i/item/9789240011007
54. Шайдуллина Э.Р., Эйдельштейн М.В., Склеенова Е.Ю., Сухорукова М.В., Козлов Р.С. Антибиотикорезистентность нозокомиальных карбапенемазопродуцирующих штаммов Enterobacterales в России: результаты эпидемиологического исследования 2014–2016 гг. Клиническая микробиология и антимикробная химиотерапия. 2018;20(4):362–369.
https://cmac-journal.ru/publication/2018/4/cmac-2018-t20-n4-p362/
55. Сухорукова М.В., Эйдельштейн М.В., Иванчик Н.В., Склеенова Е.Ю., Шайдуллина Э.Р., Шек Е.А., и соавт. Антибиотикорезистентность нозокомиальных штаммов Enterobacterales в стационарах России: результаты многоцентрового эпидемиологического исследования МАРАФОН 2015–2016. Клиническая микробиология и антимикробная химиотерапия. 2019;21(2):147–159.
https://cmac-journal.ru/publication/2019/2/cmac-2019-t21-n2-p147/
56. Попов Д.А. Сравнительная характеристика современных методов определения продукции карбапенемаз. Клиническая микробиология и антимикробная химиотерапия. 2019;21(2):125–33.
https://cmac-journal.ru/publication/2019/2/cmac-2019-t21-n2-p125/
57. Белобородов В.Б., Гусаров В.Г., Дехнич А.В., Замятин М.Н., Зубарева Н.А., Зырянов С.К. и соавт. Диагностика и антимикробная терапия инфекций, вызванных полирезистентными микроорганизмами. Вестник анестезиологии и реаниматологии. 2020;16(1):52–83.
http://www.antibiotic.ru/files/pdf/2019/guidelines-daticmm-20191204.pdf
58. Эйдельштейн М.В., Сухорукова М.В., Склеенова Е.Ю., Иванчик Н.В., Микотина А.В., Шек Е.А. и соавт. Антибиотикорезистентность нозокомиальных штаммов Pseudomonas aeruginosa в стационарах России: результаты многоцентрового эпидемиологического исследования «МАРАФОН» 2013–2014. Клиническая микробиология и антимикробная химиотерапия. 2017;19(1):37–41.
https://cmac-journal.ru/publication/2017/1/cmac-2017-t19-n1-p037/
59. Сухорукова М.В., Эйдельштейн М.В., Склеенова Е.Ю., Иванчик Н.В., Шек Е.А., Дехнич А.В. и соавт. Антибиотикорезистентность нозокомиальных штаммов Acinetobacter spp. в стационарах России: результаты многоцентрового эпидемиологического исследования «МАРАФОН» 2013–2014. Клиническая микробиология и антимикробная химиотерапия. 2017;19(1):42–48.
https://cmac-journal.ru/publication/2017/1/cmac-2017-t19-n1-p042/
60. Becker K., Denis O., Roisin S., Mellmann A., Idelevich E.A., Knaack D., et al. Detection of mecA – and mecC -Positive Methicillin-Resistant Staphylococcus aureus (MRSA) Isolates by the New Xpert MRSA Gen 3 PCR Assay. J Clin Microbiol. 2016;54(1):180–184.
https://jcm.asm.org/content/54/1/180
61. Tenover F.C., Tickler I.A., Le V.M., Dewell S., Mendes R.E., Goering R.V. Updating molecular diagnostics for detecting methicillin-susceptible and methicillinresistant Staphylococcus aureus isolates in blood culture bottles. J Clin Microbiol. 2019;57(11): e01195-19.
https://jcm.asm.org/content/57/11/e01195-19
62. AMRFinderPlus.
https://www.ncbi.nlm.nih.gov/pathogens/antimicrobial-resistance/AMRFinder/
63. ResFinder.
https://cge.cbs.dtu.dk/services/ResFinder/
64. Fontaine R.E. Describing epidemiologic data. The CDC Field Epidemiology Manual. Oxford University Press; 2018. 528 p.
https://www. cdc.gov/eis/field-epi-manual/chapters/Describing-Epi-Data.html
65. Arts DGT. Defining and improving data quality in medical registries: a literature review, case study, and generic framework. J Am Med Informatics Assoc. 2002;9(6):600–611.
https://academic.oup.com/jamia/articlelookup/doi/10.1197/jamia.M1087
66. НСИ. Федеральный справочник лабораторных исследований. Справочник лабораторных материалов и образцов. 2019.
https://nsi.rosminzdrav.ru/#!/refbook/1.2.643.5.1.13.13.11.1081/version/2.2
67. НСИ. Справочник территории (СМРПМЗ). 2016.
https://nsi.rosminzdrav.ru/#!/refbook/1.2.643.5.1.13.2.1.1.346/version/1.0
68. Stelling J. Руководство по WHONET. Глобальная система эпиднадзора за устойчивостью к противомикробным препаратам. 2016. 39 с.
http://www.whonet.org/Docs/WHONET_for_GLASS. Russian.docx
69. Всемирная организация здравоохранения. WHONET 5. Программное обеспечение базы данных микробиологической лаборатории. 128 с.
70. WHONET. Официальная документация.
https://whonet.org/documentation.html
71. WHONET for CAESAR. Manual. 2020; 71 p.
https://whonet.org/docserv/Manual_WHONET_for_CAESAR.pdf
72. WHO. Collaborating Centre for Surveillance of Antimicrobial resistance. BacLink.Excel, text files, and other desktop applications. 2006; 35 p.
https://whonet.org/Docs/BacLink 2.Excel, text files, other applications.doc
73. Pan American Health Organization. Epidemiological Surveillance of Healthcare-Associated Infections. 2011.
https://www.paho.org/hq/dmdocuments/2011/ENG_ Modulo_I_final.pdf
74. CDC. Procedure-associated Module. Surgical Site Infection (SSI) Event. 2020.
https://www.cdc.gov/nhsn/pdfs/pscmanual/9pscssicurrent.pdf
75. Infectious Diseases Society of America. Clinical practice guideline for the management of asymptomatic bacteriuria: 2019 update by the infectious diseases society of America. 2019.
https://www.idsociety.org/practiceguideline/asymptomatic-bacteriuria/